Tombusviridae
| Tombusviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

Das Kapsid des Tomato-bushy-stunt-Virus | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Tombusviridae | ||||||||||||||
| Links | ||||||||||||||
|
Die Tombusviridae sind eine Familie einzelsträngiger RNA-Pflanzenviren mit positiver Polarität. Es gibt derzeit (Stand August 2019) etwa 70 Arten oder mehr in dieser Familie, aufgeteilt in über zehn Gattungen[4][5] Die Mitglieder können entweder ein- oder zweikeimblättrige Pflanzen (Monokotyledonen oder Dikotyledonen) befallen, aber keine Art kann beide befallen.[6] Der Name leitet sich von der Typusart Tomato-bushy-stunt-Virus (TBSV) der zugehörigen Gattung Tombusvirus ab.[7]
Aufbau[Bearbeiten | Quelltext bearbeiten]
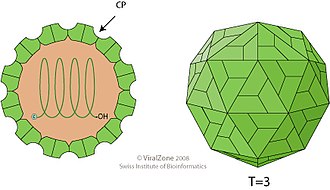
Die RNA ist in einem ikosaedrischen Kapsid mit T=3-Symmetrie eingeschlossen. Das Kapsid besteht aus 180 Einheiten eines Proteins (englisch single coat protein) besteht. Das Virion (Virusteilchen) hat einen Durchmesser von 28–35 nm und ist nicht umhüllt.[4][8]
Genom[Bearbeiten | Quelltext bearbeiten]


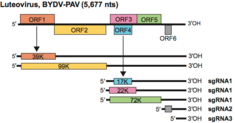
Das Genom der Tombusviridae ist linear und bei fast allen Vertretern unsegmentiert (monopartit), lediglich bei der Gattung Dianthovirus ist bekannt, dass das Genom aus zwei Segmenten besteht (bipartit segmentiert).[9] Das Genom ist ungefähr 4,6–4,8 kb lang, hat weder eine 5'-Kappe noch einen 3'-Polyadenin-Schwanz. Es kodiert 4–6 OffeneLeserahmen (ORFs). Die Polymerase kodiert ein Amber-Stopcodon, so dass ein Durchlesen ermöglicht wird. Dadurch kann ein zweites für die Replikation notwendiges Produkt erzeugt werden. Es gibt keine vom Virusgenom kodierte Helikase.[6]
Vermehrungszyklus[Bearbeiten | Quelltext bearbeiten]
Die Mitglieder der Tombusviridae replizieren im Zytoplasma, die Freisetzung nach außen erfolgt durch Lyse. Die Replikation folgt dem üblichen Replikationsmodell von Positivstrang-RNA-Viren.
Nachdem die Virionen von ihrem Kapsid befreit wurden (englisch uncoating), erfolgt die Freisetzung der viralen RNA ins Zytoplasma. Die Virionen sind gelegentlich aber auch in den Mitochondrien und Zellkernen vorhanden. Die virale RNA wird (an den Ribosomen) übersetzt um zunächst zwei Proteine zu erzeugen, die für die Synthese der Virus-RNA (Replikation und Transkription) erforderlich sind.[4] Die Translation erfolgt durch Leaky-Scanning.
Die Replikation findet dann im Zytoplasma in membranösen Vesikeln, den Virusfabriken (englisch viral factories), statt. Diese können mit dem endoplasmatischen Retikulum assoziiert sein, oder sich in modifizierten Organellen wie Peroxisomen, Mitochondrien und (seltener) Chloroplasten befinden.[6]
Zunächst wird aus dem (+)-Einzelstrang-RNA-Genom des Virus ein Doppelstrang-RNA-Genom erzeugt (dsRNA-Genom). Dieses dsRNA-Genom wird dann transkribiert bzw. repliziert, d. h. virale Messenger-RNAs (mRNAs) und ssRNA(+)-Genome werden hergestellt. Die RNA-abhängigen RNA-Polymerase (RdRp) erkennt interne subgenomische Promotoren auf der RNA negativer Polarität (ssRNA(-)) um die Kapsidproteine und Movement-Proteine zu transkribieren.[4]
Die virale RNA dient offenbar nicht nur als Vorlage für die Replikation, sondern ist auch in der Lage, die RNA-Synthese zu manipulieren und zu regulieren. Man konnte zeigen, dass die Stärke der RNA-Synthese durch die Cis-Elemente ode Cis-artige Elemente auf der RNA wie beim Red clover necrotic mosaic virus (RCNMV) beeinflusst wird.[10][11] Zu diesen gehören Kernpromotorsequenzen, die den Ort bestimmen, wo die Synthese des komplementären RNA-Strangs beginnt. Man nimmt an, dass dieser Mechanismus von der RNA-abhängigen RNA-Polymerase (RdRp) erkannt wird, die im Genom kodiert ist.[12][13]
Der Replikationsprozess hinterlässt schließlich einen Überschuss an RNA-Strängen positiver Polarität. Es wurde festgestellt, dass Tombusviridae GAPDH, ein metabolisches Wirtsenzym, zur Verwendung im Replikationszentrum kooptieren (mit hinzu benutzen). GAPDH kann an den RNA(-)-Strang binden und ihn so im Replikasekomplex halten, wodurch daraus synthetisierte ssRNA(+)-Stränge, exportiert und in der Wirtszelle akkumuliert (angereichert) werden können. Die Herabregulierung von GAPDH verringerte die Anreicherung viraler RNA und beseitigte den Überschuss an ssRNA(+)-Kopien.[14]
Der Zusammenbau der Virionen (Virus-Assembly) erfolgt im Zytoplasma. Ein Movement-Protein ermöglicht unter Umständen einen Transfer der Virionen zwischen benachbarten Zellen der Wirtspflanze auch ohne Freisetzung der Virionen durch Lyse.[4]
Die natürlichen Wirte der Tombusviridae sind Pflanzen. Die Übertragungswege sind mechanisch, über Samen und durch Kontakt (Gartenwerkzeuge).[4] Die Viren in dieser Familie werden normalerweise über den Boden übertragen; einige benutzen allerdings Pilzarten der Ordnung Chytridiales als Vektoren. Die Viren können sich sowohl durch das Wasser ausbreiten, als auch durch Wurzelwachstum in infizierten Boden und Kontakt zwischen Pflanzen, teilweise auch über Pollen oder Samen, je nach Virusspezies. Gärtnerisch konnten die Viren erfolgreich auch durch Pfropfen und mechanische Inokulation (Beimpfung) übertragen werden. erwartungsgemäß ist weder das Virion alleine (ohne Genom),noch das genetische Material alleine infektiös.[8]
Systematik[Bearbeiten | Quelltext bearbeiten]
Innere Systematik[Bearbeiten | Quelltext bearbeiten]
Die Systematik der Familie Tombusviridae ist gemäß International Committee on Taxonomy of Viruses (ICTV)[15] mit Stand Juni 2021 inkl. einiger weiterer Vorschläge nach NCBI (Stand 5. Februar 2021) wie folgt:[2]
Bereich: Riboviria, Gruppe: ssRNA(+)
- Familie Tombusviridae
- Unterfamilie Calvusvirinae
- Gattung Umbravirus
- Spezies Carrot mottle mimic virus (CMoMV)
- Spezies Carrot mottle virus (CMoV, Typusspezies)
- Spezies Ethiopian tobacco bushy top virus
- Spezies Groundnut rosette virus (Erdnuss-Rosetten-Virus, GRV)
- Spezies Lettuce speckles mottle virus (LSMV)
- Spezies Opium poppy mosaic virus
- Spezies Patrinia mild mottle virus
- Spezies Pea enation mosaic virus 2 (PEMV-2)
- Spezies Tobacco bushy top virus (TBTV)
- Spezies Tobacco mottle virus (TMoV)
- Unterfamilie Procedovirinae
- Gattung Alphacarmovirus
- Spezies Adonis mosaic virus
- Spezies Angelonia flower break virus
- Spezies Calibrachoa mottle virus (CMoV)
- Spezies Carnation mottle virus (CarMV, Typusspezies)
- Spezies Honeysuckle ringspot virus
- Spezies Nootka lupine vein clearing virus (NLVCV)
- Spezies Pelargonium flower break virus (PFBV)
- Spezies Saguaro cactus virus
- Gattung Alphanecrovirus
- Spezies Oliven-Latenz-Virus (en. Olive latent virus 1, OLV-1)
- Spezies Olive mild mosaic virus
- Spezies Tomaten-Nekrose-Virus (en. Potato necrosis virus, PoNV)
- Spezies Tabak-Nekrose-Virus A (en. Tobacco necrosis virus A, TNV-A, Typusspezies)
- Spezies „Beta vulgaris Alphanecrovirus-1“ (Vorschlag)[16]
- Gattung Aureusvirus
- Spezies Cucumber leaf spot virus
- Spezies Elderberry aureusvirus 1
- Spezies Johnsongrass chlorotic stripe mosaic virus
- Spezies Maize white line mosaic virus (MWLMV)
- Spezies Pothos latent virus (PoLV, Typusspezies)
- Spezies Yam spherical virus
- Gattung Avenavirus
- Spezies Oat chlorotic stunt virus (OCSV, Typusspezies)
- Gattung Betacarmovirus
- Spezies Cardamine chlorotic fleck virus
- Spezies Hibiscus chlorotic ringspot virus
- Spezies Japanese iris necrotic ring virus
- Spezies Turnip crinkle virus
- Gattung Betanecrovirus
- Spezies Beet-Black-Scorch-Virus (en. Beet black scorch virus, BBSV)
- Spezies Leek-white-stripe-Virus (en. Leek white stripe virus, LWSV)
- Spezies Tabak-Nekrose-Virus D (en. Tobacco necrosis virus D, TNV-D, Typusspezies)
- Gattung Gallantivirus
- Spezies Galinsoga mosaic virus (GaMV, Typusspezies)
- Gattung Gammacarmovirus
- Spezies Cowpea mottle virus
- Spezies Melon necrotic spot virus (MNSV, Typusspezies)
- Spezies Pea stem necrosis virus
- Spezies Soybean yellow mottle mosaic virus (SYMMV)
- Gattung Macanavirus
- Spezies Furcraea necrotic streak virus (FNSV, Typusspezies)
- Gattung Machlomovirus
- Spezies Maize chlorotic mottle virus (MCMV, Typusspezies)
- Gattung Panicovirus
- Spezies Cocksfoot mild mosaic virus
- Spezies Panicum mosaic virus (Hirse-Mosaik-Virus, PMV, Typusspezies)
- Spezies Thin paspalum asymptomatic virus
- Gattung Pelarspovirus[17]
- Spezies Clematis chlorotic mottle virus
- Spezies Elderberry latent virus (ElLDV)
- Spezies Jasmine mosaic-associated virus
- Spezies Pelargonium chlorotic ring pattern virus (PCRPV)
- Spezies Pelargonium line pattern virus (PLPV, Typusspezies)[17]
- Spezies Pelargonium ringspot virus (PelRSV)
- Spezies Rosa rugosa leaf distortion virus (RrLDV)
- Gattung Tombusvirus
- Spezies Artichoke mottled crinkle virus
- Spezies Carnation Italian ringspot virus
- Spezies Cucumber Bulgarian latent virus
- Spezies Cucumber necrosis virus (Cucumber-necrosis-Virus, CNV)
- Spezies Cymbidium ringspot virus
- Spezies Eggplant mottled crinkle virus
- Spezies Grapevine Algerian latent virus
- Spezies Havel River virus
- Spezies Lato River virus
- Spezies Limonium flower distortion virus
- Spezies Moroccan pepper virus
- Spezies Neckar River virus
- Spezies Pelargonium leaf curl virus
- Spezies Pelargonium necrotic spot virus
- Spezies Petunia asteroid mosaic virus (PetAMV)
- Spezies Sitke waterborne virus
- Spezies Tomato bushy stunt virus (Tomato-bushy-stunt-Virus, Tomatenzwergbusch-Virus, TBSV, Typusspezies)
- Gattung Zeavirus
- Spezies Maize necrotic streak virus (MNeSV)
- ohne zugewiesene Gattung
- Spezies Ahlum waterborne virus
- Spezies Bean mild mosaic virus
- Spezies Chenopodium-Nekrose-Virus (en. Chenopodium necrosis virus, ChNV)
- Spezies Cucumber soil-borne virus
- Spezies Trailing lespedeza virus 1 (TLV1)
- Spezies Weddel waterborne virus
- Unterfamilie Regressovirinae
- Gattung Dianthovirus
- Spezies Carnation ringspot virus (CRSV, Typusspezies)
- Spezies Red clover necrotic mosaic virus (RCNMV)
- Spezies Sweet clover necrotic mosaic virus
- ohne zugewiesene Unterfamilie
- Gattung Luteovirus (ehemals in 2020 aufgelöster Familie Luteoviridae)
- Spezies Apple associated luteovirus
- Spezies Apple luteovirus 1
- Spezies Barley yellow dwarf virus kerII
- Spezies Barley yellow dwarf virus kerIII
- Spezies Barley yellow dwarf virus MAV (BYDV-MAV, deutsch: Gelbverzwergungsvirus MAV)
- Spezies Barley yellow dwarf virus PAS (BYDV-PAS, de: Gelbverzwergungsvirus PAS)
- Spezies Barley yellow dwarf virus PAV (BYDV-PAV, de: Gelbverzwergungsvirus PAV, ex-Typus)
- Spezies Bean leafroll virus
- Spezies Cherry associated luteovirus
- Spezies Nectarine stem pitting associated virus
- Spezies Red clover associated luteovirus
- Spezies Rose spring dwarf-associated virus
- Spezies Soybean dwarf virus
Äußere Systematik[Bearbeiten | Quelltext bearbeiten]
Koonin et al (2015) vermuten, dass die Flaviviridae Ursprung der von ihnen postulierten Verwandtschaftsgruppe Negative-strand RNA viruses sind; diese Gruppe entspricht dem heutigen Phylum Negarnaviricota. Vorschlagsgemäße Schwestergruppe der die Flaviviridae wäre danach die Familie der Tombusviridae. Alle zusammen bilden sie nach diesem Vorschlag die von den Autoren postulierte Supergruppe „Flavivirus-like superfamily“.[18][19]
Shi et al (2016) bezeichnen die weitere Verwandtschaft der Flaviviridae ähnlich als „Flavi-like viruses“.[20]
Diese Vorschläge wurden inzwischen abgelöst durch die Master species List #35 des ICTV vom März 2020.[21] Eine Gegenüberstellung der Kladogramme findet sich bei Tymovirales §ICTV Master Species List #35.
Bemerkungen[Bearbeiten | Quelltext bearbeiten]
DI-Moleküle (englisch defective interfering RNA) sind RNAs, die aus dem viralen Genom hergestellt werden, die aber aufgrund von Verkürzungen und anderen Fehlern nicht in der Lage sind, Zellen (wie ein Virus) alleine zu infizieren. Stattdessen müssen sie (wie ein Satellitenvirus) mit einem intakten Helfervirus koinfiziert werden. Untersuchungen haben gezeigt, dass die Infektion von Pflanzen mit Tombusviren DI RNAs enthält, die direkt aus dem Virus-RNA-Genom und keinem Wirtsgenom stammen. Virale DI RNAs mit ihrer geringen Größe und den cis-wirkenden Elementen sind sowohl in vivo als auch in vitro gute Vorlagen für die Untersuchung der RNA-Replikation.[22][23][24]
Bei der Synthese einiger Proteine wird subgenomische RNA (englisch Sub-genomic RNA, sgRNA) verwendet. Sie entsteht durch vorzeitige Beendigung der (-)-Strangsynthese. In infizierten Zellen wurden sgRNAs und sgRNA-Negativ-Sense-Templates gefunden.[12][8]
Einzelnachweise[Bearbeiten | Quelltext bearbeiten]
- ↑ a b c d ICTV: ICTV Taxonomy history: Carrot mottle mimic virus, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- ↑ a b ICTV Master Species List 2018b v2 MSL #34v, März 2019
- ↑ P. Hopper, S. C. Harrison, R. T. Sauer: Structure of tomato bushy stunt virus. V. Coat protein sequence determination and its structural implications. In: Journal of Molecular Biology. 177. Jahrgang, Nr. 4, 25. August 1984, S. 701–713, doi:10.1016/0022-2836(84)90045-7, PMID 6481803.
- ↑ a b c d e f Viral Zone. ExPASy, abgerufen am 28. August 2019.
- ↑ ICTV: Virus Taxonomy. Abgerufen am 28. August 2019.
- ↑ a b c ICTV: Family - Tombusviridae, in: Virus Taxonomy. Ninth Report of the International Committee on Taxonomy of Viruses 2012, pp 1111–1138, 23 November 2011, doi:10.1016/B978-0-12-384684-6.00096-3
- ↑ Habili, N. and Symons, R. H. (1989). Evolutionary relationship between luteoviruses and other RNA plant viruses based on sequence motifs in their putative RNA polymerases and nucleic acid helicases. Nucleic Acids Research 17:23, S. 9543–9555
- ↑ a b c ICTVdB—The Universal Virus Database, version 3 00.074. Tombusviridae
- ↑ Wiley InterScience Encyclopedia of Life Sciences: Tombusviridae
- ↑ Lommel SA, Weston-Fina M, Xiong Z, Lomonossoff GP: The nucleotide sequence and gene organization of red clover necrotic mosaic virus RNA-2. In: Nucleic Acids Res. 16. Jahrgang, Nr. 17, September 1988, S. 8587–602, doi:10.1093/nar/16.17.8587, PMID 3047682, PMC 338578 (freier Volltext).
- ↑ Mizumoto H, Tatsuta M, Kaido M, Mise K, Okuno T: Cap-independent translational enhancement by the 3' untranslated region of red clover necrotic mosaic virus RNA1. In: J. Virol. 77. Jahrgang, Nr. 22, November 2003, S. 12113–12121, doi:10.1128/JVI.77.22.12113-12121.2003, PMID 14581548, PMC 254280 (freier Volltext).
- ↑ a b Beth L. Nicholson, Pui Kei K. Lee, K. A. White: Internal RNA replication elements are prevalent in Tombusviridae, in: Front. Microbiol., 6. August 2012, doi:10.3389/fmicb.2012.00279
- ↑ K. Andrew White, Peter D. Nagy: Advances in the Molecular Biology of Tombusviruses: Gene Expression, Genome Replication, and Recombination, in: Progress in Nucleic Acid Research and Molecular Biology, Band 78, 2004, S. 187–226, doi:10.1016/S0079-6603(04)78005-8
- ↑ Wang, R. and Nagy, P. (2008) Tomato bushy stunt virus Co-Opts the RNA-Binding Function of a Host Metabolic Enzyme for Viral Genomic RNA Synthesis. Cell Host & Microbe 3:3, S. 178–187
- ↑ ICTV: ICTV Master Species List 2020.v1, New MSL including all taxa updates since the 2019 release, March 2021 (MSL #36)
- ↑ NCBI: Beta vulgaris Alphanecrovirus-1 (species)
- ↑ a b Castaño A, Ruiz L, Hernández C (2009) Insights into the translational regulation of biologically active open reading frames of Pelargonium line pattern virus. Virology 386(2), S. 417–426
- ↑ Da diese Supergruppe (von den Autoren als englisch superfamily bezeichnet) mit den Nagarnaviricota ein Phylum enthält, muss ihr Rang notwendigerweise höher sein und ist nicht als Überfamilie zu verstehen. Ränge höher als Ordnung (wie z. B. Phylum) waren zum Zeitpunkt der 2015-er Arbeit vom ICTV aber noch gar nicht vorgegeben.
- ↑ Eugene V. Koonin, Valerian V. Dolja, Mart Krupovic: Origins and evolution of viruses of eukaryotes: The ultimate modularity, in: Virology Mai 2015; 479–480. 2–25. PMC 5898234 (freier Volltext), PMID 25771806.
- ↑ Mang Shi, Xian-Dan Lin, Nikos Vasilakis, Jun-Hua Tian, Ci-Xiu Li, Liang-Jun Chen, Gillian Eastwood, Xiu-Nian Diao, Ming-Hui Chen, Xiao Chen, Xin-Cheng Qin, Steven G Widen, Thomas G Wood, Robert B Tesh, Jianguo Xu, Edward C Holmes, Yong-Zhen Zhang: Divergent Viruses Discovered in Arthropods and Vertebrates Revise the Evolutionary History of the Flaviviridae and Related Viruses. In: Journal of Virology. 90. Jahrgang, Nr. 2, 2016, S. 659–69, doi:10.1128/JVI.02036-15, PMID 26491167, PMC 4702705 (freier Volltext).
- ↑ ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- ↑ Yoshimi Yamamura, Herman B. Scholthof: Tomato bushy stunt virus: a resilient model system to study virus-plant interactions. In: Molecular Plant Pathology. 6. Jahrgang, Nr. 5, 1. September 2005, S. 491–502, doi:10.1111/j.1364-3703.2005.00301.x, PMID 20565674.
- ↑ Karen-Beth G. Scholthof, Herman B. Scholthof, Andrew O. Jackson: The Effect of Defective Interfering RNAs on the Accumulation of Tomato Bushy Stunt Virus Proteins and Implications for Disease Attenuation. In: Virology. 211. Jahrgang, Nr. 1, 1. August 1995, S. 324–328, doi:10.1006/viro.1995.1410, PMID 7645230.
- ↑ NCBI: Defective interfering RNA-4 of tomato bushy stunt virus (TBSV-P DI-4) und Defective interfering RNA-5 of tomato bushy stunt virus (TBSV-P DI-5)