Enterobakteriophage MS2
| Enterobakteriophage MS2 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
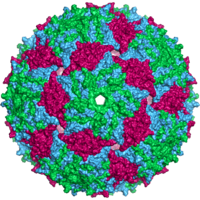
Kapsid des Bakteriophagen MS2 | ||||||||||||||||||
| Systematik | ||||||||||||||||||
| ||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||
| ||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||
| Emesvirus zinderi | ||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||
| MS2 | ||||||||||||||||||
| Links | ||||||||||||||||||
|
Escherichia-Virus MS2, offiziell Emesvirus zinderi (veraltet Enterobacteria phage MS2, deutsch Enterobakteriophage MS2, auch Enterobacteria-Phage MS2, Bakteriophage MS2 oder Coliphage MS2), kurz MS2 genannt, ist ein ikosaedrisches einzelsträngiges RNA-Virus mit positiver Polarität, der das Bakterium Escherichia coli und andere Mitglieder der Enterobacteriaceae infiziert.[4]
Phage MS2 ist eine Spezies der Gattung Emesvirus (früher Levivirus) und gehört mit ihr zu einer Familie eng verwandter bakterieller Viren, die auch Enterobakteriophage Qβ (offiziell Qubevirus durum, früher Escherichia virus Qbeta) und Enterobakteriophage BZ13 (offiziell Emesvirus japonicum, früher Escherichia virus BZ13) umfasst.[5]
Forschungsgeschichte[Bearbeiten | Quelltext bearbeiten]
Im Jahr 1961 wurde MS2 von Alvin John Clark isoliert und als RNA-haltiger Phage erkannt.[6] 1976 wurde das MS2-Genom als erstes Virus-Genom überhaupt vollständig sequenziert.[7] Dies gelang Walter Fiers und seinem Team, die bereits 1972 mit Gen des MS2-Hüllproteins das erste Gen vollständig sequenziert hatten.[8] Die erste vollständige Bestimmung eines DNA-Genoms erfolgte erst später, und zwar 1977 mit dem des Enterobakteriophagen ΦX174.[9]
Der erste Versuch einer statistischen Analyse des MS2-Genoms bestand in der Suche nach Mustern in der Nukleotidsequenz. Es wurden mehrere nichtkodierende Sequenzen (Genabschnitte, die nicht in Proteine übersetzt werden) identifiziert, wobei zum Zeitpunkt dieser Untersuchung (1979) die Funktion der nichtkodierenden Muster noch unbekannt war.[10]
Genom[Bearbeiten | Quelltext bearbeiten]
Das MS2-Genom ist eines der kleinsten bekannten Genome, bestehend aus 3569 Nukleotiden einzelsträngiger RNA.[7]

Es kodiert nur vier Proteine: das Reifungsprotein (A-Protein), das Lyse-Protein, das Hüllprotein und das Replikase-Protein.[4] Das Gen lys, das das Lyse-Protein kodiert, überlappt sowohl das 3'-Ende des Upstream-Gens cp, als auch das 5'-Ende des Downstream-Gens rep und war eines der ersten bekannten Beispiele für überlappende Gene. Das positivsträngige RNA-Genom dient als Messenger-RNA und wird übersetzt, nachdem das Virus in der Wirtszelle von seiner Hülle befreit wurde.
Obwohl die vier Proteine von derselben Messenger-RNA (d. h. Virus-RNA) kodiert werden, werden sie nicht alle auf den gleichen Ebenen exprimiert (Details s. u.).
Die Expression dieser Proteine wird durch ein komplexes Zusammenspiel zwischen Translation und RNA-Sekundärstruktur reguliert.
Kapsidstruktur[Bearbeiten | Quelltext bearbeiten]

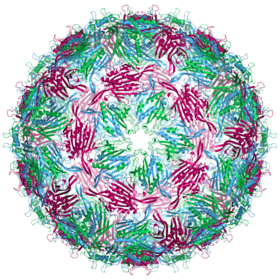
Das MS2-Virion (Viruspartikel) hat einen Durchmesser von etwa 27 nm, wie durch Elektronenmikroskopie bestimmt wurde.[11]
Es besteht aus einer Kopie des Reifungsproteins und 180 Kopien des Hüllproteins (organisiert in 90 Dimeren), die in einer ikosaedrischen Hülle mit der Triangulationszahl T=3 angeordnet sind, um das RNA-Genom im Inneren zu schützen.[12]
Die Struktur des Hüllproteins ist ein fünfsträngiges β-Faltblatt (β-sheet) mit zwei α-Helices und einer Haarnadel (hairpin). Nach Zusammenbau des Kapsids sind die Helices und die Haarnadel der Außenseite des Viruspartikels zugewandt, während das β-Sheet der Innenseite zugewandt ist.[13]
Wirkungsweise[Bearbeiten | Quelltext bearbeiten]
MS2 infiziert Enterobakterien, die den Fertilitätsfaktor F aufweisen. Dies ist ein Plasmid, mit dem die Bakterienzellen als DNA-Spender bei der bakteriellen Konjugation dienen können. Gene auf dem F-Plasmid führen zur Produktion eines F-Pilus (auch Sexpilus genannt), der als viraler Rezeptor (DNA-Empfänger) dient. MS2 bindet sich über sein einziges Reifungsprotein an die Seite des Pilus. Der genaue Mechanismus, durch den Phagen-RNA in das Bakterium eindringt, ist noch unbekannt.[4]
Sobald die virale RNA in die Zelle eingedrungen ist, beginnt sie als Messenger-RNA für die Produktion von Phagenproteinen zu fungieren. Das Gen cp für das am häufigsten vorkommende Protein, das Hüll- oder Mantelprotein, kann sofort übersetzt werden. Der Translationsstart (Start-Codon) des Replikasegens rep ist dagegen normalerweise in der RNA-Sekundärstruktur verborgen, kann aber vorübergehend geöffnet werden, wenn Ribosomen durch das Gen des Hüllproteins laufen. Die Replikase-Translation wird ebenfalls abgebrochen, sobald große Mengen an Hüllprotein hergestellt wurden. Dimere des Hüllproteins binden und stabilisieren die RNA-Operator-Hairpin, wodurch der Replikase-Start blockiert wird. Der Start des Reifungsproteingens mat ist in der RNA zugänglich, die repliziert wird, aber in der RNA-Sekundärstruktur der fertigen MS2-RNA versteckt ist. Dies gewährleistet die Translation von nur sehr wenigen Kopien des Reifungsproteins pro RNA. Schließlich kann die Übersetzung des Lyseproteingens nur durch Ribosomen gestartet werden, die die Translation des Hüllproteingens abgeschlossen haben.[4]
Auf diese Weise ist gewährleistet, dass die einzelnen Teilschritte der Virusvervielfältigung (RNA-Replikation, Bildung des Hüllproteins und schließlich Lyse (Auflösung) der Wirtszelle) in der richtigen Reihenfolge ablaufen.
Die Replikation des Plus-Strangs des MS2-Genoms erfordert die Synthese der komplementären Minus-Strang-RNA, die dann als Vorlage für die Synthese einer neuen Plus-Strang-RNA verwendet werden kann. Weil die MS2-Replikase schwer zu isolieren ist, wurde die MS2-Replikation viel weniger gut untersucht als die Replikation des nahe verwandten Bakteriophagen Qβ, dürfte aber sehr ähnlich sein.[4]
Es wird angenommen, dass die Bildung des Virions durch die Bindung von Reifungsprotein an die MS2-RNA ausgelöst wird. Nachweislich ist schon der Komplex aus Reifungsprotein und RNA infektiös. Der Aufbau der ikosaedrischen Hülle (des Kapsids) aus Hüllproteinen kann auch in Abwesenheit von RNA erfolgen, hierbei erfolgt die Kapsidanordnung durch Anbindung des Hüllprotein-Dimer an die RNA-Operator-Hairpin, so dass die Gesamtstruktur kernhaltig (mit der Virus-RNA) gebildet wird. Wenn MS2-RNA vorhanden ist, erfolgt die Montage auch schon bei viel niedrigeren Konzentrationen an Hüllprotein.[4]
Bakterienlyse und Freisetzung neu gebildeter Virionen geschieht, wenn sich genügend Lyseprotein angesammelt hat. Das Lyseprotein bildet Poren in der zytoplasmatischen Membran (Bakterienhülle), was zum Verlust des Membranpotentials und zum Abbau der Zellwand führt.[4]
Anwendungen[Bearbeiten | Quelltext bearbeiten]
Seit 1998 werden das MS2-Operator-Haarnadel- und Hüllprotein für den Nachweis von RNA in lebenden Zellen benutzt (MS2-Tagging).[14]
MS2 wurde aufgrund seiner strukturellen Ähnlichkeiten mit Noroviren, seiner ähnlich guten Proliferationsbedingungen und seiner Nicht-Pathogenität für Menschen als Ersatz für Noroviren in Studien zur Übertragung von Krankheiten verwendet.[15]
Siehe auch[Bearbeiten | Quelltext bearbeiten]
- Spiegelmans Monster §Entdeckung und Beschreibung
- Enterobakteriophage Qbeta
- Enterobakteriophage PhiX174
- Bakteriophage
Literatur[Bearbeiten | Quelltext bearbeiten]
- J. Glasgow, D. Tullman-Ercek: Production and applications of engineered viral capsids. In: Appl. Microbiol. Biotechnol., 98, 2014, S. 5847–5858, doi:10.1007/s00253-014-5787-3
Weblinks[Bearbeiten | Quelltext bearbeiten]
- Complete genome (also isolates R17, DL16, and J20)
Einzelnachweise[Bearbeiten | Quelltext bearbeiten]
- ↑ a b c d ICTV: ICTV Master Species List 2020.v1, New MSL including all taxa updates since the 2019 release, March 2021 (MSL #36)
- ↑ a b ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- ↑ ICTV Master Species List 2018b v1 MSL #34, Feb. 2019
- ↑ a b c d e f g J. van Duin, N. Tsareva: The Bacteriophages. Hrsg.: R. L. Calendar. Second Auflage. Oxford University Press, 2006, ISBN 0-19-514850-9, Single-stranded RNA phages. Chapter 15, S. 175–196.
- ↑ C. Z. Ni et al.: Crystal structure of the coat protein from the GA bacteriophage: model of the unassembled dimer. In: Protein Sci., 5, S. 2485–2493, 1996, PMC 2143325 (freier Volltext)
- ↑ J. E. Davis, J. H. Strauss, R. L. Sinsheimer: Bacteriophage MS2: another RNA phage. In: Science. 134. Jahrgang, Nr. 3488, 1961, doi:10.1126/science.134.3488.1425. S. 1427
- ↑ a b W. Fiers, R. Contreras, F. Duerinck, G. Haegeman, D. Iserentant, J. Merregaert, W. Min Jou, F. Molemans, A. Raeymaekers, A. Van den Berghe, G. Volckaert, M. Ysebaert: Complete nucleotide sequence of bacteriophage MS2 RNA: primary and secondary structure of the replicase gene. In: Nature. 260. Jahrgang, Nr. 5551, 1976, S. 500–507, doi:10.1038/260500a0, PMID 1264203.
- ↑ W. Min Jou, G. Haegeman, M. Ysebaert, W. Fiers: Nucleotide sequence of the gene coding for the bacteriophage MS2 coat protein. In: Nature. 237. Jahrgang, Nr. 5350, 1972, S. 82–88, doi:10.1038/237082a0, PMID 4555447.
- ↑ F. F. Sanger et al: Nucleotide sequence of bacteriophage φX174 DNA. In: Nature. 265. Jahrgang, Nr. 5596, 1977, S. 687–695, doi:10.1038/265687a0, PMID 870828.
- ↑ A search for patterns in the nucleotide sequence of the MS2 genome, Erickson, JW and Altman, GG; J. Mathematical Biology, April 1979, Volume 7, pp219–230
- ↑ J. H. Strauss, R. L. Sinsheimer: Purification and properties of bacteriophage MS2 and of its ribonucleic acid. In: J Mol Biol. 7. Jahrgang, Nr. 1, 1963, S. 43–54, doi:10.1016/S0022-2836(63)80017-0.
- ↑ K. Valegård, L. Lilias, K. Fridborg, T. Unge: The three-dimensional structure of the bacterial virus MS2. In: Nature. 345. Jahrgang, Nr. 6270, 1990, S. 36–41, doi:10.1038/345036a0.
- ↑ R. Golmohammadi, K. Valegård, K. Fridborg, L. Liljas: The refined structure of bacteriophage MS2 at 2·8 Å resolution. In: J Mol Biol. 234. Jahrgang, Nr. 3, 1993, S. 620–639, doi:10.1006/jmbi.1993.1616.
- ↑ E. Bertrand, P. Chartrand, M. Schaefer, S.M. Shenoy, R.H. Singer, R.M. Long: Localization of ASH1 mRNA particles in living yeast. In: Mol. Cell, 2, 1998, S. 437–445.
- ↑ Maggie Fox: Viruses spread ‘like crazy’ in an office, study finds.