Arenaviridae
| Arenaviridae | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
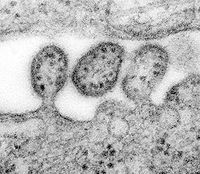
Knospung des Lassa-Virus | ||||||||||||||||
| Systematik | ||||||||||||||||
| ||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||
| ||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||
| Arenaviridae | ||||||||||||||||
| Links | ||||||||||||||||
|
Die Familie Arenaviridae oder Arenaviren umfasste ursprünglich nur eine Gattung Arenavirus von behüllten Viren mit einer einzelsträngigen, segmentierten ambisense-RNA als Genom. Zu den Arenaviridae gehören Erreger von Hämorrhagischem Fieber, Enzephalitis und Meningoenzephalitis beim Menschen, die durch Tiere (überwiegend Nagetiere) als natürliches Reservoir auf den Menschen übertragen werden. Die humanen Erkrankungen der Arenaviridae gehören damit zu den Zoonosen. Die neue Gattung Reptarenavirus umfasst neu isolierte Viren bei Reptilien (Boas und Pythons), die möglicherweise mit einer oft tödlich verlaufenden Infektion assoziiert sind.
Der Name der Familie leitet sich vom lateinisch arenosus ‚sandig‘ bzw. arena ‚Sand‘ her, um damit die sandige ribosomale Struktur im Inneren der Virionen zu beschreiben. Die ursprüngliche Bezeichnung Arenovirus wurden wegen Verwechslungsgefahr zu Adenovirus wieder fallengelassen.
Als erster Vertreter der Familie Arenaviridae wurde 1933 das Lymphozytäre-Choriomeningitis-Virus (LCMV) während einer Enzephalitis-Epidemie in St. Louis (USA) isoliert und beschrieben.
Morphologie
[Bearbeiten | Quelltext bearbeiten]Die Virionen (Virusteilchen) der Arenaviridae haben eine runde bis unregelmäßige Gestalt und sind je nach Spezies und Präparation des Untersuchungsmaterials 50 bis 300 nm (meistens zwischen 110 und 130 nm) im Durchmesser groß. In die Virushülle sind 8–10 nm lange, keulenförmige Glykoprotein-Spikes eingelagert. Die einzelnen Spikes bestehen aus einem Tetramer des viralen Hüllproteins. Die zwei Kapside zur Verpackung der zwei RNA-Segmente (Ribonukleokapside) sind ringförmig geschlossen und besitzen eine helikale Symmetrie; ihre Länge variiert zwischen 450 und 1300 nm. Jeweils ein Molekül der viralen RNA-Polymerase (L-Protein) ist an sie angelagert.
Morphologisch sehr außergewöhnlich ist die Anwesenheit einer wechselnden Anzahl von zellulären Ribosomen innerhalb des Virions, die den Viruspartikeln ihr „sandiges“ Aussehen verleihen. Ebenso findet man in gereinigten Viruspräparationen eine Anzahl unterschiedlicher zellulärer RNAs (darunter auch ribosomale RNA) sowie replikative Formen der viralen RNA, so verschiedene virale mRNAs (an die Ribosomen gebunden) und vollständige komplementäre Stränge des Virusgenoms. Diese nicht-genomischen RNAs befinden sich in wechselnder Menge alle außerhalb der Kapside.

Das virale Genom besteht aus zwei Molekülen einer einzelsträngigen RNA mit gemischter (d. h. ambisense, +/-) Polarität. Sie werden als L (large) und S (small) bezeichnet und sind etwa 7,5 bzw. 3,5 kB groß. Obwohl die Kapside ringförmig geschlossen sind, sind die RNA-Stränge linear und damit nicht zirkulär. Eine 19 bis 30 Basen lange Sequenz am 3'-Ende der RNA ist an beiden Strängen vorhanden und auch innerhalb der Virusfamilie konserviert.
Biologische Eigenschaften
[Bearbeiten | Quelltext bearbeiten]Die humanpathogenen Arenaviridae (mit Ausnahme der Spezies Tacaribe-Virus) haben verschiedene Nagetiere (Rodentia) als natürliches Reservoir. Das LCMV findet sich in Mäusen, die afrikanischen Arenaviren hauptsächlich in Vielzitzenmäuse (Mastomys) und Afrikanischen Weichratten (Praomys). Die Neuwelt-Arenaviren haben verschiedene Spezies der Neuweltmäuse (Unterfamilie Sigmodontinae) zum Wirt, darunter die Reisratten (Oryzomys), Stachelreisratten (Neacomys), Anden-Sumpfratten (Neotomys), Vespermäuse (Calomys) und Baumwollratten (Sigmodon). Von den anderen Arenaviren verschieden hat das Tacaribe-Virus Fruchtfledermäuse (Artibeus spp.) zum Wirt. Die Arenaviridae verursachen in ihren Wirten eine chronische Infektion mit Virämie und Virurie jedoch meist ohne Krankheitssymptome; dies geht auf eine langsame oder nicht vorhandene Abwehr durch das im Laufe der Zeit angepasste Immunsystem der Wirte zurück. Beim Menschen als nicht-angepassten Fehlwirt kommt es hingegen zu schweren, oft tödlichen Erkrankungen.
In der Gattung Reptarenavirus sind Arenaviren bei Schlangen zusammengefasst.
Systematik und Vorkommen
[Bearbeiten | Quelltext bearbeiten]Alle Spezies der Familie Arenaviridae wurden früher in die Gattung Arenavirus gestellt. Diese wurde aber 2014 aufgeteilt in die Gattungen Mammarenavirus mit Wirten unter den Mammalia und Reptarenavirus mit Wirten unter den Schlangen.[3][4][5] Weitere Spezies kamen später hinzu. Mit Stand März 2020 ist die Systematik nach ICTV wie folgt[6] (ergänzt um einige Kandidaten nach NCBI):
- Familie Arenaviridae
- Gattung Mammarenavirus[7]
- LCMV/Lassa-Komplex (Altwelt-Arenaviren)
- Spezies Ippy-Virus (IPPYV), Zentralafrikanische Republik (Wirt: Praomys)
- Spezies Lassa-Virus (LASV), Westafrika
- Spezies Lujo-Virus (LUJV), Sambia, Südafrika
- Spezies Lunk-Virus (NKS-1)
- Spezies Lymphozytäre-Choriomeningitis-Virus (en. Lymphocytic choriomeningitis virus, LCMV), Europa, Amerika
- Spezies Mobala-Virus (MOBV), Zentralafrikanische Republik (Wirt: Praomys)
- Spezies Mopeia-Virus (Mopeia virus, MOPV), inklusive Morogoro-Virus, Mosambik, Simbabwe
- Weitere Vorschläge zu Altwelt-Arenaviren, deren Bestätigung durch ICTV noch fehlt sind u. a.:[5][8]
- Spezies Dandenong-Virus (Dandenong virus, „DANV“)
- Spezies Gbagroube-Virus (Gbagroube virus)
- Spezies Menekre-Virus (Menekre virus)
- Tacaribe-Komplex: (Neuwelt-Arenaviren)
- Spezies Allpaahuayo-Virus (ALLV)
- Spezies Amapari-Virus (AMAV), Brasilien
- Spezies Bear-Canyon-Virus (BCNV)
- Spezies Chapare-Virus, Bolivien
- Spezies Cupixi-Virus (CPXV)
- Spezies Flexal-Virus (FLEV), Brasilien
- Spezies Guanarito-Virus (GTOV), Venezuela
- Spezies Junin-Virus (JUNV, wissenschaftlich Argentinian mammarenavirus), Argentinien
- Spezies Latino-Virus (LATV), Bolivien
- Spezies Machupo-Virus (MACV), Bolivien
- Spezies Oliveros-Virus (OLVV), Argentinien
- Spezies Parana-Virus (PARV), Paraguay
- Spezies Pichinde-Virus (PICV), Kolumbien
- Spezies Pirital-Virus (PIRV), Venezuela
- Spezies Sabiá-Virus (Sabia virus, SABV), Brasilien
- Spezies Tacaribe-Virus (TCRV), Westindische Inseln
- Spezies Tamiami-Virus (TAMV), Florida
- Spezies Whitewater-Arroyo-Virus (WWAV), New Mexico, USA
- Spezies Rio-Cacarana-Virus (RCAV), Argentinien
- Spezies Pampa-Virus (PAMV), Argentinien
- Spezies Whitewater-Arroyo-Virus
- Weitere Vorschläge zu Neuwelt-Arenaviren, deren Bestätigung durch ICTV noch fehlt sind u. a.:[5]
- Spezies Black-Mesa-Virus
- Spezies Orogrande-Virus
- Spezies Pinhal-Virus
- ohne bekannte Zuordnung zu einem der obigen Komplexe sind:
- Spezies Allpahuayo mammarenavirus
- Spezies Alxa mammarenavirus
- Spezies Bear Canyon mammarenavirus
- Spezies Brazilian mammarenavirus
- Spezies Cali mammarenavirus
- Spezies Chevrier mammarenavirus
- Spezies Gairo mammarenavirus
- Spezies Loei River mammarenavirus
- Spezies Luna mammarenavirus
- Spezies Mariental mammarenavirus
- Spezies Merino Walk mammarenavirus
- Spezies Okahandja mammarenavirus
- Spezies Paraguayan mammarenavirus
- Spezies Planalto mammarenavirus
- Spezies Ryukyu mammarenavirus
- Spezies Serra do Navio mammarenavirus
- Spezies Solwezi mammarenavirus
- Spezies Souris mammarenavirus
- Spezies Wenzhou mammarenavirus
- Spezies Xapuri mammarenavirus
- Gattung Reptarenavirus
- Spezies Ordinary Reptarenavirus
- Tavallinen-Suomalainen-Mies-Virus 2 (Tavallinen suomalainen mies virus 2, -2)[9]
- Spezies Schlangen-Reptarenavirus 1 (Alethinophid 1 reptarenavirus, offiziell Golden reptarenavirus, Typusspezies, bei Alethinophidia)[10]
- Golden-Gate-Virus (GGV)
- Spezies Schlangen-Reptarenavirus 2 (Alethinophid 2 reptarenavirus, offiziell California reptarenavirus)[11]
- ROUT-Virus (Boa-Arenavirus NL B3)
- Spezies Schlangen-Reptarenavirus 3 (Alethinophid 3 reptarenavirus, offiziell Rotterdam reptarenavirus)[12]
- CAS-Virus (California-Academy-of-Science-Virus, CASV)
- Spezies Universität-Gießen-Virus (offiziell Giessen reptarenavirus, UGV)[13]
- Nicht klassifizierte Reptarenaviren nach NCBI (Auswahl Stand November 2020):[14]
- Spezies Alethinophid 1 reptarenavirus[15]
- Spezies Alethinophid 2 reptarenavirus[16]
- „Universität-Helsinki-Virus“ („UHV“)[17]
- Spezies Aurora-Borealis-Virus (en. Aurora borealis virus, „ABV“)[18]
- Spezies Bis spoeter virus[19]
- Spezies Frankfurter Strasse virus-1[20]
- Spezies Gaucho virus-1[21]
- Spezies Gruetzi mitenand virus[22]
- Spezies Hans-Kompis-Virus (en. Hans Kompis virus, „HKV“)[23]
- Spezies Suri-Vanera-Virus (Suri Vanera virus, „SVaV“)[24]
- Spezies Tavallinen-Suomalainen-Mies-Virus 1 (Tavallinen suomalainen mies virus 1, „TSMV-1“)[25]
- Nicht klassifizierte Reptarenaviren nach NCBI (Auswahl Stand November 2020):[14]
- Gattung Hartmanivirus (nicht zu verwechseln mit der Gattung Haartmanvirus der Bakteriophagen-Familie Demerecviridae, Ordnung Caudovirales)[26]
- Spezies Haartman-Institut-Schlangen-Virus (offiziell Haartman hartmanivirus, HISV)[27]
- Haartman Institute snake virus
- Spezies Muikkunen hartmanivirus[28]
- Dante Muikkunen virus 1
- Spezies Schoolhouse hartmanivirus[29]
- Old schoolhouse virus 1
- Spezies Zurich hartmanivirus[30]
- Veterinary Pathology Zurich virus 1
- Nicht klassifizierte Hartmaniviren nach NCBI (Auswahl Stand November 2020):[31]
- Spezies Andere Heimat virus-1
- Spezies Haartman Institute snake virus 2
- Spezies Old schoolhouse virus 2
- Spezies SetPatVet virus-1
- Spezies Veterinary Pathology Zurich virus 2
- Nicht klassifizierte Hartmaniviren nach NCBI (Auswahl Stand November 2020):[31]
- Gattung Antennavirus
- Spezies Hairy antennavirus[32]
- Spezies Striated antennavirus[33]
- Wenling frogfish arenavirus 1[34]
Literatur
[Bearbeiten | Quelltext bearbeiten]- P.J. Southern: Arenaviridae: the viruses and their replication. in: David M. Knipe, Peter M. Howley et al. (eds.): Fields' Virology, 3. Auflage, Philadelphia 1996
- C.M. Fauquet, M.A. Mayo et al.: Eighth Report of the International Committee on Taxonomy of Viruses, London, San Diego, 2004
Weblinks
[Bearbeiten | Quelltext bearbeiten]Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ a b ICTV: ICTV Taxonomy history: Akabane orthobunyavirus, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- ↑ ICTV Master Species List 2018b v1 MSL #34, Feb. 2019
- ↑ ICTV proposals 2014.011a-dV et al., Mark D. Stenglein et al.
- ↑ ICTV proposals 2014.012aV et al., Michael J. Buchmeier et al.
- ↑ a b c Sheli R. Radoshitzky et al.: Past, present, and future of arenavirus taxonomy (PDF), in: Arch Virol., Springer-Verlag Wien, 3. Mai 2015, doi:10.1007/s00705-015-2418-y
- ↑ ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- ↑ SIB: Mammarenavirus, auf: ViralZone
- ↑ NCBI: unclassified Old world arenaviruses
- ↑ NCBI: Tavallinen suomalainen mies virus 2 (no rank)
- ↑ ICTV: ICTV Taxonomy history: Golden reptarenavirus
- ↑ ICTV: ICTV Taxonomy history: California reptarenavirus
- ↑ ICTV: ICTV Taxonomy history: Rotterdam reptarenavirus
- ↑ ICTV: ICTV Taxonomy history: Giessen reptarenavirus
- ↑ NCBI: unclassified Reptarenavirus
- ↑ NCBI: Alethinophid 1 reptarenavirus (species)
- ↑ NCBI: Alethinophid 2 reptarenavirus (species)
- ↑ NCBI: University of Helsinki virus (no rank)
- ↑ NCBI: Aurora borealis virus (species)
- ↑ NCBI: Bis spoeter virus (species)
- ↑ NCBI: Frankfurter Strasse virus-1 (species)
- ↑ NCBI: Gaucho virus-1 (species)
- ↑ NCBI: Gruetzi mitenand virus (species)
- ↑ NCBI: Hans Kompis virus (species)
- ↑ NCBI: Suri Vanera virus (species)
- ↑ NCBI: Tavallinen suomalainen mies virus 1 (species)
- ↑ NCBI: Haartmanvirus (genus)
- ↑ ICTV: ICTV Taxonomy history: Haartman hartmanivirus
- ↑ ICTV: ICTV Taxonomy history: Muikkunen hartmanivirus
- ↑ ICTV: ICTV Taxonomy history: Schoolhouse hartmanivirus
- ↑ ICTV: ICTV Taxonomy history: Zurich hartmanivirus
- ↑ NCBI: unclassified Hartmanivirus
- ↑ ICTV: ICTV Taxonomy history: Hairy antennavirus
- ↑ ICTV: ICTV Taxonomy history: Striated antennavirus
- ↑ NCBI: Wenling frogfish arenavirus 1 (no rank)

