Norovirus
| Norovirus | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

3D-Modell von Norovirus-Virionen | ||||||||||||||||||
| Systematik | ||||||||||||||||||
| ||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||
| ||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||
| Norovirus | ||||||||||||||||||
| Links | ||||||||||||||||||
|
Die Gattung Norovirus umfasst unbehüllte Viren mit einer einzelsträngigen RNA mit positiver Polarität (ss(+)RNA) aus der Familie Caliciviridae. Bisher wurden verschiedene Norovirus-Spezies beim Menschen sowie bei Rindern, Schweinen, Mäusen und Austern entdeckt. Besonders die humanen Noroviren haben als Erreger einer viralen Gastroenteritis eine große medizinische Bedeutung.
Der Name Norovirus ist abgeleitet von Norwalk-Virus, der Bezeichnung für die Typspezies der Gattung.
Erstbeschreibung
[Bearbeiten | Quelltext bearbeiten]Die Typspezies der Gattung Norovirus, das Norwalk-Virus, wurde in Stuhlproben eines viralen Gastroenteritis-Ausbruchs von 1968 in Norwalk, Ohio, durch Immunelektronenmikroskopie 1972 erstmals morphologisch charakterisiert.[5] Um den Zusammenhang zwischen dem gefundenen Virus und einer Erkrankung an Gastroenteritis beweisen zu können, wurde gereinigtes Stuhl-Ultrafiltrat (gewonnen aus menschlichem Kot erkrankter Patienten) an freiwillige Personen oral verabreicht, die anschließend ebenfalls erkrankten.
Morphologie
[Bearbeiten | Quelltext bearbeiten]

Noroviren bestehen aus einem unbehüllten, ikosaedrischen (zwanzigflächigen) Kapsid, das von dem Hauptkapsidprotein (englisch major capsid protein, MCP) mit Bezeichnung VP1 gebildet wird. VP1-Proteine können sich zusammensetzen zu größeren Kapsiden, die aus 180 Proteinen bestehen, einen Durchmesser von 38 bis 40 nm haben und eine T=3-Symmetrie aufweisen;[6] sie können auch zu kleineren Kapsiden (23 nm) aus 60 VP1-Proteinen mit T=1-Symmetrie assemblieren.[7] Beide Strukturen werden im elektronenmikroskopischen Bild als runde Gebilde mit unscharfem Rand dargestellt (siehe Abbildung).[8]
Am Bau der Proteinkapsel eines Virions ist außerdem ein Nebenkapsidprotein (englisch minor capsid protein, mCP) beteiligt, das sich innenseitig befindet. Biochemische Studien zeigten, dass es an der inneren Seite des Kapsids bindet und die Stabilität des Kapsids erhöht.[9][10] Im Inneren des Kapsids liegt das Virusgenom als positive Einzelstrang-RNA (ss(+)RNA) vor. An das 5′-Ende der RNA des Genoms ist das VPg-Protein kovalent gebunden, ihr 3′-Ende ist polyadenyliert.[6]
Genom
[Bearbeiten | Quelltext bearbeiten]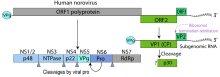
Das einzelsträngige RNA-Genom der Noroviren ist etwa 7,3 bis 7,7 kb groß und umfasst drei teilweise überlappende offene Leserahmen (ORFs).
Der ORF1 codiert für ein Polyprotein, welches nach der Translation von einer viralen Protease in sechs nicht-strukturelle Proteine geschnitten wird (NS 1/2 – NS 7; die Nummerierung entspricht der Anordnung der für die Proteine codierenden Gene auf dem Genom).[6] Hierzu zählen unter anderem die virale RNA-Polymerase (NS 7) und die virale Protease (NS 6). Durch ORF2 ist das Kapsidprotein VP1 codiert und durch ORF3 das virion-assoziierte Strukturprotein VP2 unbekannter Funktion. Das Genom der Noroviren kann bei Infektion einer Zelle mit verschiedenen Stämmen oder Varianten durch Rekombination sehr effektiv neue Varianten und Subtypen hervorbringen. Ein den Influenzaviren ähnlicher Antigendrift und ein Antigenshift werden auch bei einigen Spezies der Noroviren beobachtet. Eine hypervariable Region befindet sich in der P2-Domäne des VP1 Proteins, welche an der Außenseite des Viruskapsids liegt und als Bindestelle für Antikörper dient, somit besonders starker Selektion durch das Immunsystem ausgesetzt ist.[11]
In einer Studie von 2017 hat die Gruppe um Parra und Green umfangreiche Datensätze zu menschlichen Norovirus-Infektionen durchforstet und die ss(+)RNA von über 20 Genotypen beider Genotypgruppen I und II hinsichtlich der im Laufe von Jahren angesammelten Mutationen verglichen. Während das Genom der übrigen untersuchten Genotypen relativ stabil blieb, änderte sich das Norovirus des Typs GII.4 rasch. Es evolvierte sowohl in einem Wirt wie auch im Wechsel von einem Wirt zum anderen; insbesondere durch Sequenzänderung im ORF2 entstanden zahlreiche Varianten dieses Genotyps mit unterschiedlichem VP1-Protein. Die demgegenüber eher statischen anderen Genotypen ließen sich hinsichtlich ihrer Antigene in mehrere Untergruppen zusammenfassen, die womöglich einem „Immunotyp“ entsprechen, was die Entwicklung von Impfstoffen vereinfachen würde.[12]
Replikationszyklus
[Bearbeiten | Quelltext bearbeiten]Der Replikationszyklus ist erst teilweise verstanden. Die meisten Experimente wurden mit dem Murinen Norovirus-Subtyp durchgeführt. Es wird angenommen, dass Histo-Blutgruppenantigene, zu welchen das AB0-System der Blutgruppen, die Vorläufersubstanz H und die Lewis-Antigene gehören, entscheidend für die Bindung des Virus an seine Wirtszelle sind.[13][14] Die Antigene sind auf roten Blutkörperchen wie auch auf Epithel- und Endothelzellen vorhanden.[15] Ein Zelltropismus wurde für das Murine Norovirus beobachtet, das Virus infiziert sowohl Lymphocyten wie auch Enterozyten.[16] Humane Norovirus-Subtypen konnten in vitro sowohl in B-Lymphozyten als auch in aus Stammzellen gezüchteten Darm-Organoiden repliziert werden, jedoch nur in geringen Mengen.[17][18]
Nach der Aufnahme des Virus durch die Wirtszelle wird die virale mRNA durch die wirtseigene RNA-Polymerase abgelesen und translatiert. Dabei dient VPg (NS 5), welches kovalent am 5’-Ende des mRNA-Strangs gebunden ist, als Proteinprimer.[19] Darüber hinaus spielt VPg bei der Initiation der Translation eine Rolle.[19] Translation der einzelnen Proteine erfolgt durch Leaky-Scanning und Translations-Reinitiation.[19] Die virale Protease (NS 6) teilt das neu entstandene Polyprotein in einzelne nicht-strukturelle Proteine, die zur Replikation des Virus benötigt werden.[20] Virale mRNA wird im Cytoplasma durch die RNA-abhängige RNA-Polymerase (NS 7) dupliziert. Da diese, ähnlich wie bei anderen RNA-Viren, keine Korrekturfunktion besitzt, hat die neuerzeugte RNA eine deutlich erhöhte Mutationsrate. (Einbau einer Mutation pro 10000 bp). Das führt zur Bildung von Quasispezies, welche eine schnelle Adaptation und Immunevasion erlauben.[21] Das Kapsidprotein VP1 bildet das Kapsid, in welches die virale mRNA verpackt wird, bevor das Virus die Zelle verlässt.[20]
Für die nicht-strukturellen Proteine NS1/2, NS 3 und NS4, auch bekannt unter den Namen p48, NTPase und p22, wurden immunregulierende Funktionen beobachtet.[19]
Systematik
[Bearbeiten | Quelltext bearbeiten]- Gattung Norovirus (veraltet Norwalk-like virus)
- Spezies Norwalk-Virus (Humanes Norovirus, en. Norwalk virus)
- vorläufig (per Vorschlag) klassifizierte Kandidaten:
- Spezies ‚Humanes Norovirus-Alphatron‘
- Spezies ‚Humanes Norovirus Saitama‘
- Spezies ‚Bovines Norovirus-CH126‘
- Spezies ‚Bovines Norovirus-Jena‘
- Spezies ‚Murines Norovirus 1‘
- Spezies ‚Porcines Norovirus‘ (Norovirus des Schweines)
- Spezies ‚Norovirus der Auster‘
Humane Noroviren
[Bearbeiten | Quelltext bearbeiten]Für Informationen über die klinischen Verläufe, Übertragungen, Epidemiologie etc. siehe Humane Noroviren (Norwalk-Virus).
Literatur
[Bearbeiten | Quelltext bearbeiten]- M. K. Koopmans u. a.: Genus Norovirus. In: C. M. Fauquet, M. A. Mayo u. a.: Virus taxonomy: classification and nomenclature of viruses: eighth report of the International Committee on the Taxonomy of Viruses. Academic Press, London/ San Diego 2005, ISBN 0-12-249951-4, S. 847f.
Weblinks
[Bearbeiten | Quelltext bearbeiten]- Gattung Norovirus in der Datenbank des ICTV
- Spezies, Subtypen und Isolate der Gattung Norovirus (NCBI)
- Josephine Franke: Noroviren verändern bakterielles Mikrobiom: Auch Darmbakterien reagieren auf eine Infektion mit enterischen Viren. Auf: scinexx.de vom 27. Januar 2022
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ Juliana D. Siqueira, Maria Gloria Dominguez-Bello, Monica Contreras, Orlana Lander, Eric Delwart: Complex virome in feces from Amerindian children in isolated Amazonian villages. In: Nature Communications. 9(1), Dezember 2018, doi:10.1038/s41467-018-06502-9
- ↑ Aase B Mikalsen, Pål Nilsen, Marianne Frøystad-Saugen, Karine Lindmo, Trygve M Eliassen, Marit Rode, Øystein Evensen: Characterization of a Novel Calicivirus Causing Systemic Infection in Atlantic Salmon (Salmo salar L.): Proposal for a New Genus of Caliciviridae. In: PLoS ONE. 9(9):e107132, September 2014, doi:10.1371/journal.pone.0107132
- ↑ a b c d e ICTV: ICTV Taxonomy history: Rabbit hemorrhagic disease virus, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- ↑ ICTV Master Species List 2018b v1 MSL #34, Feb. 2019.
- ↑ R. Dolin, N. R. Blacklow u. a.: Biological properties of Norwalk agent of acute infectious nonbacterial gastroenteritis. In: Proceedings of the Society of Experimental Biological Medicine. Band 140, Nummer 2, 1972, S. 578–583. PMID 4624851.
- ↑ a b c Caliciviridae - ICTV Ninth Report. Archiviert vom (nicht mehr online verfügbar) am 1. Februar 2021; abgerufen am 3. Januar 2021. Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis.
- ↑ L. J. White, M. E. Hardy, M. K. Estes: Biochemical characterization of a smaller form of recombinant Norwalk virus capsids assembled in insect cells. In: Journal of Virology. Oktober 1997, PMC 192173 (freier Volltext).
- ↑ D. C. Lewis: Three serotypes of norwalk‐like virus demonstrated by solid‐phase immune electron microscopy. Journal of Medical Virology, Januar 1990, doi:10.1002/jmv.1890300117.
- ↑ Sompong Vongpunsawad, B. V. Venkataram Prasad, Mary K. Estes: Norwalk Virus Minor Capsid Protein VP2 Associates within the VP1 Shell Domain. In: Journal of Virology. April 2013, doi:10.1128/JVI.03508-12.
- ↑ Y. Lin, L. Fengling, W. Lianzhu, Z. Yuxiu, J. Yanhua: Function of VP2 protein in the stability of the secondary structure of virus-like particles of genogroup II norovirus at different pH levels: function of VP2 protein in the stability of NoV VLPs. In: Journal of Microbiology. 3. Oktober 2014, doi:10.1007/s12275-014-4323-6.
- ↑ D. J. Allen, J. J. Gray, C. I. Gallimore, J. Xerry, M. Iturriza-Gómara: Analysis of Amino Acid Variation in the P2 Domain of the GII-4 Norovirus VP1 Protein Reveals Putative Variant-Specific Epitopes. In: PLOS ONE. 23. Januar 2008, doi:10.1371/journal.pone.0001485.
- ↑ Gabriel I. Parra, R. Burke Squires, Consolee K. Karangwa, Jordan A. Johnson, Cara J. Lepore, Stanislav V. Sosnovtsev, Kim Y. Green: Static and evolving Norovirus genotypes: Implications for epidemiology and immunity. In: PLoS Pathogens. 2017/13(1) e1006136. doi:10.1371/journal.ppat.1006136 (freier Volltext).
- ↑ V. R. Graziano, J. Wei, C. B. Wilen: Norovirus Attachment and Entry. In: Viruses. 30. Mai 2019, doi:10.3390/v11060495.
- ↑ Julie E Heggelund, Annabelle Varrot, Anne Imberty, Ute Krengel: Histo-blood group antigens as mediators of infections. In: Current Opinion in Structural Biology. Juni 2017, doi:10.1016/j.sbi.2017.04.001.
- ↑ S. Marionneau, A. Cailleau-Thomas, J. Rocher, B. Le Moullac-Vaidye, N. Ruvoën, M. Clément, J. Le Pendu: ABH and Lewis histo-blood group antigens, a model for the meaning of oligosaccharide diversity in the face of a changing world. In: Biochimie. Juli 2001, doi:10.1016/S0300-9084(01)01321-9.
- ↑ Christiane E. Wobus: The Dual Tropism of Noroviruses. Journal of Virology, 31. Juli 2018, doi:10.1128/JVI.01010-17.
- ↑ V. Costantini, E. K. Morantz, H. Browne u. a.: Human Norovirus Replication in Human Intestinal Enteroids as Model to Evaluate Virus Inactivation. Emerging Infectious Diseases, August 2018, doi:10.3201/eid2408.180126.
- ↑ Melissa K Jones, Katrina R Grau, Veronica Costantini u. a.: Human norovirus culture in B cells. In: Nature Protocols. 1. Juni 2016, doi:10.1038/nprot.2015.121.
- ↑ a b c d Lucy G. Thorne, Ian G. Goodfellow: Norovirus gene expression and replication. In: Journal of General Virology. 1. Februar 2014, doi:10.1099/vir.0.059634-0.
- ↑ a b Michele E. Hardy: Norovirus protein structure and function. In: FEMS Microbiology Letters. 1. Dezember 2005, doi:10.1016/j.femsle.2005.08.031.
- ↑ Eric F. Donaldson, Lisa C. Lindesmith, Anna D. LoBue, Ralph S. Baric: Viral shape-shifting: norovirus evasion of the human immune system. In: Nature Reviews Microbiology. 2. Februar 2010, doi:10.1038/nrmicro2296.