Ramachandran-Plot

Ein Ramachandran-Plot (auch Ramachandran-Diagramm) ist ein Diagramm, das die statistische Verteilung von Kombinationen von jeweils zwei Diederwinkeln (φ und ψ) eines bestimmten Protein-Backbones darstellt. Es ist nach dem indischen Wissenschaftler G. N. Ramachandran benannt.
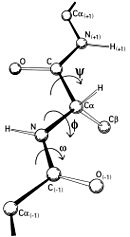
Mathematisch gesehen ist der Ramachandran-Plot also eine Darstellung einer Funktion . φ ist der Diederwinkel für die Atome C(i),Cα(i),N(i),C(i+1) und ψ für N(i-1),C(i),Cα(i),N(i). Diese zwei Diederwinkel beschreiben also die Konformation der Proteinhauptkette an der Aminosäure i.
Ramachandran-Plots sind für jeweilige bestimmte Typen von Proteinmolekülen charakteristisch. Man kann anhand eines Ramachandran-Plots auch die Häufigkeit von α-Helices, β-Faltblättern und anderer Protein-Sekundärstrukturen im Molekül abschätzen. Dabei ist aber zu beachten, dass auch die Aminosäuren der Schlaufenregionen, die also nicht die Sekundärstruktur einer α-Helix oder eines β-Faltblattstranges einnehmen, in einem der energiearmen („erlaubten“) Bereiche des Ramachandranplots liegen müssen. Die Beobachtung, dass die Hauptkettenkonformation einer Aminosäure in dem für α-Helices oder β-Faltblätter typischen Bereich liegt, bedeutet also nicht, dass diese Aminosäure auch Teil einer α-Helix oder eines β-Faltblattstranges ist.
Der Ramachandran-Plot ist ein wichtiges Modul in Computerprogrammen, die automatisch die Qualität von Strukturbestimmungen (Kristallstrukturanalysen, Kernspinresonanzspektroskopie, Molekulare Modellierung usw.) von Proteinen überprüfen, zum Beispiel in WHATIF,[1] PROCHECK[2] oder USF.[3] Alternativ kann ein Janin-Plot verwendet werden. Diese Überprüfung wird meist als „Validierung“ von Proteinstrukturen bezeichnet. Die Beobachtung, dass alle Aminosäuren einer Proteinstruktur in erlaubten (energiearmen) Bereichen des Ramachandrandiagramms liegen, bedeutet aber nicht, dass die Konformation dieser Bereiche experimentell oder durch Molekulare Modellierung korrekt bestimmt wurde. Die Konformation ist dann lediglich hinsichtlich dieses Parameters der Stereochemie von Proteinen unauffällig. Weiterhin können einzelne Aminosäuren in energiereichen, nicht erlaubten Bereichen des Ramachadrandiagramms liegen. Dies sind meist für die Faltung oder Aktivität des Proteins essentielle Aminosäuren oder Proteinbereiche. Die Evolution hat dann also keine Möglichkeit gefunden, die gegebene Aktivität (z. B. Enzymkatalyse oder Rezeptoraktivierung) mit diesem Faltungstyp in einer energiearmen Konformation der betreffenden Aminosäure zu realisieren. Andererseits können Aminosäuren in nicht erlaubten Bereichen aber auch falsch modelliert sein (Validierung).
Literatur
[Bearbeiten | Quelltext bearbeiten]- G. N. Ramachandran, C. Ramakrishnan und V. Sasisekharan (1963): Stereochemistry of polypeptide chain configurations. In: J. Mol. Biol. Bd. 7, S. 95–99, PMID 13990617.
Weblinks
[Bearbeiten | Quelltext bearbeiten]Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ WHAT IF homepage. Abgerufen am 26. März 2023.
- ↑ PROCHECK home page. Abgerufen am 26. März 2023.
- ↑ USF - Uppsala Software Factory
