#This source code is public domain
#Author: Christian Schirm
import numpy, scipy.spatial
import matplotlib.pyplot as plt
import imageio
numpy.random.seed(50)
# Covariance matrix
def covMat(x1, x2, covFunc, noise=0):
cov = covFunc(scipy.spatial.distance_matrix(numpy.atleast_2d(x1).T, numpy.atleast_2d(x2).T))
if noise: cov += numpy.diag(numpy.ones(len(cov))*noise)
return cov
# Decomposition of e.g. sum of signals into components
def decompose(xIn, yIn, xOut, covFuncIn, covFuncListOut):
Ckk = covMat(xIn, xIn, covFuncIn, noise=0)
n = len(covFuncListOut)
N = len(xOut)
Cuu = numpy.zeros((n*len(xOut), n*len(xOut)))
Cuk = numpy.zeros((n*len(xOut), len(xOut)))
for i,covOut in enumerate(covFuncListOut):
Cuu[i*N:(i+1)*N, i*N:(i+1)*N] = covMat(xOut, xOut, covOut, noise=0)
Cuk[i*N:(i+1)*N,:] = covMat(xOut, xIn, covOut, noise=0)
CkkInv = numpy.linalg.inv(Ckk)
y = Cuk.dot(CkkInv.dot(yIn))
sigmaSplit = (Cuu - Cuk.dot(CkkInv.dot(Cuk.T)))
return y, sigmaSplit
# Covariance function 1: smooth random signal underground
covFunc1 = lambda d: 2.7**2*numpy.exp(-((d/1.)**2))
# Covariance function 2: periodic signal
covFunc2 = lambda d: 2.7**2*numpy.exp(-0.4*numpy.abs((numpy.sin(numpy.pi*d/2.5))))
# Covariance function 3: white gaussian noise
covFunc3 = lambda d: d*0 + 0.8**2*(numpy.abs(d)<0.00001)
# Covariance function of sum
covFuncSum = lambda d: covFunc1(d) + covFunc2(d) + covFunc3(d)
x = numpy.linspace(0, 10, 300)
# Generate random signales
Y = []
for covFunc in covFunc1, covFunc2, covFunc3:
y = numpy.random.multivariate_normal(x.ravel()*0, covMat(x, x, covFunc))
Y += [y]
# perform decomposition
YSplit = []
YSigma = []
ySplit, sigmaSplit = decompose(x, Y[0]+Y[1]+Y[2], x, covFuncSum, [covFunc1, covFunc2, covFunc3])
YSplit = ySplit.reshape(3,len(x))
# set prior mean of signals 1 and 2
meanShift = 3
YSplit[0] += meanShift
Y[0] += meanShift
YSplit[1] -= meanShift
Y[1] -= meanShift
# Random gaussian process signals
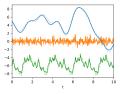
fig = plt.figure(figsize=(4.2,3.0))
for i,c in (2,1), (0,0), (1,2):
plt.plot(x, Y[i], color='C'+str(c), label=u'Prediction',alpha=1)
plt.axis([0,10,-10,10])
plt.xlabel('t')
plt.tight_layout()
plt.savefig('GaussianProcessDecomposition_3RandomSignals.svg')
plt.show()
# Sum of all 3 signals
fig = plt.figure(figsize=(4.2,3.0))
plt.plot(x, (Y[0]+Y[1]+Y[2]), 'r-', label=u'Prediction')
plt.axis([0,10,-10,10])
plt.xlabel('t')
plt.tight_layout()
plt.savefig('GaussianProcessDecomposition_SumOf3Signals.svg')
plt.show()
# plot figures
# Decomposion of sum into single signals
fig = plt.figure(figsize=(4.2,3.0))
for i,c in (2,1), (0,0), (1,2):
plt.plot(x, Y[i], '--', color='C'+str(c), label=u'Prediction',alpha=0.4)
plt.plot(x, YSplit[i], color='C'+str(c), label=u'Prediction',alpha=1)
plt.axis([0,10,-10,10])
plt.xlabel('t')
plt.tight_layout()
plt.savefig('GaussianProcessDecomposition_DecomposedSignals.svg')
plt.show()
# Uncertainty animation
t = numpy.arange(0, 1, 0.02)
covFunc = lambda d: numpy.exp(-(3*numpy.sin(d*numpy.pi))**2) # Covariance function
chol = numpy.linalg.cholesky(covMat(t, t, covFunc, noise=1E-5))
r = chol.dot(numpy.random.randn(len(t), len(sigmaSplit)))
cov = sigmaSplit+1E-5*numpy.identity(len(sigmaSplit))
rSmooth = numpy.linalg.cholesky(cov).dot(r.T).reshape(3,len(x),len(t))
images = []
fig = plt.figure(figsize=(4.2,3.0))
for ti in [0]+range(len(t)):
for i,c in (2,1), (0,0), (1,2):
plt.plot(x, YSplit[i] + rSmooth[i,:,ti], color='C'+str(c), label=u'Prediction',alpha=1)
plt.axis([0,10,-10,10])
plt.xlabel('t')
plt.tight_layout()
fig.canvas.draw()
s, (width, height) = fig.canvas.print_to_buffer()
images.append(numpy.fromstring(s, numpy.uint8).reshape((height, width, 4)))
fig.clf()
# Save GIF animation
imageio.mimsave('GaussianProcessDecomposition_Uncertainty.gif', images[1:])