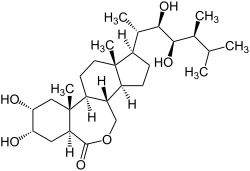
Brassinolid
| Strukturformel | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| |||||||||||||
| Allgemeines | |||||||||||||
| Name | Brassinolid | ||||||||||||
| Andere Namen |
(3aS,5S,6R,7aR,7bS,9aS,10R,12aS,12bS)-10-[(2S,3R,4R,5S)-3,4-Dihydroxy-5,6-dimethyl-2-heptanyl]-5,6-dihydroxy-7a,9a-dimethylhexadecahydro-3H-benzo[c]indeno[5,4-e]oxepin-3-on (IUPAC) | ||||||||||||
| Summenformel | C28H48O6 | ||||||||||||
| Externe Identifikatoren/Datenbanken | |||||||||||||
| |||||||||||||
| Eigenschaften | |||||||||||||
| Molare Masse | 480,7 g·mol−1 | ||||||||||||
| Aggregatzustand |
fest[1] | ||||||||||||
| Sicherheitshinweise | |||||||||||||
| |||||||||||||
| Soweit möglich und gebräuchlich, werden SI-Einheiten verwendet. Wenn nicht anders vermerkt, gelten die angegebenen Daten bei Standardbedingungen. | |||||||||||||

Brassinolid ist ein Pflanzenhormon, welches als erstes Brassinosteroid aus Rapspollen isoliert wurde.[2] Brassinolid spielt eine entscheidende Rolle in pflanzlichen Wachstumsprozessen wie Keimung und Zellteilung.[3]
Biosynthese[Bearbeiten | Quelltext bearbeiten]
Die Biosynthese des Brassinolid geht vom strukturell nah verwandten Membransterol Campesterol aus. Dieses wird zuerst vom Enzym DET2 reduziert, danach erfolgen sukzessive durch Cytochrom-P-450-Enzyme durchgeführte Oxidationen, welche Hydroxylgruppen an das Molekül anbringen. Biologisch sehr bedeutsam ist dabei die C6-Oxidation, bei der am C6-Kohlenstoff ein Keton entsteht. Diese Reaktion allein erhöht die biologische Aktivität um den Faktor 200. Je nachdem, wann diese C6-Oxidation erfolgt, spricht man vom frühen bzw. späten C6-Oxidationsweg. Beide parallelen Synthesepfade konnten in Arabidopsis-Sämlingen nachgewiesen werden, wobei im Licht anscheinend der späte C6-Oxidationsweg dominiert, während im Dunkeln der Frühe vorliegt. Ist die Pflanze nicht fähig, die C6-Oxidation durchzuführen, bildet sich ein sogenannter Dwarf-Phänotyp aus, der starke Wachstumsdefizite zeigt.[4]
Zum Schluss erfolgt die in Arabidopsis durch die beiden homologen Enzyme CYP85A1 und CYP85A2 durchgeführte Baeyer-Villiger-Lactonisierung, das Brassinolid entsteht.[5] Alternativ wurde zudem ein möglicher Synthesepfad über die C27-Brassinosteroide ausgehend vom Cholesterol postuliert.[6]
Wirkungsmechanismus[Bearbeiten | Quelltext bearbeiten]
Signaltransduktion[Bearbeiten | Quelltext bearbeiten]
Brassinosteroide, vor allem das hochwirksame Brassinolid besitzt eine Schlüsselrolle bei der Regulation mehrerer pflanzlichen Prozesse dazu gehört die Keimung, Seneszenz und die Toleranz gegenüber abiotischem und biotischem Stress.[7] Deswegen wurden die Modellorganismen Catharanthus roseus und Arabidopsis seit der Extraktion im Jahre 1979[3] intensiv aus aller Welt erforscht und sehr gut von der Rezeption bis hin zur Genexpression charakterisiert.

Die Rezeption bei Arabidsopis beginnt am BRI1-Rezeptor (Brassinosteroid Insensitive 1), eine Leucin-reiche-Rezeptor-Kinase, welche extrazellulär die Bindung von Brassinolid ermöglicht. Dadurch ändert sich die Konformation und BRI1 wechselwirkt mit der BRI1-Associated-Rezeptor-Kinase 1 (BAK1). Die Komplexformation führt zur gegenseitigen Phosphorylierung und löst eine intrazelluläre Kaskade an Aktivierungen und Repressionen von Kinasen, Phosphatasen und Transkriptionsfaktoren. So werden die BR Signaling Kinasen (BSK1, BSK2 und BSK3) phosphoryliert und aktiviert, welche die Phosphatase BRI1 Supressor1 (BSU1) aktiviert. Diese kann durch die Hydrolyse eine Phosphatgruppe am Brassinosteroid Insensitive 2 (BIN2) abspalten, welches diese Kinase inhibiert. Dadurch kann das Protein Phosphotase 2A (PP2A) die beiden Transkriptionsfaktoren Brassinazole-Resistant-1 (BZR1) und BRI1-EMS-Supressor-1 (BES1) dephosphorylieren, so dass diese sich im Nukleus akkumulieren und die Expression der Zielgene regulieren. Wenn Brassinolid fehlt, verhindert der Regulator BRI1-Kinase-Inhibitor (BKI1) die Wechselwirkung von dem Rezeptor BRI1 und den Co-Rezeptor BAK1. BIN2 ist nicht Inhibiert, sodass die beiden Transkriptionsfaktoren BZR1 und BES1 phosphoryliert werden und mit der Proteineinheit 14-3-3 interagieren. Sodass BES1 und BZR1 sich im Cytoplasma anreichern und proteasomal durch ein 26S Proteasom abgebaut werden. Somit fungiert die BIN2 Kinase als ein wichtiger negativer Regulator, welches die Aktivität der beiden zentralen Transkriptionsfaktoren BES1 und BZR1 inhibiert.[7]

Homöostase[Bearbeiten | Quelltext bearbeiten]
Die beiden Transkriptionsfaktoren BES1 und BZR1 regulieren eine große Anzahl an Genen, welche sich an der Synthese von Hormonen, an Wachstums Prozessen sowie Stressreaktion beteiligen. Dazu kommt die Inhibition der BR Biosynthese, welche schon 15 Minuten nach Brassinolid-Behandlung erkennbar ist.[8] So wird die Expression mehrerer Biosynthetischen Genen wie dem CPD, DWF4 und CYP85A2 Gen inhibiert[7]. Diese codieren für Brassinolid-Biosynthese Enzyme, so codiert das CPD Gen für den Cytochrome P450 Monooxygenase, welches im späten C6-Oxidationsweg das 6-deoxo-Cathasterone zu 6-deoxo-Teasterone oxidiert und im frühen C6-Oxidationsschritt die Reaktion von Cathasterone zum Teasterone katalysiert. Das Enzym CYP85A2 katalysiert den letzten Biosynthese Schritt nämlich die Baeyer-Villiger-Lactonisierung von Castasterone zu Brassinolid. Diese negative Rückkopplung sorgt für die Homöostase des Hormones Brassinolid.
Zellwachstum[Bearbeiten | Quelltext bearbeiten]
Brassinolid induziert Gene, welche das Wachstum fördern wie die TCH4 und SAUR-Ac. Das Gen SAUR-Ac steht für small auxin upregulated RNAs, diese gehören zu den Auxinhormon induzierten Genen und sorgen für die Zellerweiterung.[9] So inhibieren SAURs die Phosphatase PP2C-D, sodass die Plasma Membran H+-ATPase nicht dephosphoryliert wird. Die aktive phosphorylierte Protonen-Pumpe kann somit einen elektrochemischen Gradienten in der Zellwand aufbauen. Die Acidität steigt von 6 auf 4,5-6 und laut der Säurewachstumshypothese sorgt es für die Aktivierung von Expansine, welche die Bindung der Cellulose und Hemicellulose spaltet.[10] TCH4 wurde als xyloglucan endotransglycosylase (XETs) durch Sequenzeanalyse und Enzymaktivität identifiziert. Hauptaufgabe ist die Modifikation von Zellwänden[11]. So bestehen die Hemicellulose aus Xyloglucane, welche aus 1,4-ß- verknüpften Glucosepolymere mit seitlichen 1,6-ß-verknüpften Xylolresten aufgebaut ist. Die Xylogucans können Wasserstoffbrückenbindungen mit der Cellulose Mikrofibrillen eingehen und somit die Zellwand strukturell stabilisieren.[12] Das bedeutet, XET kann die Zellwandstruktur modifizieren. Es spaltet Xyloglucan Moleküle, speichert einen Teil der Energie und verbraucht sie anschließend nach der Ausdehnung zum Verknüpfen wieder. So kann XET während der Zellwanderweiterung die Zellwand weiter auflockern, welche für die Aufnahme von Wasser sorgt. Der dadurch entstehenden Innendruck (Turgor) wird durch die Zellwandausdehnung ausgeglichen, sodass nach der Wiederverknüpfung das Ergebnis eine expandierte Zelle ist.[11]
Cross-Talk mit anderen Phytohormonen[Bearbeiten | Quelltext bearbeiten]
Gibberellin[Bearbeiten | Quelltext bearbeiten]
Brassinolid und Gibberellin sind beide voneinander abhängig. BZR1 in Arabidsopsis hemmt DELLAs ein negativer Regulator für die Gibberellin Transduktion. Somit wird die Aktivität am Promotor zum Binden verringert, sodass Gibberellin die Zellelongation fördert. Und DELLAs Interaktion sorgt für eine Verringerung der BZR1 Bindeaffinität zur DNA.
Jasmonsäure[Bearbeiten | Quelltext bearbeiten]
Brassinolid hemmt die Jasmonsäure (JA) induzierte pathogen defensive für ein idealen Kompromiss zwischen Wachstum und Defensive. Der Transkriptionsfaktor BES1 inhibiert hierbei die Genexpression von PDF1.2a und PDF1.2b, um die der antimikrobielle Proteine Defensins zu reduzieren. Des Weiteren interagiert BES1 mit den Transkriptionsfaktoren MYBs und reduziert deren Aktivität, um die Glucosinolate (GS) Biosynthese zu reduzieren, welche eine wichtige Vorstufe für Abwehrstoffe gegenüber Fressfeinden ist.[13]
Einzelnachweise[Bearbeiten | Quelltext bearbeiten]
- ↑ a b Datenblatt Brassinolide bei Sigma-Aldrich, abgerufen am 27. August 2023 (PDF).
- ↑ J. W. Mitchell, N. Mandava, J. F. Worley, J. R. Plimmer, M. V. Smith: Brassins—a New Family of Plant Hormones from Rape Pollen. In: Nature. Band 225, Nr. 5237, März 1970, S. 1065–1066, doi:10.1038/2251065a0.
- ↑ a b Gerald J. Bishop, Csaba Koncz: Brassinosteroids and Plant Steroid Hormone Signaling. In: The Plant Cell. Band 14, 2002, S. S97–S110, doi:10.1105/tpc.001461.
- ↑ Gerard J. Bishop, Takahito Nomura, Takao Yokota, Kate Harrison, Takahiro Noguchi, Shozo Fujioka, Suguru Takatsuto, Jonathan D. G. Jones, Yuji Kamiya: The tomato DWARF enzyme catalyses C-6 oxidation in brassinosteroid biosynthesis. In: Proceedings of the National Academy of Sciences. Band 96, Nr. 4, 1999, S. 1761–1766, doi:10.1073/pnas.96.4.1761, PMID 9990098.
- ↑ Tae-Wuk Kim ,Jung-Yun Hwang, Young-Soo Kim, Se-Hwan Joo, Soo Chul Chang, June Seung Lee, Suguru Takatsuto, Seopng-Ki Kim: Arabidopsis CYP85A2, a Cytochrom P450, mediates the Baeyer-Villiger Oxidation of Castasterone to Brassinolide in Brassinosteroid Biosynthesis. In: The Plant Cell. 2005, S. 2397–2412, doi:10.1105/tpc.105.033738.
- ↑ Tae-Wuk Kim, Soo Chul Chang, June Seung Lee, Suguru Takatsuto, Takao Yokota, Seong-Ki Kim: Novel Biosynthetic Pathway of Castasterone from Cholesterol in Tomato. In: Plant Physiology. 2004, S. 1231–1242, doi:10.1104/pp.104.043588.
- ↑ a b c Cécile Vriet, Eugenia Russinova, Christophe Reuzeau: From Squalene to Brassinolide: The Steroid Metabolic and Signaling Pathways across the Plant Kingdom. In: Molecular Plant. Band 6, Nr. 6, 2013, S. 1738–1757, doi:10.1093/mp/sst096.
- ↑ Jun-Xian He, Joshua M. Gendron, Yu Sun, Srinivas S. L. Gampala, Nathan Gendron, Catherine Qing Sun, Zhi-Yong Wang: BZR1 Is a Transcriptional Repressor with Dual Roles in Brassinosteroid Homeostasis and Growth Responses. In: Science. Band 307, Nr. 5715, 2005, S. 1634–1638, doi:10.1126/science.1107580, PMID 15681342.
- ↑ Angela K. Spartz, Hong Ren, Mee Yeon Park, Kristin N. Grandt, Sang Ho Lee, Angus S. Murphy, Michael R. Sussman, Paul J. Overvoorde, William M. Gray: SAUR Inhibition of PP2C-D Phosphatases Activates Plasma Membrane H+-ATPases to Promote Cell Expansion in Arabidopsis. In: The Plant Cell. 2014, S. 2129–2142, doi:10.1105/tpc.114.126037.
- ↑ Pyae Phyo, Ying Gu, Mei Hong: Impact of acidic pH on plant cell wall polysaccharide structure and dynamics: insights into the mechanism of acid growth in plants from solid-state NMR. In: Cellulose. Band 26, Nr. 1, 2019, S. 291–304, doi:10.1007/s10570-018-2094-7.
- ↑ a b Wei Xu, Mary M. Purugganan, Diana H. Polisensky, Danuta M. Antosiewicz, Stephen C. Fry, Janet Braam: Arabidopsis TCH4, Regulated by Hormones and the Environment, Encodes a Xyloglucan Endotransglycosylase. In: The Plant Cell. Band 7, Nr. 10, 1995, S. 1555–1567, doi:10.2307/3870019.
- ↑ M. M. Purugganan, J. Braam, S. C. Fry: The Arabidopsis TCH4 Xyloglucan Endotransglycosylase (Substrate Specificity, pH Optimum, and Cold Tolerance). In: Plant Physiology. 1997, S. 181–190, doi:10.1104/pp.115.1.181.
- ↑ Alfredo Kono, Yanhai Yin: Updates on BES1/BZR1 Regulatory Networks Coordinating Plant Growth and Stress Responses. In: Frontiers in Plant Science. Band 11, 2020, doi:10.3389/fpls.2020.617162, PMID 33343611.