„Mimivirus“ – Versionsunterschied
| [gesichtete Version] | [gesichtete Version] |
Ernsts (Diskussion | Beiträge) K Typo |
Ernsts (Diskussion | Beiträge) Überarbeitet auf Basis des en und ru Artikels, sowie weiterer Recherchen zur Linie A |
||
| Zeile 1: | Zeile 1: | ||
<!-- Für Informationen zum Umgang mit dieser Vorlage siehe bitte [[Wikipedia:Viroboxen]]. --> |
<!-- Für Informationen zum Umgang mit dieser Vorlage siehe bitte [[Wikipedia:Viroboxen]]. --> |
||
{{Infobox Virus |
{{Infobox Virus |
||
| Name= Mimivirus |
| Name = Mimivirus |
||
| Bild=Electron microscopic image of a mimivirus - journal.ppat.1000087.g007 crop.png |
| Bild = Electron microscopic image of a mimivirus - journal.ppat.1000087.g007 crop.png |
||
| Bild_legende = [[Elektronenmikroskop|EM]]-Aufnahme eines |
| Bild_legende = [[Elektronenmikroskop|EM]]-Aufnahme eines [[Virion]]s (Virusteilchens) des<br />''[[Mimivirus]]'', links oben das sog. ‚Stargate‘ (Öffnung,<br />durch die die DNA in die Wirtszelle wandert).<ref name=Diesend2018 /> |
||
| Wiss_Name = Acanthamoeba polyphaga mimivirus |
| Wiss_Name = Acanthamoeba polyphaga mimivirus |
||
| Wiss_KurzName = APMV |
| Wiss_KurzName = APMV |
||
| Ordnung = ‚[[NCLDV|Megavirales]]‘ |
| Ordnung = ‚[[NCLDV|Megavirales]]‘ |
||
| Familie = [[Mimiviridae]] |
| Familie = [[Mimiviridae]] |
||
| Subfamilie = ''Mimivirinae‘<ref name="Deeg2018" />''/''‚Megamimivirinae''<ref name=Schulzetal2018 /> |
|||
| Subfamilie = ‚Megamimivirinae''/''Mimivirinae‘<ref name="Schulzetal2018">Frederik Schulz, Lauren Alteio, Danielle Goudeau, Elizabeth M. Ryan, Feiqiao B. Yu, Rex R. Malmstrom, Jeffrey Blanchard, Tanja Woyke: [https://www.nature.com/articles/s41467-018-07335-2#ref-CR38 Hidden diversity of soil giant viruses], in: Nature Communicationsvolume 9, Article number: 4881 (2018) vom 19. November 2018, {{DOI|10.1038/s41467-018-07335-2}}</ref> |
|||
| Gattung = |
| Gattung = Mimivirus |
||
| Spezies = Acanthamoeba polyphaga mimivirus |
| Spezies = Acanthamoeba polyphaga mimivirus |
||
| Subspezies = |
| Subspezies = |
||
| Zeile 20: | Zeile 20: | ||
| ICTV = 00.110.00.01 |
| ICTV = 00.110.00.01 |
||
}} |
}} |
||
Das '''''Mimivirus''''' (''mimi'' ist eine Abkürzung für englisch ''mimicking microbe'') ist eines der größten bisher entdeckten [[Viren]] und gehört wie die ganze Familie ''[[Mimiviridae]]'' zur Gruppe der [[NCLDV|Nucleocytoplasmic large DNA viruses]] (NCLDV), für die vorgeschlagen wurde, sie als ''Megavirales'' in den Rang einer Virusordnung zu erheben. Zur Abgrenzung von anderen putativen ''Mimiviridae''-Unterfamilien ([[Cafeteria-roenbergensis-Virus|Cafeteriavirus]]-Gruppe, [[Klosneuvirus]]-Gruppe – als Unterfamilie ‚''Klosneuvirinae''‘ und der [[OLPG]]-Gruppe – als Unterfamilie ‚''Mesomimivirinae''‘), wurde weiter vorgeschlagen, eine eigene Unterfamilie ''Megamimivirinae'' (oder ''Mimivirinae'') zu bilden, u. a. mit den Mitgliedern ''[[Mamavirus]]'' und ''[[Megavirus]]''.<ref name="Schulzetal2018">Frederik Schulz, Lauren Alteio, Danielle Goudeau, Elizabeth M. Ryan, Feiqiao B. Yu, Rex R. Malmstrom, Jeffrey Blanchard, Tanja Woyke: [https://www.nature.com/articles/s41467-018-07335-2#ref-CR38 Hidden diversity of soil giant viruses], in: Nature Communicationsvolume 9, Article number: 4881 (2018) vom 19. November 2018, {{DOI|10.1038/s41467-018-07335-2}}</ref> Das ''Mimivirus'' befällt [[Amöbe]]n und bringt es mit einem Durchmesser von 400 [[Nanometer|nm]] auf die Größe eines kleinen [[Bakterium]]s. |
|||
'''''Mimivirus''''' ist eine Gattung von [[Viren]] aus der Familie der ''[[Mimiviridae]]'',<ref name=ViralZone>{{cite web|title=ViralZone: Mimivirus|url=http://viralzone.expasy.org/all_by_species/580.html|publisher=ExPASy|accessdate=8. Juli 2019}}</ref> |
|||
Es wurde 1992 bei Forschungsarbeiten über die [[Legionärskrankheit|Legionellose]] in einem Industriekühlturm in [[Bradford]] (England) entdeckt, wo es sich in der Amöbe ''[[Acanthamoeba polyphaga]]'' vermehrt. Im Jahr 2003 wurde es an der ''Université de la Méditerranée'' in [[Marseille]] von einer Arbeitsgruppe um [[Didier Raoult]] identifiziert.<ref>B. La Scola, S. Audic, C. Robert, L. Jungang, X. de Lamballerie, M. Drancourt, R. Birtles, J. M. Claverie, D. Raoult: ''A giant virus in amoebae.'' In: ''Science.'' 299, 2003, S. 2033. PMID 12663918</ref> Wegen seiner Größe und seiner äußeren Ähnlichkeit mit bestimmten Bakterien ([[Kokken]]) hielt man es zunächst für ein [[grampositiv]]es Bakterium und nannte es ''Bradfordcoccus''. Als man den Irrtum erkannte, benannte man das neu entdeckte Virus ''Mimicking Virus'', täuschendes Virus, und schließlich kurz ''Mimivirus''. |
|||
denen [[Amöben]] als natürliche Wirte dienen. Mit der Familie ''Mimiviridae'' gehört ''Mimivirus'' zu den Riesenviren in der Gruppe der [[NCLDV|Nucleocytoplasmic large DNA viruses]] (NCLDV), für die vorgeschlagen wurde, sie als ''Megavirales'' in den Rang einer Virusordnung zu erheben.<ref name="YWK2014_Megavirales" /> |
|||
In der Gattung ''Mimivirus'' gibt es mit Stand März 2019 eine einzige vom [[International Committee on Taxonomy of Viruses]] (ICTV) bestätigte Art '''''Acanthamoeba polyphaga mimivirus''''' ('''APMV'''), die daher auch die Typusart ist.<ref>[[International Committee on Taxonomy of Viruses|ICTV]]: {{cite web |url=https://talk.ictvonline.org/files/master-species-lists/m/msl/8266 |title=Master Species List 2018b.v2 |accessdate=2019-08-06}} MSL #34v</ref> |
|||
Im Oktober 2004 wurde die Struktur seines Erbguts in der Fachzeitschrift ''Science'' veröffentlicht.<ref>D. Raoult, S. Audic, C. Robert, C. Abergel, P. Renesto, H. Ogata, B. La Scola, M. Suzan, J. M. Claverie: ''The 1.2-Mb Genome Sequence of Mimivirus.'' In: ''Science.'' 306, 2004, S. 1344–1350. PMID 15486256</ref> Seine [[Desoxyribonukleinsäure|DNA]] ist 800 nm lang, verfügt über etwa 1,2 Millionen Basenpaare und 1260 [[Gen]]e mit einem Anteil von nur zehn Prozent [[Nichtcodierende Desoxyribonukleinsäure|nichtcodierender DNA]]. Bei der Analyse stieß man auf Gene, die bisher nur von zellulären Organismen bekannt waren. |
|||
In der Umgangssprache und in älterer Literatur wird APMV üblicherweise nur als ''Mimivirus'' bezeichnet. Es gibt jedoch eine ganze Reihe phylogenetisch verwandter großer Viren, die als weitere Mitglieder dieser Gattung vorgeschlagen sind. |
|||
== Entdeckung und Forschungsgeschichte == |
|||
Auf Grund der außergewöhnlich komplexen genetischen Ausstattung des Virus stellt sich für einige Forscher die Frage neu, wo die Grenze zwischen ''[[Leben|belebter]]'' und ''unbelebter [[Natur]]'' verlaufe, also wie „[[Lebewesen]]“ zu definieren ist. |
|||
{{Anker|Entdeckung}}Das ''Mimivirus'' APMV wurde 1992 bei Forschungsarbeiten über die [[Legionärskrankheit]] (Legionellose) in einem Industriekühlturm in [[Bradford]] (England) entdeckt, wobei man feststellte, dass es sich in der Amöbe ''[[Acanthamoeba]] polyphaga'' vermehrt. Im Jahr 2003 wurde es an der ''Université de la Méditerranée'' in [[Marseille]] von einer Arbeitsgruppe um [[Didier Raoult]] identifiziert.<ref name=LaScola2003 /> Mit einem Durchmesser von 400 [[Nanometer|nm]] haben die Viruspartikel ([[Virion]]en) von APMV die Größe von kleinen [[Bakterium|Bakterien]]. Wegen dieser Größe und der äußeren Ähnlichkeit mit kugelförmigen Bakterien ([[Kokken]]) hielt man es zunächst für ein [[grampositiv]]es Bakterium und nannte es ''Bradfordcoccus''. Als man den Irrtum erkannte, benannte man das neu entdeckte Virus in Anspielung auf seine Größe und Färbungseigenschaften ''Mimicking Virus'', täuschendes Virus. Schließlich wurde daraus kurz ''Mimivirus'', mit dem Namensteil ''mimi'' als eine Abkürzung für {{enS|mimicking microbe}}. Im Oktober 2004 wurde dann von Didier Raoult und Kollegen die Struktur seines Erbguts in der Fachzeitschrift ''Science'' veröffentlicht.<ref name=Raoult2004 /> |
|||
Das gleiche Team, das das APMV entdeckte, entdeckte später ein etwas größeres Virus, das ''[[Acanthamoeba castellanii mamavirus]]'' (ACMV, kurz ''Mamavirus'') zusammen mit dem [[Sputnik-Virus|Sputnik-Virophagen]], der es infiziert. ACMV und APMV sind so nahe verwandt, dass sie gewöhnlich in dieselbe Gattung ''Mimivirus'' gestellt werden.<ref name=Pearson2008 /> |
|||
Bis 2013, als ein noch größeres Virus, das ''[[Pandoravirus]]'', beschrieben wurde, wiesen die Viren der Gattung ''Mimivirus'' den größten Kapsiddurchmesser aller bekannten Viren auf;<!--{{cite news|title=World's biggest virus found in sea off Chile|url=https://www.telegraph.co.uk/science/88--nicht mehr verfügbar--> inzwischen wird es aber auch vom nahe Verwandten ''[[Megavirus chilensis]]'' und vom ''[[Pithovirus]]'' übertroffen. |
|||
== Aufbau == |
|||
[[Datei:Cryoelectron microscopy of the Giant Mimivirus - journal.pbio.1000092.g005.png|thumb|upright=1.5|right| |
|||
Rekonstruktion der Struktur des ''Mimivirus''-Kapsids auf Basis von [[Kryoelektronenmikroskopie|kryoelektronenmikroskopischen]] Daten. Die Farbe kodiert den Abstand von der Mitte des Kapsids: grau - 0-180 nm, rot - 180-210 nm, Regenbogenfarben von rot nach blau - 210-270 nm. Maßstab: 100 nm<ref name=Xiao2009 /> Bild A zeigt wieder deutlich das ‚Stargate’, Bild B zeigt die relative Länge der Strahlen, Bild E zeigt die Absenkung des Nukleokapsids unter dem Scheitelpunkt des Stargates.]] |
|||
<!--[[Datei:Mimivirus svg.svg|mini|right|upright=2|Schemazeichnung eines ''Mimivirus''-Virions]] -- Fehler: Das Stargate fehlt. Die DNA besteht nach der Zeichnung aus 2 Strängen, was nicht belegt ist.--> |
|||
[[Datei:Mimivirus fibers - journal.pbio.1000092.g007.png|thumb|right| |
|||
'''A:''' [[Rasterkraftmikroskop]]-Aufnahme mehrerer Oberflächenfasern, die an einem gemeinsamen zentralen Punkt befestigt sind.<br /> |
|||
'''B''': AFM-Aufnahme von zwei abgelösten Oberflächenfasern des ''Mimivirus''.<br /> |
|||
'''C:''' [[Cryo-EM]]-Bild eines ''Mimivirus'' nach teilweisem Abbau der Fibrillen mittels [[Bromelain]].<br /> |
|||
'''D:''' AFM-Bild von inneren Fasern des ''Mimivirus''. |
|||
]] |
|||
Die [[Kapsid]]e der [[Virion]]en (Viruspartikel) von APMV erscheinen unter einem [[Elektronenmikroskop]] sechseckig, daher ist die Kapsidgeometrie |
|||
[[ikosaedrisch]].<ref name=Xiao2009 /> |
|||
Es scheint keine äußere virale Hülle zu geben, was darauf hindeutet, dass das Virus die [[Wirtszelle]] nicht durch [[Exozytose]] verlässt.<ref name=SuzanMonti2006 /> |
|||
Das Hauptprotein des ''Mimivirus''-Kapsids <!--ist ein Produkt des L425-Gens -- zu technisch, kein QV -- und -->besteht aus zwei Domänen vom Biskuitrollen-Typ ({{enS|Jelly roll fold}}). Dieses Protein bildet [[Trimer|homotrimere]] [[Kapsomer]]e als Organisationseinheit der Kapside. Die Kapsomere sind hexagonal in Form von „Gänseblümchen“ gepackt: Sechs Kapsomere umgeben eine Vertiefung zwischen ihnen.<ref name=Xiao2009 /><!-- dito -- Auch ein Strukturprotein des L410-Kerns befindet sich im Kapsid.--><ref name=Claverie2009 /> |
|||
Die Virionen von ''Mimivirus'' habe einen Kapsiddurchmesser von 400 [[Meter#nm|nm]]. |
|||
=== Fibrillen === |
|||
Das Kapsid ist mit einer kompakten Schicht von [[Fibrille]]n bedeckt. |
|||
Die aus der Oberfläche des Kapsids herausragenden Proteinfilamente haben eine Länge von etwa 100 nm (80-120 nm), und bringen damit die Gesamtlänge eines Virions auf 600 nm.<ref name=Diesend2018 /> |
|||
Abweichungen in der wissenschaftlichen Literatur lassen die Zahlen als sehr ungenau erscheinen, wenn zum Beispiel die ‚Größe’ des Virions gelegentlich als irgendwo zwischen 400 und 800 nm angegeben wird. Abgesehen von Unterschieden zwischen den einzelnen Virusstämmen in der Gattung ''Mimivirus'' ist manchmal die Gesamtgröße mit Filamenten, und manchmal der reine Kapsiddurchmesser angegeben. |
|||
Untersuchungen dieser Filamente unter Verwendung eines [[Rasterkraftmikroskop]]s haben ergeben, dass diese häufig an einer gemeinsamen Tragstruktur befestigt sind und mit einer kleinen Kugel enden. |
|||
Es ist jedoch immer noch nicht bekannt, an welchen Teilen der Kapsidoberfläche sie befestigt sind.<ref name=Xiao2009 /> |
|||
Die Proteinfilamente sind resistent gegen [[Protease]]n, außer sie werden mit [[Lysozym]] behandelt. Daher scheinen die Filamente mit [[Peptidoglycan]] beschichtet zu sein. |
|||
Dies steht in guter Übereinstimmung mit der Tatsache, dass das ''Mimivirus'' durch die ''Gram-Methode'' gefärbt wird. |
|||
Es wird angenommen, dass die stark glykosylierte Oberfläche der Filamente eine Rolle bei der Annäherung an Wirtsamöben spielt.><ref name=Klose2010 /> |
|||
=== Stargate === |
|||
Auffällig ist die fünfeckige, sternförmige Struktur an einer der Ecken des Kapsids, das so genannte ‚Stargate‘ (englisch für „Sternentor“). Blickt man direckt auf diesen Eckpunkt (den Mittelpunkt des Sterns) so scheinen zwischen dessen Strahlen fünf dreieckige Flächen zu liegen. |
|||
Die Strahlen haben eine Breite von ungefähr 50 nm, eine Dicke von 40 nm und eine Länge von 200 nm; sie erreichen fast die benachbarten Eckpunkte der ikosaedrischen Kapsids. |
|||
Das Vorhandensein dieser Struktur verändert die Geometrie des Kapsids, indem seine Geometrie von der idealen Ikosaederform abweicht: Tatsächlich verläuft bei genauer Betrachtung nur eine einzige Achse mit Fünfstrahlsymmetrie durch das Virion, die durch den Mittelpunkt des Sterns (genannt Scheitelpunkt) verläuft.<ref name=Xiao2009 /> |
|||
Die Symmetrie des Kapsids wird unterschiedlich angegeben mit T=972–1141 oder T=1200.<ref name=ViralZone /> |
|||
Da auf der Oberfläche der Sternstruktur keine hexagonal geordneten Vertiefungen zu beobachten sind, wird vermutet, dass es sich bei dieser um ein Protein handelt, das sich vom Hauptkapsidprotein unterscheidet.<ref name="Xiao2009" /> |
|||
Das Stargate spielt eine besondere Rolle bei der Infektion der Wirtszelle: |
|||
Während der Infektion öffnet sich der „Verschluss“ am Scheitelpunkt und es erfolgt die Freisetzung des viralen Kerns (mit DNA und vorgefertigten Proteinen) aus dem Kapsid in das [[Zytosol]] der Wirtszelle (per [[Phagocytose]]). |
|||
Das ist der Grund, warum die Sternstruktur als „Sternentor“ ({{enS|stagate}}) bezeichnet wird.<ref name=Zauberman2008 /> |
|||
=== Nukleokapsid === |
|||
Das ''Mimivirus'' hat mehrere morphologische Merkmale mit allen Mitgliedern der NCLDV-Virengruppe gemeinsam. |
|||
Unmittelbar unter dem Kapsid des ''Mimivirus'' befinden sich beispielsweise zwei elektronendichte Schichten, die als Membranen gedeutet werden.<ref name="Klose2010" /> |
|||
Unter diesen Membranen befindet sich eine etwa 7 nm dicke Proteinhülle, in der die lineare doppelsträngige DNA des Virus eingeschlossen ist. |
|||
Dieser kondensierte Zentralkern des Virions erscheint unter dem Elektronenmikroskop als dunkler Bereich, das sogenannte ‚Nukleokapsid‘. |
|||
In diesem Bereich befindet sich das große Genom des Virus, daneben auch [[mRNA]]s und vorgefertigte [[Protein]]e. |
|||
Da alle anderen NCLDVs eine interne [[Lipid]]schicht besitzen, die den zentralen Kern umgibt, vermutet man das auch bei ''Mimivirus''. |
|||
Die Wände des Nukleokapsids liegen etwa 30 nm hinter den Wänden des Kapsids zurück, im Bereich der Sternstruktur (dem Stargate) ist die Oberfläche des Nukleokapsids zusätzlich abgesenkt.<ref name="Xiao2009" /> |
|||
Es wird angenommen, dass der Raum zwischen der Spitze der Sternstruktur und dem Nukleokapsid mit [[Hydrolyse|hydrolytischen]] Enzymen gefüllt ist, die für das Eindringen des Virus in die Wirtszelle erforderlich sind. |
|||
Zwischen dem Kapsid und dem Nukleokapsid wurden interne Proteinstränge entdeckt, die anscheinend die gegenseitige räumliche Positionierung der beiden Teile zueinander stabilisieren.<ref name="Klose2010" /> |
|||
== Genom == |
|||
APMV hat damit im Vergleich zu den meisten anderen Viren ein großes und komplexes [[Genom]], das eis einem einzelnen linearen [[DNA]]-Doppelstrang (dsDNA) besteht. Die DNA von APMV ist 800 nm lang, verfügt über 1.181.404 [[Basenpaar]]e und <!-- offenbar veraltet -- 986 bis -->1260 [[Offener Leserahmen|Offene Leserahmen]] (ORFs, {{enS|open reading frames}}), darunter geschätzt 911–979 [[Gen]]e, die [[Protein]]e [[Genetischer Code|kodieren]]. |
|||
Dies geht weit über die Mindestausstattung von 4 Genen hinaus, die für ein Virus erforderlich sind, wie etwa bei den Phagen [[Enterobakteriophage MS2|MS2]] und [[Enterobakteriophage Qbeta|Qβ]].<ref name=Prescott1993 /> |
|||
Detaillierte Studien zum Genom laufen noch: Sequenzfehler werden korrigiert und neue Leserahmen entdeckt.<ref name=Claverie2 /> |
|||
Der Anteil von [[Nichtcodierende Desoxyribonukleinsäure|nichtcodierender DNA]] beträgt nur etwa 9,5 bis 10 %. Offene Leserahmen sind durch Lücken von ungefähr 157 Nukleotidpaaren getrennt. Zwei DNA-Abschnitte mit der Bezeichnung R ({{enS|right}} - rechts) und L ({{enS|left}} - links) kodieren ungefähr die gleiche Anzahl von Genen (450 bzw. 465, gemäß Daten von 2010). Der [[GC-Gehalt]] ist mit 28 % niedrig. In der Nähe der Enden des DNA-Moleküls wurden ‚Invertierte Wiederholungen‘ ({{enS|inverted repeats}}) mit 617 Nukleotidpaaren gefunden. Es wird vermutet, dass die gegenseitige Wechselwirkung dieser Stellen zur Bildung einer Q-Struktur führen kann - zirkuläre DNA mit zwei kleinen Fortsätzen.<ref name=Claverie2 /> |
|||
Bei der Analyse stieß man auf mindestens 21 Gene mit [[Homologie (Biologie)|Homologie]] zu bekannten Proteinen, darunter solche, die man bis dato von keinem anderen Virus, sondern nur von zellulären Organismen kannte, inklusive [[Aminoacyl-tRNA-Synthetase]].<ref name=Raoult2004 /><ref name=LaScola2003 /><ref name=Claverie2006 /> |
|||
Wie andere Riesenviren (NCLDVs) enthält das ''Mimivirus'' mehrere Gene für den Zucker-, Lipid- und Aminosäurestoffwechsel. Es gab auch Stoffwechselgene, die zuvor in keinem anderen Virus gefunden wurden.<ref name=SuzanMonti2006 /> |
|||
Aus gereinigten Virionen konnten mehrere [[mRNA]]-[[Transkription (Biologie)|Transkripte]] gewonnen werden. |
|||
Wie schon bei anderen NCLDVs wurden insbesondere Transkripte für [[DNA-Polymerase]], ein Kapsidprotein und ein TFII-ähnlicher [[Transkriptionsfaktor]] gefunden werden. Es wurden jedoch auch drei verschiedene [[Aminoacyl-tRNA-Synthetase]]-Transkripte und vier unbekannte mRNA-Moleküle gefunden, die für das ''Mimivirus'' spezifisch sind. Diese vorverpackten Transkripte können ohne virale Genexpression translatiert werden und sind wahrscheinlich für die Replikation von ''Mimivirus'' erforderlich. Andere DNA-Viren, wie das ''[[Humanes Cytomegalievirus|Humane Cytomegalievirus]]'' und das ''[[Herpes-simplex-Virus]]'' Typ 1, enthalten ebenfalls gepackte mRNA-Transkripte.<ref name=SuzanMonti2006 /> |
|||
''Mimivirus'' ist eines der wenigen dsDNA-Viren, in deren Genom eine Intein-kodierende Sequenz nachgewiesen wurde. [[Inteine]] sind Proteindomäne, die ihre eigene Entfernung von einem Trägermolekül und die anschließende Verknüpfung der gebildeten Enden [[Katalysator|katalysieren]]. |
|||
Eine solche Sequenz ist im ''Mimivirus''-Gen für DNA-Polymerase B vorhanden.<ref name=Ogata2005 /><!-- |
|||
{| class="wikitable" style="text-align:center" |
|||
|- |
|||
! Genus !! Structure || [[Capsid#Triangulation number|Symmetry]] !! Capsid !! Genomic arrangement !! Genomic segmentation |
|||
|- |
|||
|Mimivirus || Icosahedral || {{nobr|''T'' {{=}} 972–1141}} or {{nobr|''T'' {{=}} 1200}} ({{nobr|''h'' {{=}} 19 ± 1}}, {{nobr|''k'' {{=}} 19 ± 1}}) || || Linear || Monopartite |
|||
|}--> |
|||
Auf Grund der außergewöhnlich komplexen genetischen Ausstattung des Virus stellt sich für einige Forscher die Frage neu, wo die Grenze zwischen ''[[Leben|belebter]]'' und ''unbelebter [[Natur]]'' verlaufe, also wie „[[Lebewesen]]“ zu definieren ist.<br clear=all> |
|||
== Vermehrungszyklus == |
|||
[[Datei:Mimivirus - intracellular viral Factories and DNA packaging - journal.pbio.0060114.g006.png|thumb|300px| |
|||
''Mimivirus'' - Intrazelluläre [[Viroplasma|Virusfabriken]] und DNA-Verpackung<ref name=Zauberman2008 /><br /> |
|||
'''A)''' [[Transmissionselektronenmikroskop|TEM-Aufnahme]] einer intrazellulären Virusfabrik, mit [[Virion|Viruspartikeln]] von ''Mimivirus'' in verschiedenen Montagestadien<br /> |
|||
- '''VF:''' Virusfabrik (Viroplasma)<br /> |
|||
- '''Cyt:''' [[Cytoplasma]]<br /> |
|||
- '''violette Pfeile:''' leere, noch faserlose Virionen im Anfangsstadium der Zusammensetzung, in unmittelbarer Nähe zur Peripherie der Virusfabrik auftretend<br /> |
|||
- '''blaue Pfeile:''' Teilweise montierte leere, faserlose Virionen<br /> |
|||
- '''gelbe Pfeile:''' reife, faserbedeckte Virionen, die sich jetzt weiter von der Virusfabrik entfernt befinden als die unreifen Partikel<br /> |
|||
- '''rote Pfeile:''' bei mehreren Viruspartikeln ist ein ‚Stargate’, das sich regelmäßig an der [[distal (Anatomie)|distalen]] Stelle der Fabrik befindet, zu erkennen<br /> |
|||
'''B)''' und '''C)''' TEM-Aufnahme der ''Mimivirus''-Partikel<br /> |
|||
- '''grüne Pfeile:''' ''Mimivirus''-Partikel im Stadium des ‚DNA-Packaging’ (DNA-Verpackung)<br /> |
|||
- '''rote Pfeile:''' zwei Kanten eines Stargates gegenüber der Stelle der DNA-Verpackung |
|||
]] |
|||
Die Einzelheiten und die verschiedenen Stadien im Replikationszyklus von 'Mimivirus'', die offensichtlichste Bindung an die Zelloberfläche und den Eintritt, die Freisetzung des Viruskerns, die DNA-Replikation, die Transkription, die Translation, und schließlich den Zusammenbau und die Freisetzung von Tochter-Virionen, sind noch nicht ausreichend bekannt. Die Wissenschaftler haben jedoch den oben angegebenen allgemeinen Überblick anhand elektronenmikroskopischer Aufnahmen infizierter Zellen erstellt. |
|||
Alle Stadien des Vermehrungszyklus verlaufen im [[Cytoplasma]] der Wirtszelle.<ref name=Mutsafi2010 /> |
|||
Die Infektion der Amöbe mit einem Mimivirus erfolgt vermutlich nach folgendem Szenario: |
|||
# Die ''Mimivirus''-Virionen ähneln in ihrer Größe und dem Vorhandensein charakteristischer [[Polysaccharide]] auf der Oberfläche Bakterien (siehe Gram-Färbung, Name). Sie werden daher von der Amöbe als Nahrung während eines [[Endozytose]]prozesses absorbiert. Die Polysaccharide fungieren dabei als chemischer Rezeptor und leiten die Anlagerung ein. Als Ergebnis der Endozytose befinden sich die Virionen in [[Endosom]]en innerhalb der Zelle. |
|||
# Die Proteinfilamente werden in den Endosomen teilweise [[Lyse (Biologie)|lysiert]], wodurch das Kapsid mit der Endosomenmembran in Wechselwirkung treten kann. |
|||
# ~2 Stunden nach der Infektion: Das Kapsid öffnet sich im Bereich der Sternstruktur (Stargate), die innere Membran fusioniert mit der Endosomenmembran und der Inhalt des Kapsids wird in das Cytoplasma freigesetzt. |
|||
# Nachdem das Kernteilchen (der innere Teil des Nukleokapsids) ins Cytoplasma ausgetreten ist, beginnt aufgrund der Anwesenheit des viralen Transkriptionsapparats die Synthese der viralen [[mRNA]]. Diese mRNAs reichern sich im Inneren des Kernpartikels in Form von Granula an.<ref name=Mutsafi2010 /><!-- Es wird angenommen, dass Gene, die unter der Kontrolle des AAAATTGA-Promotors stehen, zuerst unter der Wirkung von viraler RNA-Polymerase transkribiert werden (siehe Abschnitt Genom)--> Äußerlich betrachtet scheint das Virus verschwunden und alles in der Zelle sieht normal aus (Dunkelphase, ({{enS|eclipse phase}}). |
|||
# 4-5 Stunden nach der Infektion: Die virale DNA verlässt das Kernteilchen und wird entpackt, so dass die Replikation beginnen kann. Infolgedessen entsteht neben der leeren Hülle des Kernpartikels eine sogenannte „[[Viroplasma|Virusfabrik]]“ - ein Ort für die Synthese der einzelnen Komponenten der Virionen und ihren anschließenden Zusammenbau.<!--{{cite web|url=http://www.membrana.ru/particle/3930|title=Открыто уникальное поведение крупнейшего вируса|publisher=Membrana|date=2010-04-12|accessdate=2013-02-07|lang=ru|archiveurl=https://www.webcitation.org/6EL52uJ85?url=http://www.membrana.ru/particle/3930|archivedate=2013-02-10|deadlink=no}}--> Wenn mehrere Viruspartikel in die Zelle gelangt sind, verschmelzen die von ihnen gebildeten „Fabriken“ beim Wachstum zu einer einzigen. Man erkennt jetzt kleine Ansammlungen in einigen Bereichen der Zelle. |
|||
# 6–9 Stunden nach der Infektion: Zusammensetzung (Assemblierung) der Kapside mit gleichzeitiger DNA-Packung an der Peripherie der „Virusfabriken“. Eine ungewöhnliche Eigenschaft des ''Mimivirus'' ist, dass DNA gepackt ist<!-- und das Kapsid durch zwei verschiedene Öffnungen verlässt -- ?? -->.<ref name=Zauberman2008 /> Die ''Mimivirus''-Virionen werden in der Zelle deutlich sichtbar. |
|||
# 14-24 Stunden nach der Infektion: Die Amöbenzellen werden lysiert, d. h. sie platzen auf und die Virionen werden freigesetzt. Pro Wirtszelle werden so mehr als 300 Einheiten erzeugt.<ref name=Claverie2 /><ref name=SuzanMonti2006 /> |
|||
Die Übertragung geschieht durch passive Diffusion<ref name=ViralZone /><!-- |
|||
{| class="wikitable" style="text-align:center" |
|||
|- |
|||
! Genus !! Host details !! Tissue tropism !! Entry details !! Release details !! Replication site !! Assembly site !! Transmission |
|||
|- |
|||
|Mimivirus||Zooplankton||None||Unknown||Unknown||Cytoplasm||Nucleus||Passive diffusion |
|||
|}--> |
|||
== Wirte und Pathogenität == |
|||
Der erste bekannte ''Mimivirus''-Wirt ist die [[Amöbe]] ''[[Acanthamoeba]] polyphaga'' ([[Amoebozoa]]). Es hat sich gezeigt, dass nur andere Vertreter der Gattung ''Acanthamoeba'', ''A. castellanii'' und ''A. mauritaniensis'' , als Wirte dieses Virus dienen können, keine Zellen anderer einzelliger oder mehrzelliger Organismen.<ref name=SuzanMonti2006 /> |
|||
Es wurde spekuliert, dass ''Mimivirus'' Erreger bestimmter Formen von [[Lungenentzündung]] (Pneumonia) sein könnte. Dies beruht hauptsächlich auf indirekten Nachweisen in Form von Antikörpern gegen das bei Lungenentzündungspatienten entdeckte Virus.<ref name=LaScola2005 /> Aufgrund der wenigen bisherigen Veröffentlichung ist die Einstufung des ''Mimivirus'' als möglicher Krankheitserreger ist derzeit jedoch schwierig. Ein großer Teil der Fälle von Lungenentzündug verläuft ohne feststellbare Ursache.<ref name="Marrie1989">{{cite journal |author=Marrie TJ, Durant H, Yates L |title=Community-Acquired Pneumonia Requiring Hospitalization: 5-Year Prospective Study|journal=Reviews of Infectious Diseases|volume=11|issue=4|pages=586–99|year=1989|pmid=2772465 |doi=10.1093/clinids/11.4.586}}</ref> Zwar wurde ein ''Mimivirus'' bei iner an Lungenentzündung leidenden Tunesierin isoliert,<ref name="pmid23709652">{{cite journal |author=Saadi H, Pagnier I, Colson P, Cherif JK, Beji M, Boughalmi M, Azza S, Armstrong N, Robert C, Fournous G, La Scola B, Raoult D |title=First isolation of Mimivirus in a patient with pneumonia |journal=Clinical Infectious Diseases |volume=57 |issue=4 |pages=e127–34 |date=August 2013 |pmid=23709652 |doi=10.1093/cid/cit354 }}</ref> |
|||
und es gibt Hinweise darauf, dass ''Mimivirus'' [[Makrophagen]] infizieren kann. Unter experimentellen Bedingungen wurde beobachtet, dass ''Mimivirus'' humane Makrophagen infizieren kann, d. h. via [[Phagocytose]] in die Zellen eindringen, und sich dort replizieren kann.<ref name=Vincent2010 /><ref name="Ghigo">{{cite journal |author=Ghigo E., Kartenbeck J., Lien P., Pelkmans L., Capo C., Mege J. L., Raoult D. |title=Ameobal pathogen mimivirus infects macrophages through phagocytosis |url=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2398789/ |journal=PLoS Pathog |year=2008 |volume=4 |issue=6 |pages=e1000087 |doi=10.1371/journal.ppat.1000087 |pmid=18551172 }}</ref> Außerdem wurden in mehreren Studien bei einer kleinen Anzahl von Patienten mit Lungenentzündung Antikörper gegen das Mimivirus gefunden.<ref name="LaScola2005">{{cite journal |author=La Scola B., Marrie T. J., Auffray J. P., Raoult D. |title=Mimivirus in pneumonia patients |url=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3298252/ |journal=Emerg Infect Dis. |year=2005 |volume=11 |issue=3 |pages=449—452 |pmid=1575756 3}}</ref><ref name="Berger">{{cite journal |author=Berger P., Papazian L., Drancourt M., La Scola B., Auffray J. P., Raoult D.|title=Ameba-associated microorganisms and diagnosis of nosocomial pneumonia |url=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3373093/ |journal=Emerg Infect Dis|year=2006 |volume=12 |issue=2 |pages=248—255 |pmid=16494750 }}</ref> |
|||
Es wurde auch ein Einzelfall einer Lungenentzündung in einem Laborassistenten beschrieben, der mit Kulturen dieses Virus arbeitete. Der Gehalt an Antikörpern gegen Mimivirus in seinem Blut war ebenfalls erhöht.<ref name="Raoult">{{cite journal |author=Raoult D., Renesto P., Brouqui P.|title=Laboratory infection of a technician by mimivirus |url=http://annals.org/article.aspx?articleid=723087 |journal=Ann Intern Med |year=2006 |volume=144 |issue=9 |pages=702—703 |pmid=16670147 }}</ref> |
|||
Das Vorhandensein von Antikörpern gegen das Virus an sich ist jedoch kein Hinweis auf seine Pathogenität. Es ist möglich, dass das ''Mimivirus'' einfach starke immunogene Eigenschaften aufweist, d. h. eine deutliche Immunantwort auslöst.<ref name="Claverie2" />. |
|||
Auch war es in keinem der registrierten Fälle möglich, das Virus in seiner reinen Form aus Proben von Flüssigkeiten zu isolieren, die von Patienten erhalten wurden.<ref name="Vanspauwen2012">{{cite journal |author=Vanspauwen M. J. ''et al.'' |title=Infections with mimivirus in patients with chronic obstructive pulmonary disease |url=http://www.sciencedirect.com/science/article/pii/S0954611112003150 |journal=Respiratory Medicine |year=2012 |volume=106 |issue=12 |pages=1690—1694 |doi=10.1016/j.rmed.2012.08.019 }}</ref> |
|||
==Systematik == |
|||
=== Äußere Systematik === |
|||
Die Gattung ''Mimivirus'' und einige andere – vom ICTV mit Stand März 2019 noch nicht bestätigte – genetisch ähnliche Gattungen und Spezies der Familie ''Mimiviridae'' (wie zum Beispiel ''[[Mamavirus]]'', ''[[Megavirus]]'' und ''[[Moumouvirus]]'') bilden eine als Gruppe I bezeichnete [[Klade]] der Mimiviren im weiteren Sinn. |
|||
Für diese wurde vorgeschlagen, sie als ''Megamimivirinae''<ref name=Schulzetal2018 /> oder auch ''Mimivirinae''<ref name="Deeg2018" /> in den Rang einer Unterfamilie zu erheben und so von anderen ebenfalls vorgeschlagenen Unterfamilien der ''Mimiviridae'' abzugrenzen. Solche wären: |
|||
* [[Cafeteria-roenbergensis-Virus|Cafeteriavirus]]-Gruppe (Gruppe II) |
|||
* [[Klosneuvirus]]-Gruppe – als mögliche Unterfamilie ‚''Klosneuvirinae''‘ |
|||
* [[OLPG]]-Gruppe (Gruppe III) – als Unterfamilie ‚''Mesomimivirinae''‘ |
|||
Innerhalb dieser putativen Unterfamilie zeichnen sich die folgenden Linien ab: |
|||
:* Linie A: ''Mimivirus''-Gruppe: Mimiviren im engeren Sinn |
|||
:* Linie B: ''[[Moumouvirus]]''-Gruppe (Moumouviren) |
|||
:* Linie C: Courdo11-Gruppe mit ''[[Megavirus chilensis]]'' (Spezies), dazu Courdo11-Virus |
|||
:* ''[[Tupanvirus]]''-Gruppe (Tupanviren) |
|||
Da bisher (März 2019) vom ICTV lediglich die Gattung ''Mimivirus'' bestätigt wurde, bleibt zur Zeit noch offen, welche Kandidaten für diese putative Unterfamilie dieser Gattung einmal zugeordnet werden, und für welche eigene Gattungen einzurichten sind. Sicher scheint aber, dass die Vertreter der Linie A in jedem Fall zur Gattung ''Mimivirus'' gehören. |
|||
=== Innere Systematik === |
|||
Systematik der Linie A (Mimiviren im engeren Sinn): |
|||
* Gattung ''Mimivirus'' |
|||
:* Spezies: Acanthamoeba polyphaga mimivirus (APMV)<ref name=NCBIApMV>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?mode=Tree&id=212035 Acanthamoeba polyphaga mimivirus] (Spezies)</ref> (vom ICTV mit Stand März 2018 bestätigt) – Fundort: [[Bradford]], England, UK<ref name=Diesend2018 /> |
|||
::* Mimivirus Bombay (alias Bombay-Virus)<ref name=NCBIApMV /><ref name=Diesend2018 /> – Fundort: [[Mumbai]], Indien |
|||
::* Mimivirus shirakomae<!-- sic! das Virus mit e--> (alias Shirakomae-Virus) – Fundort: [[Sakuho|Shirakoma Pond]]<!--siehe en WP-->, [[Nagano]]<!--District-->, Japan<ref name=Diesend2018 /><ref name=Takemura2016 /> |
|||
::* Mimivirus kasaii (alias Kasaii-Virus) – Fundort: [[Arakawa]] (Fluss), [[Tokio]], Japan<ref name=Diesend2018 /><ref name=Takemura2016 /> |
|||
::* Samba-Virus<ref name=NCBIApMV /><ref name=Campos2014/> – Fundort: [[Rio Negro (Amazonas)|Rio Negro]], Brasilien<ref name=Diesend2018 /> |
|||
::* Amazonian virus (alias Amazonia virus)<ref name=Diesend2018 /><ref name=Assis2015 /> – Fundort: Roi Negro, Brasilien<ref name=Diesend2018 /> |
|||
::* Oyster virus – Fundort: [[Florianópolis]], Brasilien<ref name=Diesend2018 /><ref name=Assis2015 /> |
|||
::* Kroon-Virus<ref name=Boratto2018 /><ref name=Assis2015 /> – Fundort: Lagoa Santa<!--welches? – es gibt mehrere-->, Brasilien<ref name=Diesend2018 /> |
|||
:: Das NCBI führt unter APMV noch das Saudi moumouvirus (SDMV) an,<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1956188 Saudi moumouvirus]</ref> verweist aber selbst als Referenz die Arbeit von Bajrai ''et al.'' (2016), die im Titel "''Saudi Moumouvirus, the First Group B Mimivirus Isolated from Asia''"<ref name=Bajrai2016 /> korrekt auf Gruppe d. h. Linie B ([[Moumouvirus]]-Gruppe) verweist.<!-- |
|||
--> |
|||
:* Spezies: ''[[Acanthamoeba castellanii mamavirus]]'' (ACMV)<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=554168 Acanthamoeba castellanii mamavirus] (Species)</ref> – Fundort: Paris, Frankreich<ref name=Diesend2018 /><!-- |
|||
--> |
|||
:* Spezies: ''Hirudovirus''<ref name=NCBIHirudo>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1420594 Hirudovirue strain sangsue]</ref><ref name=Boughalmi2013 /> – Fundort: Tunesien<ref name=Diesend2018 /> |
|||
::* Hirudovirus sangsue<!-- |
|||
--> |
|||
:* Spezies: ''Niemeyer-Virus''<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1766765 Niemeyer virus] (Species)</ref><ref name=Boratto2015 /> – Fundort: [[Belo Horizonte]], Brasilien<ref name=Diesend2018 /><!-- |
|||
--> |
|||
:* Spezies: ''Mimivirus battle86''<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1642687 Mimivirus battle86] (Species)</ref><ref name=Cha2016 /><!-- |
|||
--> |
|||
:* Spezies: ''Mimivirus Cher''<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1189585 Mimivirus Cher] (Species)</ref><ref name=Cha2016 /><!-- |
|||
--> |
|||
:* Spezies: ''Mimivirus fauteuil'' (alias ''Fauteuil-Virus'')<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1128144 Mimivirus fauteuil]</ref><ref name=Desnues2012 /><ref name=Diesend2018 /> – vgl. ''Fauteuil virus FD''<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?mode=Undef&id=706155 Fauteuil virus FD]</ref><!-- |
|||
--> |
|||
:* Spezies: ''Mimivirus lactour''<!--sic!--> (alias ''Lactours-Virus'')<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1128145 Mimivirus lactour] (Species)</ref><ref name=Desnues2012 /> – vgl. ''Lactours virus LT2''<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=706156 Lactours virus LT2] (Species)</ref><!-- |
|||
--> |
|||
:* Spezies: ''Mimivirus lentille'' (alias ''Lentillevirus''?)<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1128146 Mimivirus lentille] (Species)</ref><ref name=Desnues2012 /> – vgl. Lentille virus CL<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=706157 Lentille virus CL] (Species)</ref><!-- |
|||
--> |
|||
:* Spezies: ''Mimivirus longchamps'' (alias ''Longchamps-Virus'')<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1128147 Mimivirus longchamps] (Species)</ref><ref name=Desnues2012 /><ref name=Diesend2018 /> – vgl. ''Longchamps virus FPL''<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=706158 Longchamps virus FPL] (Species)</ref><!-- |
|||
--> |
|||
:* Spezies: ''Mimivirus marais'' (alias ''Marais-Virus'')<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1128148 Mimivirus marais] (Species)]</ref><ref name=Desnues2012 /><!-- |
|||
--> |
|||
:* Spezies: ''Mimivirus montadette2''<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1686768 Mimivirus montadette2] (Species)</ref><ref name=Cha2016 /><!-- |
|||
--> |
|||
:* Spezies: ''Mimivirus pointerouge1''<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?&id=1128149 Mimivirus pointerouge1] (Species)</ref> – vgl. ''Pointerouge virus 1'' (alias ''Pointe-Rouge 1 virus'')<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=706161 Pointerouge virus 1] (Species)</ref><ref name=Diesend2018 /> – Fundort: [[Marseille]], Frankreich<ref name=Diesend2018 /> |
|||
<!-- |
|||
:* Spezies: ''Mimivirus pointerouge2''<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?&id=1128150 Mimivirus pointerouge1] (Species)</ref> – vgl. ''Pointerouge virus 2'' (alias ''Pointe-Rouge 2 virus'')<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=706164 Pointerouge virus 1] (Species)</ref><ref name=Diesend2018 /> – Fundort: Marseille, Frankreich<ref name=Diesend2018 /> |
|||
--> |
|||
:* Spezies: ''Mimivirus terra2'' (alias ''Terra2-Virus'')<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1128151 Mimivirus terra2] (Spezies)</ref><ref name=Desnues2012 /> – Fundort: Marseille, Frankreich<ref name=Diesend2018 /> |
|||
::* Terravirus2 TAO-TJA<ref name=Cha2016 /><!-- |
|||
--> |
|||
:* Spezies: ''Mimivirus univirus''<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1686769 Mimivirus univirus] (Species)</ref><ref name=Cha2016 /> |
|||
== Resistenzeigenschaft == |
|||
Das ''[[Zamilon-Virus]]'' ist ein [[Satellitenvirus]], das Mimiviren der Linien B und C befällt, nicht aber die Mimiviren der Linie A. Diese weisen nämlich eine MIMIVIRE, {{enS|mimivirus virophage resistance element}}), genannte [[Resistenz]] auf, die ähnlich funktioniert wie das [[CRISPR/Cas-System]].<ref name=MIMIVIRE>Anthony Levasseur, Meriem Bekliz, Eric Chabrière, Pierre Pontarotti, Bernard La Scola, Didier Raoult: ''MIMIVIRE is a defence system in mimivirus that confers resistance to virophage.'' In: ''[[Nature]]'', 2016, [[doi:10.1038/nature17146]].</ref><ref>Ewen Callaway: [http://www.nature.com/news/crispr-like-immune-system-discovered-in-giant-virus-1.19462 CRISPR-like 'immune' system discovered in giant virus], in: [[Nature]]:News, vom 29. Juni 2016</ref> |
|||
== Literatur == |
== Literatur == |
||
* Stefanie Reinberger: ''Revolution der Riesenviren.'' In: ''[[Spektrum der Wissenschaft]].'' [[Spektrum der Wissenschaft Verlagsgesellschaft]], Heidelberg, Mai 2012, S. |
* Stefanie Reinberger: ''[https://www.spektrum.de/magazin/revolution-der-riesenviren/1149102 Revolution der Riesenviren].'' In: ''[[Spektrum der Wissenschaft]].'' [[Spektrum der Wissenschaft Verlagsgesellschaft]], Heidelberg, Mai 2012, S. 14–16. |
||
== Weblinks == |
|||
<!--* [http://www.cnrs.fr/cw/en/pres/compress/mimivirus.htm Mimivirus: discovery of a giant virus]--> |
|||
* Jean-Michel Claverie (Hrsg.): [http://www.giantvirus.org/gallery.html Bilder des Mimivirus], von: Structural & Genomic Information Laboratory CNRS UPR, Marseille |
|||
* NCBI: [http://www.ncbi.nlm.nih.gov/entrez/viewer.fcgi??db=nucleotide&val=NC_006450 Referenzsequenz NC_006450] |
|||
* David R. Wessner: [https://www.nature.com/scitable/topicpage/discovery-of-the-giant-mimivirus-14402410/ Discovery of the Giant Mimivirus], auf: [[Nature]] Masterclasses |
|||
* Philippe Colson, Bernard La Scola, Anthony Levasseur, Gustavo Caetano-Anollés,& Didier Raoult: [https://www.nature.com/articles/nrmicro.2016.197 Mimivirus: leading the way in the discovery of giant viruses of amoebae], in: [[Nature]] Reviews Microbiology, Band 15, S. 243–254, 27. Februar 2017, [[doi:10.1038/nrmicro.2016.197]] |
|||
* [https://www.sciencedirect.com/topics/neuroscience/mimivirus Mimivirus – an overview], auf: ScienceDirect, 2012 |
|||
* Scinexx: [https://www.scinexx.de/news/biowissen/riesenviren-an-der-grenze-zum-leben/ Riesenviren an der Grenze zum Leben], vom 28. Februar 2018 |
|||
* Andreas Jahn: [https://www.spektrum.de/news/das-virus-virus/964070 Das Virus-Virus], auf: Spektrum.de vom 6. August 2008 |
|||
* Biologie Seite: [https://www.biologie-seite.de/Biologie/Mimivirus Mimivirus] |
|||
== Einzelnachweise == |
== Einzelnachweise == |
||
<references> |
|||
<ref name=Assis2015> |
|||
Felipe L. Assis, Leena Bajrai, Jonatas S. Abrahao, Erna G. Kroon, Fabio P. Dornas, Kétyllen R. Andrade, Paulo V. M. Boratto, Mariana R. Pilotto, Catherine Robert, Samia Benamar, Bernard La Scola, Philippe Colson: [https://www.mdpi.com/1999-4915/7/7/2782/htm Pan-Genome Analysis of Brazilian Lineage A Amoebal Mimiviruses], in: Viruses 7(7), 2015, S. :3483-3499, [[doi:10.3390/v7072782]]. Die hier abgehandelten Kandidaten stellen nach den Autoren eine Vewandtschaftsgruppe dar, wobei aus den anderen Quellen hervorgeht, dass Samba- und Kroon-Virus zur Spezies APMV gehören. Das gilt dann für die beiden anderen. |
|||
</ref> |
|||
<ref name=Bajrai2016> |
|||
Leena H. Bajrai, Felipe L. de Assis, Esam I. Azhar, Priscilla Jardot, Catherine Robert, Jônatas Abrahão, Didier Raoult, Bernard La Scola: [https://www.frontiersin.org/articles/10.3389/fmicb.2016.02029/full Saudi Moumouvirus, the First Group B Mimivirus Isolated from Asia], in: Front. Microbiol., 20. Dezember 2016, [[doi:10.3389/fmicb.2016.02029]] |
|||
</ref> |
|||
<ref name=Boratto2015> |
|||
Paulo V. M. Boratto, Thalita S. Arantes, Lorena C. F. Silva, Felipe L. Assis, Erna G. Kroon, Bernard La Scola, Jônatas S. Abrahão: [https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4639698/ Niemeyer Virus: A New Mimivirus Group A Isolate Harboring a Set of Duplicated Aminoacyl-tRNA Synthetase Genes], in: Front Microbiol. 2015; 6: 1256. [[doi:10.3389/fmicb.2015.01256]], {{PMC|4639698}}, PMID 26635738 |
|||
</ref> |
|||
<ref name=Boratto2018> |
|||
Paulo Victor Miranda Boratto, Fábio Pio Dornas, Lorena Christine Ferreira da Silva, Rodrigo Araújo Lima Rodrigues, Graziele Pereira Oliveira, Juliana Reis Cortines, Betânia Paiva Drumond, Jônatas Santos Abrahão: [https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5752926/ Analyses of the Kroon Virus Major Capsid Gene and Its Transcript Highlight a Distinct Pattern of Gene Evolution and Splicing among Mimiviruses], in: J Virol. 92(2), 15. Januar 2018, e01782-17, [[doi:10.1128/JVI.01782-17]], {{PMC|5752926}}, PMID 29118120 |
|||
</ref> |
|||
<ref name=Boughalmi2013> |
|||
Mondher Boughalmi, Isabelle Pagnier, Sarah Aherfi, Philippe Colson, Didier Raoult, Bernard La Scola: [https://www.mdpi.com/1999-4915/5/12/2920/pdf First Isolation of a Giant Virus from Wild ''Hirudo medicinalis'' Leech: ''Mimiviridae'' isolation in ''Hirudo medicinalis''], in: Viruses 2013, 5, S. 2920–2930, [[doi:10.3390/v5122920]] |
|||
</ref> |
|||
<ref name=Campos2014> |
|||
Rafael K Campos, Paulo V Boratto, Felipe L Assis, Eric RGR Aguiar, Lorena CF Silva, Jonas D Albarnaz, Fabio P Dornas, Giliane S Trindade, Paulo P Ferreira, João T Marques, Catherine Robert, Didier Raoult, Erna G Kroon, Bernard La Scola, Jônatas S Abrahão: [https://virologyj.biomedcentral.com/articles/10.1186/1743-422X-11-95 Samba virus: a novel mimivirus from a giant rain forest, the Brazilian Amazon], in: Virology Journal 2014 11:95, [[doi:10.1186/1743-422X-11-95]] |
|||
</ref> |
|||
<ref name=Cha2016> |
|||
Anirvan Chatterjee, Thomas Sicheritz-Pontén, Rajesh Yadav, Kiran Kondabagil: [https://www.researchgate.net/publication/303712856_Isolation_and_complete_genome_sequencing_of_Mimivirus_bombay_a_Giant_Virus_in_sewage_of_Mumbai_India Isolation and complete genome sequencing of Mimivirus bombay, a Giant Virus in sewage of Mumbai, India], in: Genomics Data 9(C), Mai 2016, [[doi:10.1016/j.gdata.2016.05.013]], Fig. 1 |
|||
</ref> |
|||
<ref name=Claverie2006> |
|||
{{cite journal |author=Jean-Michel Claverie, Hiroyuki Ogata, Stéphane Audic, Chantal Abergel, Pierre-Edouard Fournier, Karsten Suhre<!--last=Claverie |first=Jean-Michel |display-authors=etal--> |year=2006 |title=Mimivirus and the emerging concept of 'giant' virus |journal=Virus Research |volume=117 |issue=1 |pages=133–144 |doi=10.1016/j.virusres.2006.01.008 |url= |accessdate= |quote= |pmid=16469402 |arxiv=q-bio/0506007 }} |
|||
</ref> |
|||
<ref name=Claverie2009> |
|||
{{cite journal |author=Jean-Michel Claverie, Chantal Abergel, Hiroyuki Ogata |title=Mimivirus |url=https://link.springer.com/chapter/10.1007/978-3-540-68618-7_3 |journal=Curr Top Microbiol Immunol |year=2009 |volume=328 |pages=89—121 |pmid=19216436 }} |
|||
</ref> |
|||
<ref name=Claverie2> |
|||
{{cite journal |author=Jean-Michel Claverie, Chantal Abergel |title=Mimivirus and its virophage |journal=Annu Rev Genet |year=2009 |volume=43 |issue=49—66 |doi=10.1146/annurev-genet-102108-134255 |pmid=19653859 }} |
|||
</ref>. |
|||
<ref name=Deeg2018> |
|||
Christoph M. Deeg, Cheryl-Emiliane T. Chow, Curtis A. Suttle: [https://www.researchgate.net/publication/324041351_The_kinetoplastid-infecting_Bodo_saltans_virus_BsV_a_window_into_the_most_abundant_giant_viruses_in_the_sea_A_Flow_cytometry_profile_of_uninfected_Bodo_saltans_stained_with_Lysotracker_blue_arrow_head The kinetoplastid-infecting Bodo saltans virus (BsV), a window into the most abundant giant viruses in the sea…], in: eLife Sciences 7, März 2018, [[doi:10.7554/eLife.33014]] |
|||
</ref> |
|||
<ref name=Desnues2012> |
|||
Christelle Desnues, Bernard La Scola, Natalya Yutin, Ghislain Fournous ''et al.'': Provirophages and transpovirons as the diverse mobilome of giant viruses, in: PNAS 109(44), 30. Oktober 2012,S. 18078–18083, [[doi:10.1073/pnas.1208835109]] |
|||
</ref> |
|||
<ref name=Diesend2018> |
|||
Jan Diesend, Janis Kruse, Monica Hagedorn, Christian Hammann: [https://www.frontiersin.org/articles/10.3389/fcimb.2017.00527/full Amoebae, Giant Viruses, and Virophages Make Up a Complex, Multilayered Threesome], in: Frontiers in Cellular and Infection Microbiology 7, Januar 2018, [doi:10.3389/fcimb.2017.00527], [https://www.researchgate.net/publication/322397006_Amoebae_Giant_Viruses_and_Virophages_Make_Up_a_Complex_Multilayered_Threesome via ResearchGate], Fig. 1 (NCLDVs und ‚''Megavirales''‘ werden in dieser Arbeit nicht ganz korrekt als ‚Familien‘ bezeichnet, gemeint sind ‚Gruppen‘). |
|||
</ref> |
|||
<ref name=Klose2010> |
|||
{{cite journal |author=Klose T., Kuznetsov Y. G., Xiao C., Sun S., McPherson A., Rossmann M. G. |title=The three-dimensional structure of Mimivirus |url=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2895761/ |journal=Intervirology |year=2010 |volume=53 |issue=5 |pages=268—273 |doi=10.1159/000312911 |pmid=20551678 }} |
|||
</ref> |
|||
<ref name=LaScola2003> |
|||
Bernard La Scola, S. Audic, C. Robert, L. Jungang, X. de Lamballerie, M. Drancourt, R. Birtles, J. M. Claverie, D. Raoult: ''A giant virus in amoebae.'' In: ''Science.'' 299, 2003, S. 2033. PMID 12663918 |
|||
</ref> |
|||
<ref name=Mutsafi2010> |
|||
{{cite journal |author=Mutsafi Y., Zauberman N., Sabanay I., Minsky A. |title=Vaccinia-like cytoplasmic replication of the giant Mimivirus |url=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2851855/ |journal=Proc Natl Acad Sci USA (PNAS)|year=2010 |volume=107 |issue=13 |pages=5978—5982 |doi=10.1073/pnas.0912737107 |pmid=20231474 }} |
|||
</ref>. |
|||
<ref name=Ogata2005> |
|||
{{cite journal |author=Hiroyuki Ogata, Didier Raoult, Jean-Michel Claverie |title=A new example of viral intein in Mimivirus |url=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC549080/ |journal=Virol J |year=2005 |volume=2 |issue=8 |pmid=15707490 }} |
|||
</ref>. |
|||
<ref name=Pearson2008> |
|||
{{cite journal |author=H. Pearson |title='Virophage' suggests viruses are alive |journal=[[Nature]] |volume=454 |issue=7205 |year=2008 |pages=677–677 |issn=0028-0836 |doi=10.1038/454677a |bibcode=2008Natur.454..677P }} |
|||
</ref> |
|||
<ref name=Prescott1993> |
|||
{{cite book |title=Microbiology |last=Prescott |first=Lansing M. |year=1993 |publisher=Wm. C. Brown Publishers |location=Dubuque, IA |isbn=0-697-01372-3 |pages= |url= |editor=2. Auflage }} |
|||
</ref> |
|||
<ref name=Raoult2004> |
|||
Didier Raoult, S. Audic, C. Robert, C. Abergel, P. Renesto, H. Ogata, B. La Scola, M. Suzan, J. M. Claverie: ''The 1.2-Mb Genome Sequence of Mimivirus.'' In: ''Science.'' 306, 2004, S. 1344–1350. PMID 15486256 |
|||
</ref> |
|||
<ref name=Schulzetal2018> |
|||
Frederik Schulz, Lauren Alteio, Danielle Goudeau, Elizabeth M. Ryan, Feiqiao B. Yu, Rex R. Malmstrom, Jeffrey Blanchard, Tanja Woyke: [https://www.nature.com/articles/s41467-018-07335-2#ref-CR38 Hidden diversity of soil giant viruses], in: Nature Communicationsvolume 9, Article number: 4881 (2018) vom 19. November 2018, [[doi:10.1038/s41467-018-07335-2]] |
|||
</ref> |
|||
<ref name=SuzanMonti2006> |
|||
{{cite journal |author=Suzan-Monti M, La Scola B, Raoult D |title=Genomic and evolutionary aspects of Mimivirus |journal=Virus Research |volume=117 |issue=1 |pages=145–155 |date=April 2006 |pmid=16181700 |doi=10.1016/j.virusres.2005.07.011 }} |
|||
</ref> |
|||
<ref name=Takemura2016> |
|||
Masaharu Takemura, Tatsuya Mikami, Shingo Murono: [https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5146454/ Nearly Complete Genome Sequences of Two ''Mimivirus'' Strains Isolated from a Japanese Freshwater Pond and River Mouth], in: Genome Announc. 4(6), November/Dezember 2016, e01378-16, [[doi:10.1128/genomeA.01378-16]], {{PMC|5146454}}, PMID 27932662. Die beiden Viruslinien sind nac Angaben der Autoren eng mit Mimivirus Bombay verwandt, gehören also ebenfalls zur Spezies APMV. |
|||
</ref> |
|||
<ref name=Vincent2010> |
|||
{{cite journal |author=Vincent A., La Scola B., Papazian L. |title=Advances in Mimivirus pathogenicity |journal=Intervirology |year=2010 |volume=53 |issue=5 |pages=304—309 |doi=10.1159/000312915 |pmid=20551682 }} |
|||
</ref>. |
|||
<ref name=Xiao2009> |
|||
{{cite journal |author=Xiao C., Kuznetsov Y. G., Sun S., Hafenstein S. L., Kostyuchenko V. A., Chipman P. R., Suzan-Monti M., Raoult D., McPherson A., Rossmann M. G. |title=Structural studies of the giant mimivirus |url=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2671561/ |journal=PLoS Biol |year=2009 |volume=7 |issue=4 |pages=e92 |doi=10.1371/journal.pbio.1000092 |pmid=19402750 }} |
|||
</ref> |
|||
<ref name="YWK2014_Megavirales"> |
|||
Natalya Yutin, Y. I. Wolf, E. V. Koonin: ''Origin of giant viruses from smaller DNA viruses not from a fourth domain of cellular life.'' In: ''Virology.'' 2014: „{{lang|en|The ‘Megavirales’ unite 7 families of viruses …}}“. |
|||
</ref> |
|||
<ref name=Zauberman2008> |
|||
{{cite journal |author=Zauberman N., Mutsafi Y., Halevy D. B., Shimoni E., Klein E., Xiao C., Sun S., Minsky A. |title=Distinct DNA exit and packaging portals in the virus Acanthamoeba polyphaga mimivirus |url=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2430901/ |journal=PLoS Biol |year=2008 |volume=6 |issue=5 |pages=e114 |doi=10.1371/journal.pbio.0060114 |pmid=18479185 }}</ref>]] |
|||
</references> |
|||
<references /> |
<references /> |
||
== Weblinks == |
|||
* [http://www.cnrs.fr/cw/en/pres/compress/mimivirus.htm Mimivirus: discovery of a giant virus] |
|||
* [http://www.giantvirus.org/gallery.html Bilder des Mimivirus] |
|||
* [http://www.ncbi.nlm.nih.gov/entrez/viewer.fcgi??db=nucleotide&val=NC_006450 Referenzsequenz NC_006450] |
|||
[[Kategorie:Viren, Viroide und Prionen]] |
[[Kategorie:Viren, Viroide und Prionen]] |
||
Version vom 11. August 2019, 10:28 Uhr
| Mimivirus | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|

EM-Aufnahme eines Virions (Virusteilchens) des | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Taxonomische Merkmale | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Acanthamoeba polyphaga mimivirus | ||||||||||||
| Kurzbezeichnung | ||||||||||||
| APMV | ||||||||||||
| Links | ||||||||||||
|
Mimivirus ist eine Gattung von Viren aus der Familie der Mimiviridae,[4] denen Amöben als natürliche Wirte dienen. Mit der Familie Mimiviridae gehört Mimivirus zu den Riesenviren in der Gruppe der Nucleocytoplasmic large DNA viruses (NCLDV), für die vorgeschlagen wurde, sie als Megavirales in den Rang einer Virusordnung zu erheben.[5]
In der Gattung Mimivirus gibt es mit Stand März 2019 eine einzige vom International Committee on Taxonomy of Viruses (ICTV) bestätigte Art Acanthamoeba polyphaga mimivirus (APMV), die daher auch die Typusart ist.[6] In der Umgangssprache und in älterer Literatur wird APMV üblicherweise nur als Mimivirus bezeichnet. Es gibt jedoch eine ganze Reihe phylogenetisch verwandter großer Viren, die als weitere Mitglieder dieser Gattung vorgeschlagen sind.
Entdeckung und Forschungsgeschichte
Das Mimivirus APMV wurde 1992 bei Forschungsarbeiten über die Legionärskrankheit (Legionellose) in einem Industriekühlturm in Bradford (England) entdeckt, wobei man feststellte, dass es sich in der Amöbe Acanthamoeba polyphaga vermehrt. Im Jahr 2003 wurde es an der Université de la Méditerranée in Marseille von einer Arbeitsgruppe um Didier Raoult identifiziert.[7] Mit einem Durchmesser von 400 nm haben die Viruspartikel (Virionen) von APMV die Größe von kleinen Bakterien. Wegen dieser Größe und der äußeren Ähnlichkeit mit kugelförmigen Bakterien (Kokken) hielt man es zunächst für ein grampositives Bakterium und nannte es Bradfordcoccus. Als man den Irrtum erkannte, benannte man das neu entdeckte Virus in Anspielung auf seine Größe und Färbungseigenschaften Mimicking Virus, täuschendes Virus. Schließlich wurde daraus kurz Mimivirus, mit dem Namensteil mimi als eine Abkürzung für englisch mimicking microbe. Im Oktober 2004 wurde dann von Didier Raoult und Kollegen die Struktur seines Erbguts in der Fachzeitschrift Science veröffentlicht.[8]
Das gleiche Team, das das APMV entdeckte, entdeckte später ein etwas größeres Virus, das Acanthamoeba castellanii mamavirus (ACMV, kurz Mamavirus) zusammen mit dem Sputnik-Virophagen, der es infiziert. ACMV und APMV sind so nahe verwandt, dass sie gewöhnlich in dieselbe Gattung Mimivirus gestellt werden.[9]
Bis 2013, als ein noch größeres Virus, das Pandoravirus, beschrieben wurde, wiesen die Viren der Gattung Mimivirus den größten Kapsiddurchmesser aller bekannten Viren auf; inzwischen wird es aber auch vom nahe Verwandten Megavirus chilensis und vom Pithovirus übertroffen.
Aufbau


B: AFM-Aufnahme von zwei abgelösten Oberflächenfasern des Mimivirus.
C: Cryo-EM-Bild eines Mimivirus nach teilweisem Abbau der Fibrillen mittels Bromelain.
D: AFM-Bild von inneren Fasern des Mimivirus.
Die Kapside der Virionen (Viruspartikel) von APMV erscheinen unter einem Elektronenmikroskop sechseckig, daher ist die Kapsidgeometrie ikosaedrisch.[10] Es scheint keine äußere virale Hülle zu geben, was darauf hindeutet, dass das Virus die Wirtszelle nicht durch Exozytose verlässt.[11]
Das Hauptprotein des Mimivirus-Kapsids besteht aus zwei Domänen vom Biskuitrollen-Typ (englisch Jelly roll fold). Dieses Protein bildet homotrimere Kapsomere als Organisationseinheit der Kapside. Die Kapsomere sind hexagonal in Form von „Gänseblümchen“ gepackt: Sechs Kapsomere umgeben eine Vertiefung zwischen ihnen.[10][12]
Die Virionen von Mimivirus habe einen Kapsiddurchmesser von 400 nm.
Fibrillen
Das Kapsid ist mit einer kompakten Schicht von Fibrillen bedeckt. Die aus der Oberfläche des Kapsids herausragenden Proteinfilamente haben eine Länge von etwa 100 nm (80-120 nm), und bringen damit die Gesamtlänge eines Virions auf 600 nm.[3] Abweichungen in der wissenschaftlichen Literatur lassen die Zahlen als sehr ungenau erscheinen, wenn zum Beispiel die ‚Größe’ des Virions gelegentlich als irgendwo zwischen 400 und 800 nm angegeben wird. Abgesehen von Unterschieden zwischen den einzelnen Virusstämmen in der Gattung Mimivirus ist manchmal die Gesamtgröße mit Filamenten, und manchmal der reine Kapsiddurchmesser angegeben.
Untersuchungen dieser Filamente unter Verwendung eines Rasterkraftmikroskops haben ergeben, dass diese häufig an einer gemeinsamen Tragstruktur befestigt sind und mit einer kleinen Kugel enden. Es ist jedoch immer noch nicht bekannt, an welchen Teilen der Kapsidoberfläche sie befestigt sind.[10] Die Proteinfilamente sind resistent gegen Proteasen, außer sie werden mit Lysozym behandelt. Daher scheinen die Filamente mit Peptidoglycan beschichtet zu sein. Dies steht in guter Übereinstimmung mit der Tatsache, dass das Mimivirus durch die Gram-Methode gefärbt wird. Es wird angenommen, dass die stark glykosylierte Oberfläche der Filamente eine Rolle bei der Annäherung an Wirtsamöben spielt.>[13]
Stargate
Auffällig ist die fünfeckige, sternförmige Struktur an einer der Ecken des Kapsids, das so genannte ‚Stargate‘ (englisch für „Sternentor“). Blickt man direckt auf diesen Eckpunkt (den Mittelpunkt des Sterns) so scheinen zwischen dessen Strahlen fünf dreieckige Flächen zu liegen. Die Strahlen haben eine Breite von ungefähr 50 nm, eine Dicke von 40 nm und eine Länge von 200 nm; sie erreichen fast die benachbarten Eckpunkte der ikosaedrischen Kapsids. Das Vorhandensein dieser Struktur verändert die Geometrie des Kapsids, indem seine Geometrie von der idealen Ikosaederform abweicht: Tatsächlich verläuft bei genauer Betrachtung nur eine einzige Achse mit Fünfstrahlsymmetrie durch das Virion, die durch den Mittelpunkt des Sterns (genannt Scheitelpunkt) verläuft.[10] Die Symmetrie des Kapsids wird unterschiedlich angegeben mit T=972–1141 oder T=1200.[4]
Da auf der Oberfläche der Sternstruktur keine hexagonal geordneten Vertiefungen zu beobachten sind, wird vermutet, dass es sich bei dieser um ein Protein handelt, das sich vom Hauptkapsidprotein unterscheidet.[10]
Das Stargate spielt eine besondere Rolle bei der Infektion der Wirtszelle: Während der Infektion öffnet sich der „Verschluss“ am Scheitelpunkt und es erfolgt die Freisetzung des viralen Kerns (mit DNA und vorgefertigten Proteinen) aus dem Kapsid in das Zytosol der Wirtszelle (per Phagocytose). Das ist der Grund, warum die Sternstruktur als „Sternentor“ (englisch stagate) bezeichnet wird.[14]
Nukleokapsid
Das Mimivirus hat mehrere morphologische Merkmale mit allen Mitgliedern der NCLDV-Virengruppe gemeinsam. Unmittelbar unter dem Kapsid des Mimivirus befinden sich beispielsweise zwei elektronendichte Schichten, die als Membranen gedeutet werden.[13] Unter diesen Membranen befindet sich eine etwa 7 nm dicke Proteinhülle, in der die lineare doppelsträngige DNA des Virus eingeschlossen ist. Dieser kondensierte Zentralkern des Virions erscheint unter dem Elektronenmikroskop als dunkler Bereich, das sogenannte ‚Nukleokapsid‘. In diesem Bereich befindet sich das große Genom des Virus, daneben auch mRNAs und vorgefertigte Proteine. Da alle anderen NCLDVs eine interne Lipidschicht besitzen, die den zentralen Kern umgibt, vermutet man das auch bei Mimivirus. Die Wände des Nukleokapsids liegen etwa 30 nm hinter den Wänden des Kapsids zurück, im Bereich der Sternstruktur (dem Stargate) ist die Oberfläche des Nukleokapsids zusätzlich abgesenkt.[10] Es wird angenommen, dass der Raum zwischen der Spitze der Sternstruktur und dem Nukleokapsid mit hydrolytischen Enzymen gefüllt ist, die für das Eindringen des Virus in die Wirtszelle erforderlich sind. Zwischen dem Kapsid und dem Nukleokapsid wurden interne Proteinstränge entdeckt, die anscheinend die gegenseitige räumliche Positionierung der beiden Teile zueinander stabilisieren.[13]
Genom
APMV hat damit im Vergleich zu den meisten anderen Viren ein großes und komplexes Genom, das eis einem einzelnen linearen DNA-Doppelstrang (dsDNA) besteht. Die DNA von APMV ist 800 nm lang, verfügt über 1.181.404 Basenpaare und 1260 Offene Leserahmen (ORFs, englisch open reading frames), darunter geschätzt 911–979 Gene, die Proteine kodieren. Dies geht weit über die Mindestausstattung von 4 Genen hinaus, die für ein Virus erforderlich sind, wie etwa bei den Phagen MS2 und Qβ.[15] Detaillierte Studien zum Genom laufen noch: Sequenzfehler werden korrigiert und neue Leserahmen entdeckt.[16]
Der Anteil von nichtcodierender DNA beträgt nur etwa 9,5 bis 10 %. Offene Leserahmen sind durch Lücken von ungefähr 157 Nukleotidpaaren getrennt. Zwei DNA-Abschnitte mit der Bezeichnung R (englisch right - rechts) und L (englisch left - links) kodieren ungefähr die gleiche Anzahl von Genen (450 bzw. 465, gemäß Daten von 2010). Der GC-Gehalt ist mit 28 % niedrig. In der Nähe der Enden des DNA-Moleküls wurden ‚Invertierte Wiederholungen‘ (englisch inverted repeats) mit 617 Nukleotidpaaren gefunden. Es wird vermutet, dass die gegenseitige Wechselwirkung dieser Stellen zur Bildung einer Q-Struktur führen kann - zirkuläre DNA mit zwei kleinen Fortsätzen.[16]
Bei der Analyse stieß man auf mindestens 21 Gene mit Homologie zu bekannten Proteinen, darunter solche, die man bis dato von keinem anderen Virus, sondern nur von zellulären Organismen kannte, inklusive Aminoacyl-tRNA-Synthetase.[8][7][17] Wie andere Riesenviren (NCLDVs) enthält das Mimivirus mehrere Gene für den Zucker-, Lipid- und Aminosäurestoffwechsel. Es gab auch Stoffwechselgene, die zuvor in keinem anderen Virus gefunden wurden.[11]
Aus gereinigten Virionen konnten mehrere mRNA-Transkripte gewonnen werden. Wie schon bei anderen NCLDVs wurden insbesondere Transkripte für DNA-Polymerase, ein Kapsidprotein und ein TFII-ähnlicher Transkriptionsfaktor gefunden werden. Es wurden jedoch auch drei verschiedene Aminoacyl-tRNA-Synthetase-Transkripte und vier unbekannte mRNA-Moleküle gefunden, die für das Mimivirus spezifisch sind. Diese vorverpackten Transkripte können ohne virale Genexpression translatiert werden und sind wahrscheinlich für die Replikation von Mimivirus erforderlich. Andere DNA-Viren, wie das Humane Cytomegalievirus und das Herpes-simplex-Virus Typ 1, enthalten ebenfalls gepackte mRNA-Transkripte.[11]
Mimivirus ist eines der wenigen dsDNA-Viren, in deren Genom eine Intein-kodierende Sequenz nachgewiesen wurde. Inteine sind Proteindomäne, die ihre eigene Entfernung von einem Trägermolekül und die anschließende Verknüpfung der gebildeten Enden katalysieren. Eine solche Sequenz ist im Mimivirus-Gen für DNA-Polymerase B vorhanden.[18]
Auf Grund der außergewöhnlich komplexen genetischen Ausstattung des Virus stellt sich für einige Forscher die Frage neu, wo die Grenze zwischen belebter und unbelebter Natur verlaufe, also wie „Lebewesen“ zu definieren ist.
Vermehrungszyklus
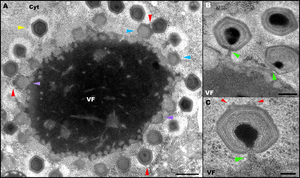
A) TEM-Aufnahme einer intrazellulären Virusfabrik, mit Viruspartikeln von Mimivirus in verschiedenen Montagestadien
- VF: Virusfabrik (Viroplasma)
- Cyt: Cytoplasma
- violette Pfeile: leere, noch faserlose Virionen im Anfangsstadium der Zusammensetzung, in unmittelbarer Nähe zur Peripherie der Virusfabrik auftretend
- blaue Pfeile: Teilweise montierte leere, faserlose Virionen
- gelbe Pfeile: reife, faserbedeckte Virionen, die sich jetzt weiter von der Virusfabrik entfernt befinden als die unreifen Partikel
- rote Pfeile: bei mehreren Viruspartikeln ist ein ‚Stargate’, das sich regelmäßig an der distalen Stelle der Fabrik befindet, zu erkennen
B) und C) TEM-Aufnahme der Mimivirus-Partikel
- grüne Pfeile: Mimivirus-Partikel im Stadium des ‚DNA-Packaging’ (DNA-Verpackung)
- rote Pfeile: zwei Kanten eines Stargates gegenüber der Stelle der DNA-Verpackung
Die Einzelheiten und die verschiedenen Stadien im Replikationszyklus von 'Mimivirus, die offensichtlichste Bindung an die Zelloberfläche und den Eintritt, die Freisetzung des Viruskerns, die DNA-Replikation, die Transkription, die Translation, und schließlich den Zusammenbau und die Freisetzung von Tochter-Virionen, sind noch nicht ausreichend bekannt. Die Wissenschaftler haben jedoch den oben angegebenen allgemeinen Überblick anhand elektronenmikroskopischer Aufnahmen infizierter Zellen erstellt. Alle Stadien des Vermehrungszyklus verlaufen im Cytoplasma der Wirtszelle.[19]
Die Infektion der Amöbe mit einem Mimivirus erfolgt vermutlich nach folgendem Szenario:
- Die Mimivirus-Virionen ähneln in ihrer Größe und dem Vorhandensein charakteristischer Polysaccharide auf der Oberfläche Bakterien (siehe Gram-Färbung, Name). Sie werden daher von der Amöbe als Nahrung während eines Endozytoseprozesses absorbiert. Die Polysaccharide fungieren dabei als chemischer Rezeptor und leiten die Anlagerung ein. Als Ergebnis der Endozytose befinden sich die Virionen in Endosomen innerhalb der Zelle.
- Die Proteinfilamente werden in den Endosomen teilweise lysiert, wodurch das Kapsid mit der Endosomenmembran in Wechselwirkung treten kann.
- ~2 Stunden nach der Infektion: Das Kapsid öffnet sich im Bereich der Sternstruktur (Stargate), die innere Membran fusioniert mit der Endosomenmembran und der Inhalt des Kapsids wird in das Cytoplasma freigesetzt.
- Nachdem das Kernteilchen (der innere Teil des Nukleokapsids) ins Cytoplasma ausgetreten ist, beginnt aufgrund der Anwesenheit des viralen Transkriptionsapparats die Synthese der viralen mRNA. Diese mRNAs reichern sich im Inneren des Kernpartikels in Form von Granula an.[19] Äußerlich betrachtet scheint das Virus verschwunden und alles in der Zelle sieht normal aus (Dunkelphase, (englisch eclipse phase).
- 4-5 Stunden nach der Infektion: Die virale DNA verlässt das Kernteilchen und wird entpackt, so dass die Replikation beginnen kann. Infolgedessen entsteht neben der leeren Hülle des Kernpartikels eine sogenannte „Virusfabrik“ - ein Ort für die Synthese der einzelnen Komponenten der Virionen und ihren anschließenden Zusammenbau. Wenn mehrere Viruspartikel in die Zelle gelangt sind, verschmelzen die von ihnen gebildeten „Fabriken“ beim Wachstum zu einer einzigen. Man erkennt jetzt kleine Ansammlungen in einigen Bereichen der Zelle.
- 6–9 Stunden nach der Infektion: Zusammensetzung (Assemblierung) der Kapside mit gleichzeitiger DNA-Packung an der Peripherie der „Virusfabriken“. Eine ungewöhnliche Eigenschaft des Mimivirus ist, dass DNA gepackt ist.[14] Die Mimivirus-Virionen werden in der Zelle deutlich sichtbar.
- 14-24 Stunden nach der Infektion: Die Amöbenzellen werden lysiert, d. h. sie platzen auf und die Virionen werden freigesetzt. Pro Wirtszelle werden so mehr als 300 Einheiten erzeugt.[16][11]
Die Übertragung geschieht durch passive Diffusion[4]
Wirte und Pathogenität
Der erste bekannte Mimivirus-Wirt ist die Amöbe Acanthamoeba polyphaga (Amoebozoa). Es hat sich gezeigt, dass nur andere Vertreter der Gattung Acanthamoeba, A. castellanii und A. mauritaniensis , als Wirte dieses Virus dienen können, keine Zellen anderer einzelliger oder mehrzelliger Organismen.[11]
Es wurde spekuliert, dass Mimivirus Erreger bestimmter Formen von Lungenentzündung (Pneumonia) sein könnte. Dies beruht hauptsächlich auf indirekten Nachweisen in Form von Antikörpern gegen das bei Lungenentzündungspatienten entdeckte Virus.[20] Aufgrund der wenigen bisherigen Veröffentlichung ist die Einstufung des Mimivirus als möglicher Krankheitserreger ist derzeit jedoch schwierig. Ein großer Teil der Fälle von Lungenentzündug verläuft ohne feststellbare Ursache.[21] Zwar wurde ein Mimivirus bei iner an Lungenentzündung leidenden Tunesierin isoliert,[22] und es gibt Hinweise darauf, dass Mimivirus Makrophagen infizieren kann. Unter experimentellen Bedingungen wurde beobachtet, dass Mimivirus humane Makrophagen infizieren kann, d. h. via Phagocytose in die Zellen eindringen, und sich dort replizieren kann.[23][24] Außerdem wurden in mehreren Studien bei einer kleinen Anzahl von Patienten mit Lungenentzündung Antikörper gegen das Mimivirus gefunden.[20][25] Es wurde auch ein Einzelfall einer Lungenentzündung in einem Laborassistenten beschrieben, der mit Kulturen dieses Virus arbeitete. Der Gehalt an Antikörpern gegen Mimivirus in seinem Blut war ebenfalls erhöht.[26] Das Vorhandensein von Antikörpern gegen das Virus an sich ist jedoch kein Hinweis auf seine Pathogenität. Es ist möglich, dass das Mimivirus einfach starke immunogene Eigenschaften aufweist, d. h. eine deutliche Immunantwort auslöst.[16]. Auch war es in keinem der registrierten Fälle möglich, das Virus in seiner reinen Form aus Proben von Flüssigkeiten zu isolieren, die von Patienten erhalten wurden.[27]
Systematik
Äußere Systematik
Die Gattung Mimivirus und einige andere – vom ICTV mit Stand März 2019 noch nicht bestätigte – genetisch ähnliche Gattungen und Spezies der Familie Mimiviridae (wie zum Beispiel Mamavirus, Megavirus und Moumouvirus) bilden eine als Gruppe I bezeichnete Klade der Mimiviren im weiteren Sinn. Für diese wurde vorgeschlagen, sie als Megamimivirinae[2] oder auch Mimivirinae[1] in den Rang einer Unterfamilie zu erheben und so von anderen ebenfalls vorgeschlagenen Unterfamilien der Mimiviridae abzugrenzen. Solche wären:
- Cafeteriavirus-Gruppe (Gruppe II)
- Klosneuvirus-Gruppe – als mögliche Unterfamilie ‚Klosneuvirinae‘
- OLPG-Gruppe (Gruppe III) – als Unterfamilie ‚Mesomimivirinae‘
Innerhalb dieser putativen Unterfamilie zeichnen sich die folgenden Linien ab:
- Linie A: Mimivirus-Gruppe: Mimiviren im engeren Sinn
- Linie B: Moumouvirus-Gruppe (Moumouviren)
- Linie C: Courdo11-Gruppe mit Megavirus chilensis (Spezies), dazu Courdo11-Virus
- Tupanvirus-Gruppe (Tupanviren)
Da bisher (März 2019) vom ICTV lediglich die Gattung Mimivirus bestätigt wurde, bleibt zur Zeit noch offen, welche Kandidaten für diese putative Unterfamilie dieser Gattung einmal zugeordnet werden, und für welche eigene Gattungen einzurichten sind. Sicher scheint aber, dass die Vertreter der Linie A in jedem Fall zur Gattung Mimivirus gehören.
Innere Systematik
Systematik der Linie A (Mimiviren im engeren Sinn):
- Gattung Mimivirus
- Spezies: Acanthamoeba polyphaga mimivirus (APMV)[28] (vom ICTV mit Stand März 2018 bestätigt) – Fundort: Bradford, England, UK[3]
- Mimivirus Bombay (alias Bombay-Virus)[28][3] – Fundort: Mumbai, Indien
- Mimivirus shirakomae (alias Shirakomae-Virus) – Fundort: Shirakoma Pond, Nagano, Japan[3][29]
- Mimivirus kasaii (alias Kasaii-Virus) – Fundort: Arakawa (Fluss), Tokio, Japan[3][29]
- Samba-Virus[28][30] – Fundort: Rio Negro, Brasilien[3]
- Amazonian virus (alias Amazonia virus)[3][31] – Fundort: Roi Negro, Brasilien[3]
- Oyster virus – Fundort: Florianópolis, Brasilien[3][31]
- Kroon-Virus[32][31] – Fundort: Lagoa Santa, Brasilien[3]
- Das NCBI führt unter APMV noch das Saudi moumouvirus (SDMV) an,[33] verweist aber selbst als Referenz die Arbeit von Bajrai et al. (2016), die im Titel "Saudi Moumouvirus, the First Group B Mimivirus Isolated from Asia"[34] korrekt auf Gruppe d. h. Linie B (Moumouvirus-Gruppe) verweist.
- Spezies: Acanthamoeba castellanii mamavirus (ACMV)[35] – Fundort: Paris, Frankreich[3]
- Spezies: Hirudovirus[36][37] – Fundort: Tunesien[3]
- Hirudovirus sangsue
- Spezies: Niemeyer-Virus[38][39] – Fundort: Belo Horizonte, Brasilien[3]
- Spezies: Mimivirus battle86[40][41]
- Spezies: Mimivirus Cher[42][41]
- Spezies: Mimivirus fauteuil (alias Fauteuil-Virus)[43][44][3] – vgl. Fauteuil virus FD[45]
- Spezies: Mimivirus lactour (alias Lactours-Virus)[46][44] – vgl. Lactours virus LT2[47]
- Spezies: Mimivirus lentille (alias Lentillevirus?)[48][44] – vgl. Lentille virus CL[49]
- Spezies: Mimivirus longchamps (alias Longchamps-Virus)[50][44][3] – vgl. Longchamps virus FPL[51]
- Spezies: Mimivirus marais (alias Marais-Virus)[52][44]
- Spezies: Mimivirus montadette2[53][41]
- Spezies: Mimivirus pointerouge1[54] – vgl. Pointerouge virus 1 (alias Pointe-Rouge 1 virus)[55][3] – Fundort: Marseille, Frankreich[3]
- Spezies: Mimivirus terra2 (alias Terra2-Virus)[56][44] – Fundort: Marseille, Frankreich[3]
- Terravirus2 TAO-TJA[41]
Resistenzeigenschaft
Das Zamilon-Virus ist ein Satellitenvirus, das Mimiviren der Linien B und C befällt, nicht aber die Mimiviren der Linie A. Diese weisen nämlich eine MIMIVIRE, englisch mimivirus virophage resistance element), genannte Resistenz auf, die ähnlich funktioniert wie das CRISPR/Cas-System.[58][59]
Literatur
- Stefanie Reinberger: Revolution der Riesenviren. In: Spektrum der Wissenschaft. Spektrum der Wissenschaft Verlagsgesellschaft, Heidelberg, Mai 2012, S. 14–16.
Weblinks
- Jean-Michel Claverie (Hrsg.): Bilder des Mimivirus, von: Structural & Genomic Information Laboratory CNRS UPR, Marseille
- NCBI: Referenzsequenz NC_006450
- David R. Wessner: Discovery of the Giant Mimivirus, auf: Nature Masterclasses
- Philippe Colson, Bernard La Scola, Anthony Levasseur, Gustavo Caetano-Anollés,& Didier Raoult: Mimivirus: leading the way in the discovery of giant viruses of amoebae, in: Nature Reviews Microbiology, Band 15, S. 243–254, 27. Februar 2017, doi:10.1038/nrmicro.2016.197
- Mimivirus – an overview, auf: ScienceDirect, 2012
- Scinexx: Riesenviren an der Grenze zum Leben, vom 28. Februar 2018
- Andreas Jahn: Das Virus-Virus, auf: Spektrum.de vom 6. August 2008
- Biologie Seite: Mimivirus
Einzelnachweise
- ↑ a b Christoph M. Deeg, Cheryl-Emiliane T. Chow, Curtis A. Suttle: The kinetoplastid-infecting Bodo saltans virus (BsV), a window into the most abundant giant viruses in the sea…, in: eLife Sciences 7, März 2018, doi:10.7554/eLife.33014
- ↑ a b Frederik Schulz, Lauren Alteio, Danielle Goudeau, Elizabeth M. Ryan, Feiqiao B. Yu, Rex R. Malmstrom, Jeffrey Blanchard, Tanja Woyke: Hidden diversity of soil giant viruses, in: Nature Communicationsvolume 9, Article number: 4881 (2018) vom 19. November 2018, doi:10.1038/s41467-018-07335-2
- ↑ a b c d e f g h i j k l m n o p q r s Jan Diesend, Janis Kruse, Monica Hagedorn, Christian Hammann: Amoebae, Giant Viruses, and Virophages Make Up a Complex, Multilayered Threesome, in: Frontiers in Cellular and Infection Microbiology 7, Januar 2018, [doi:10.3389/fcimb.2017.00527], via ResearchGate, Fig. 1 (NCLDVs und ‚Megavirales‘ werden in dieser Arbeit nicht ganz korrekt als ‚Familien‘ bezeichnet, gemeint sind ‚Gruppen‘).
- ↑ a b c ViralZone: Mimivirus. ExPASy, abgerufen am 8. Juli 2019.
- ↑ ICTV: Master Species List 2018b.v2. Abgerufen am 6. August 2019. MSL #34v
- ↑ a b Bernard La Scola, S. Audic, C. Robert, L. Jungang, X. de Lamballerie, M. Drancourt, R. Birtles, J. M. Claverie, D. Raoult: A giant virus in amoebae. In: Science. 299, 2003, S. 2033. PMID 12663918
- ↑ a b Didier Raoult, S. Audic, C. Robert, C. Abergel, P. Renesto, H. Ogata, B. La Scola, M. Suzan, J. M. Claverie: The 1.2-Mb Genome Sequence of Mimivirus. In: Science. 306, 2004, S. 1344–1350. PMID 15486256
- ↑ H. Pearson: 'Virophage' suggests viruses are alive. In: Nature. 454. Jahrgang, Nr. 7205, 2008, ISSN 0028-0836, S. 677–677, doi:10.1038/454677a, bibcode:2008Natur.454..677P.
- ↑ a b c d e f g Xiao C., Kuznetsov Y. G., Sun S., Hafenstein S. L., Kostyuchenko V. A., Chipman P. R., Suzan-Monti M., Raoult D., McPherson A., Rossmann M. G.: Structural studies of the giant mimivirus. In: PLoS Biol. 7. Jahrgang, Nr. 4, 2009, S. e92, doi:10.1371/journal.pbio.1000092, PMID 19402750 (nih.gov).
- ↑ a b c d e Suzan-Monti M, La Scola B, Raoult D: Genomic and evolutionary aspects of Mimivirus. In: Virus Research. 117. Jahrgang, Nr. 1, April 2006, S. 145–155, doi:10.1016/j.virusres.2005.07.011, PMID 16181700.
- ↑ Jean-Michel Claverie, Chantal Abergel, Hiroyuki Ogata: Mimivirus. In: Curr Top Microbiol Immunol. 328. Jahrgang, 2009, S. 89—121, PMID 19216436 (springer.com).
- ↑ a b c Klose T., Kuznetsov Y. G., Xiao C., Sun S., McPherson A., Rossmann M. G.: The three-dimensional structure of Mimivirus. In: Intervirology. 53. Jahrgang, Nr. 5, 2010, S. 268—273, doi:10.1159/000312911, PMID 20551678 (nih.gov).
- ↑ a b c Zauberman N., Mutsafi Y., Halevy D. B., Shimoni E., Klein E., Xiao C., Sun S., Minsky A.: Distinct DNA exit and packaging portals in the virus Acanthamoeba polyphaga mimivirus. In: PLoS Biol. 6. Jahrgang, Nr. 5, 2008, S. e114, doi:10.1371/journal.pbio.0060114, PMID 18479185 (nih.gov).
- ↑ Lansing M. Prescott: Microbiology. Hrsg.: 2. Auflage. Wm. C. Brown Publishers, Dubuque, IA 1993, ISBN 0-697-01372-3.
- ↑ a b c d Jean-Michel Claverie, Chantal Abergel: Mimivirus and its virophage. In: Annu Rev Genet. 43. Jahrgang, Nr. 49—66, 2009, doi:10.1146/annurev-genet-102108-134255, PMID 19653859.
- ↑ Jean-Michel Claverie, Hiroyuki Ogata, Stéphane Audic, Chantal Abergel, Pierre-Edouard Fournier, Karsten Suhre: Mimivirus and the emerging concept of 'giant' virus. In: Virus Research. 117. Jahrgang, Nr. 1, 2006, S. 133–144, doi:10.1016/j.virusres.2006.01.008, PMID 16469402, arxiv:q-bio/0506007.
- ↑ Hiroyuki Ogata, Didier Raoult, Jean-Michel Claverie: A new example of viral intein in Mimivirus. In: Virol J. 2. Jahrgang, Nr. 8, 2005, PMID 15707490 (nih.gov).
- ↑ a b Mutsafi Y., Zauberman N., Sabanay I., Minsky A.: Vaccinia-like cytoplasmic replication of the giant Mimivirus. In: Proc Natl Acad Sci USA (PNAS). 107. Jahrgang, Nr. 13, 2010, S. 5978—5982, doi:10.1073/pnas.0912737107, PMID 20231474 (nih.gov).
- ↑ a b La Scola B., Marrie T. J., Auffray J. P., Raoult D.: Mimivirus in pneumonia patients. In: Emerg Infect Dis. 11. Jahrgang, Nr. 3, 2005, S. 449—452, PMID 1575756 3 (nih.gov).
- ↑ Marrie TJ, Durant H, Yates L: Community-Acquired Pneumonia Requiring Hospitalization: 5-Year Prospective Study. In: Reviews of Infectious Diseases. 11. Jahrgang, Nr. 4, 1989, S. 586–99, doi:10.1093/clinids/11.4.586, PMID 2772465.
- ↑ Saadi H, Pagnier I, Colson P, Cherif JK, Beji M, Boughalmi M, Azza S, Armstrong N, Robert C, Fournous G, La Scola B, Raoult D: First isolation of Mimivirus in a patient with pneumonia. In: Clinical Infectious Diseases. 57. Jahrgang, Nr. 4, August 2013, S. e127–34, doi:10.1093/cid/cit354, PMID 23709652.
- ↑ Vincent A., La Scola B., Papazian L.: Advances in Mimivirus pathogenicity. In: Intervirology. 53. Jahrgang, Nr. 5, 2010, S. 304—309, doi:10.1159/000312915, PMID 20551682.
- ↑ Ghigo E., Kartenbeck J., Lien P., Pelkmans L., Capo C., Mege J. L., Raoult D.: Ameobal pathogen mimivirus infects macrophages through phagocytosis. In: PLoS Pathog. 4. Jahrgang, Nr. 6, 2008, S. e1000087, doi:10.1371/journal.ppat.1000087, PMID 18551172 (nih.gov).
- ↑ Berger P., Papazian L., Drancourt M., La Scola B., Auffray J. P., Raoult D.: Ameba-associated microorganisms and diagnosis of nosocomial pneumonia. In: Emerg Infect Dis. 12. Jahrgang, Nr. 2, 2006, S. 248—255, PMID 16494750 (nih.gov).
- ↑ Raoult D., Renesto P., Brouqui P.: Laboratory infection of a technician by mimivirus. In: Ann Intern Med. 144. Jahrgang, Nr. 9, 2006, S. 702—703, PMID 16670147 (annals.org).
- ↑ Vanspauwen M. J. et al.: Infections with mimivirus in patients with chronic obstructive pulmonary disease. In: Respiratory Medicine. 106. Jahrgang, Nr. 12, 2012, S. 1690—1694, doi:10.1016/j.rmed.2012.08.019 (sciencedirect.com).
- ↑ a b c NCBI: Acanthamoeba polyphaga mimivirus (Spezies)
- ↑ a b Masaharu Takemura, Tatsuya Mikami, Shingo Murono: Nearly Complete Genome Sequences of Two Mimivirus Strains Isolated from a Japanese Freshwater Pond and River Mouth, in: Genome Announc. 4(6), November/Dezember 2016, e01378-16, doi:10.1128/genomeA.01378-16, PMC 5146454 (freier Volltext), PMID 27932662. Die beiden Viruslinien sind nac Angaben der Autoren eng mit Mimivirus Bombay verwandt, gehören also ebenfalls zur Spezies APMV.
- ↑ Rafael K Campos, Paulo V Boratto, Felipe L Assis, Eric RGR Aguiar, Lorena CF Silva, Jonas D Albarnaz, Fabio P Dornas, Giliane S Trindade, Paulo P Ferreira, João T Marques, Catherine Robert, Didier Raoult, Erna G Kroon, Bernard La Scola, Jônatas S Abrahão: Samba virus: a novel mimivirus from a giant rain forest, the Brazilian Amazon, in: Virology Journal 2014 11:95, doi:10.1186/1743-422X-11-95
- ↑ a b c Felipe L. Assis, Leena Bajrai, Jonatas S. Abrahao, Erna G. Kroon, Fabio P. Dornas, Kétyllen R. Andrade, Paulo V. M. Boratto, Mariana R. Pilotto, Catherine Robert, Samia Benamar, Bernard La Scola, Philippe Colson: Pan-Genome Analysis of Brazilian Lineage A Amoebal Mimiviruses, in: Viruses 7(7), 2015, S. :3483-3499, doi:10.3390/v7072782. Die hier abgehandelten Kandidaten stellen nach den Autoren eine Vewandtschaftsgruppe dar, wobei aus den anderen Quellen hervorgeht, dass Samba- und Kroon-Virus zur Spezies APMV gehören. Das gilt dann für die beiden anderen.
- ↑ Paulo Victor Miranda Boratto, Fábio Pio Dornas, Lorena Christine Ferreira da Silva, Rodrigo Araújo Lima Rodrigues, Graziele Pereira Oliveira, Juliana Reis Cortines, Betânia Paiva Drumond, Jônatas Santos Abrahão: Analyses of the Kroon Virus Major Capsid Gene and Its Transcript Highlight a Distinct Pattern of Gene Evolution and Splicing among Mimiviruses, in: J Virol. 92(2), 15. Januar 2018, e01782-17, doi:10.1128/JVI.01782-17, PMC 5752926 (freier Volltext), PMID 29118120
- ↑ NCBI: Saudi moumouvirus
- ↑ Leena H. Bajrai, Felipe L. de Assis, Esam I. Azhar, Priscilla Jardot, Catherine Robert, Jônatas Abrahão, Didier Raoult, Bernard La Scola: Saudi Moumouvirus, the First Group B Mimivirus Isolated from Asia, in: Front. Microbiol., 20. Dezember 2016, doi:10.3389/fmicb.2016.02029
- ↑ NCBI: Acanthamoeba castellanii mamavirus (Species)
- ↑ NCBI: Hirudovirue strain sangsue
- ↑ Mondher Boughalmi, Isabelle Pagnier, Sarah Aherfi, Philippe Colson, Didier Raoult, Bernard La Scola: First Isolation of a Giant Virus from Wild Hirudo medicinalis Leech: Mimiviridae isolation in Hirudo medicinalis, in: Viruses 2013, 5, S. 2920–2930, doi:10.3390/v5122920
- ↑ NCBI: Niemeyer virus (Species)
- ↑ Paulo V. M. Boratto, Thalita S. Arantes, Lorena C. F. Silva, Felipe L. Assis, Erna G. Kroon, Bernard La Scola, Jônatas S. Abrahão: Niemeyer Virus: A New Mimivirus Group A Isolate Harboring a Set of Duplicated Aminoacyl-tRNA Synthetase Genes, in: Front Microbiol. 2015; 6: 1256. doi:10.3389/fmicb.2015.01256, PMC 4639698 (freier Volltext), PMID 26635738
- ↑ NCBI: Mimivirus battle86 (Species)
- ↑ a b c d e Anirvan Chatterjee, Thomas Sicheritz-Pontén, Rajesh Yadav, Kiran Kondabagil: Isolation and complete genome sequencing of Mimivirus bombay, a Giant Virus in sewage of Mumbai, India, in: Genomics Data 9(C), Mai 2016, doi:10.1016/j.gdata.2016.05.013, Fig. 1
- ↑ NCBI: Mimivirus Cher (Species)
- ↑ NCBI: Mimivirus fauteuil
- ↑ a b c d e f Christelle Desnues, Bernard La Scola, Natalya Yutin, Ghislain Fournous et al.: Provirophages and transpovirons as the diverse mobilome of giant viruses, in: PNAS 109(44), 30. Oktober 2012,S. 18078–18083, doi:10.1073/pnas.1208835109
- ↑ NCBI: Fauteuil virus FD
- ↑ NCBI: Mimivirus lactour (Species)
- ↑ NCBI: Lactours virus LT2 (Species)
- ↑ NCBI: Mimivirus lentille (Species)
- ↑ NCBI: Lentille virus CL (Species)
- ↑ NCBI: Mimivirus longchamps (Species)
- ↑ NCBI: Longchamps virus FPL (Species)
- ↑ NCBI: Mimivirus marais (Species)]
- ↑ NCBI: Mimivirus montadette2 (Species)
- ↑ NCBI: Mimivirus pointerouge1 (Species)
- ↑ NCBI: Pointerouge virus 1 (Species)
- ↑ NCBI: Mimivirus terra2 (Spezies)
- ↑ NCBI: Mimivirus univirus (Species)
- ↑ Anthony Levasseur, Meriem Bekliz, Eric Chabrière, Pierre Pontarotti, Bernard La Scola, Didier Raoult: MIMIVIRE is a defence system in mimivirus that confers resistance to virophage. In: Nature, 2016, doi:10.1038/nature17146.
- ↑ Ewen Callaway: CRISPR-like 'immune' system discovered in giant virus, in: Nature:News, vom 29. Juni 2016