„Hepatitis-B-Virus“ – Versionsunterschied
| [gesichtete Version] | [gesichtete Version] |
K Wikisyntax |
Ghilt (Diskussion | Beiträge) neu / aus EN-Wiki übersetzt, + Bild |
||
| Zeile 1: | Zeile 1: | ||
{{Infobox Virus |
|||
#REDIRECT [[Hepatitis B]] |
|||
| Name= Hepatitis-B-Virus |
|||
| Bild= Hepatitis B virus 01.jpg |
|||
| Bild_legende = |
|||
| Wiss_Name = Hepatitis B virus |
|||
| Wiss_KurzName = HBV |
|||
| Ordnung = |
|||
| Familie = Hepadnaviridae |
|||
| Subfamilie = |
|||
| Gattung = Orthohepadnaviridae |
|||
| Spezies = Hepatitis-B-Virus |
|||
| Subspezies = |
|||
| Genom = dsDNA |
|||
| Baltimore = 1 |
|||
| Kapsid = |
|||
| Virushülle = |
|||
| NCBI_Tax = |
|||
| NCBI_Ref = |
|||
| ICTV = |
|||
}} |
|||
Das '''Hepatitis-B-Virus''' (HBV) ist ein [[Virushülle|behülltes]] [[DNA-Virus]] mit überwiegend doppelsträngigem [[Genom]] und der Auslöser der [[Hepatitis B]].<ref name="pmid18824707" /> Das Hepatitis-B-Virus entstammt der Gattung der ''Orthohepadnaviren'' innerhalb der Familie der ''Hepadnaviridae''.<ref name="RichardHuntUSC">{{cite web | url=http://pathmicro.med.sc.edu/virol/hepatitis-virus.htm | title=Hepatitis viruses | accessdate=2008-03-13 | last=Hunt | first=Richard | date=2007-11-21 | publisher=University of Southern California, Department of Pathology and Microbiology }}</ref> |
|||
== Erkrankung == |
|||
{{Hauptartikel|Hepatitis B}} |
|||
Zusätzlich zur Hepatitis B verursacht HBV in chronischen Verläufen eine [[Leberzirrhose]] und [[Leberkrebs]].<ref name="pmid18263618">{{cite journal |author=Schwalbe M, Ohlenschläger O, Marchanka A, ''et al.'' |title=Solution structure of stem-loop alpha of the hepatitis B virus post-transcriptional regulatory element |journal=Nucleic Acids Res. |volume=36 |issue=5 |pages=1681–9 |year=2008 |month=March |pmid=18263618 |pmc=2275152 |doi=10.1093/nar/gkn006 |url=http://nar.oxfordjournals.org/cgi/pmidlookup?view=long&pmid=18263618}}</ref> Möglicherweise existiert auch ein Zusammenhang bei [[Pankreaskrebs]].<ref name="pmid18824707">{{cite journal |author=Hassan MM, Li D, El-Deeb AS, ''et al.'' |title=Association between hepatitis B virus and pancreatic cancer |journal=J. Clin. Oncol. |volume=26 |issue=28 |pages=4557–62 |year=2008 |month=October |pmid=18824707 |pmc=2562875 |doi=10.1200/JCO.2008.17.3526 |url=http://www.jco.org/cgi/pmidlookup?view=long&pmid=18824707}}</ref> |
|||
== Klassifizierung == |
|||
HBV ist ein typischer Vertreter der ''[[Orthohepadnaviren]]'', die drei weitere Vertreter aufweist: das ''Ground squirrel hepatitis virus'', das ''Woodchuck hepatitis virus'', und das ''Woolly monkey hepatitis B virus''. Die Familie der ''[[Hepadnaviridae]]'' enthält noch zwei weitere Gattungen, die ''[[Avihepadnaviren]]'' und eine noch zu Benennende. Die Familie ist noch keiner Ordnung zugeordnet.<ref>{{cite web | url=http://www.ncbi.nlm.nih.gov/ICTVdb/Ictv/fs_hepad.htm | title=00.030. Hepadnaviridae |work=ICTVdB Index of Viruses | accessdate=2009-03-13 | author=Mason, W.S., ''et al.'' | date=2008-07-08 | publisher=International Committee on Taxonomy of Viruses }}</ref> HBV-ähnliche Viren wurden bereits in allen [[Altweltaffe]]n gefunden ([[Orang-Utan]], [[Gibbon]]s, [[Gorilla]]s and [[Schimpanse]]n) und auch in Neuweltaffen wie dem [[Wollaffe]]n nachgewiesen, was einen älteren Ursprung dieses Virus in Primaten nahelegt. |
|||
HBV werden in vier [[Serotyp]]en (adr, adw, ayr, ayw) eingeteilt, je nach den [[antigen]]en [[Epitop]]en auf ihren Hüllproteinen, genetisch werden sie, je nach ihren [[Mutation]]en, acht Genotypen (A-H) zugeordnet. Die Genotype haben eine unterschiedliche geografische Verteilung und werden zur epidemiologischen Bestimmung der Transmission und der viralen Evolution herangezogen. Die unterschiedlichen Genotype weisen auch Unterschiede in den [[Krankheitsverlauf|Krankheitsverläufen]], der Häufgikeit von [[Komplikation]]en sowie in der [[Therapie]]- und der [[Impfstoff]]wahl.<ref name="pmid15752827">{{cite journal |author=Kramvis A, Kew M, François G |title=Hepatitis B virus genotypes |journal=Vaccine |volume=23 |issue=19 |pages=2409–23 |year=2005 |pmid=15752827 |doi=10.1016/j.vaccine.2004.10.045}}</ref><ref name="pmid8666521">{{cite journal |author=Magnius LO, Norder H |title=Subtypes, genotypes and molecular epidemiology of the hepatitis B virus as reflected by sequence variability of the S-gene |journal=Intervirology |volume=38 |issue=1–2 |pages=24–34 |year=1995 |pmid=8666521 |doi=}}</ref> |
|||
== Aufbau == |
|||
==== Virion ==== |
|||
Das [[Virion]] besitzt eine [[Virushülle]] und ein ikosahedrales [[Capsid]]. Das Capsid umschließt die virale DNA und eine [[DNA-Polymerase]] mit einer [[Reverse Transkriptase]]-Aktivität (RT), ähnlich einer [[Retrovirus|retroviralen]] Polymerase.<ref name="pmid15192795">{{cite journal |author=Locarnini S |title=Molecular virology of hepatitis B virus |journal=Semin. Liver Dis. |volume=24 |issue=Suppl 1 |pages=3–10 |year=2004 |pmid=15192795 |doi=10.1055/s-2004-828672}}</ref> Die Virushülle enthält [[Membranprotein]]e zur Bindung an die [[Tropismus (Virologie)|Wirtszelle]]. Mit einem Durchmesser von 42 nm gehört das sphärische HBV zu den kleineren Tierviren, es existieren aber auch nicht-infektiöse filamentöse und sphärische Formen ohne Capsid. Diese [[Virus-like particle|virusartigen Partikel]] bestehen aus [[HBsAg]] in einer [[Lipiddoppelschicht]].<ref name="pmid3014045">{{cite journal |author=Howard CR |title=The biology of hepadnaviruses |journal=J. Gen. Virol. |volume=67 |issue= 7|pages=1215–35 |year=1986 |pmid=3014045 |doi=10.1099/0022-1317-67-7-1215}}</ref> |
|||
==== Bestandteile ==== |
|||
HBV besteht aus: |
|||
* [[HBsAg]] |
|||
* [[HBcAg]] (HBeAg ist eine [[Alternatives Spleißen|Spleißvariante]]) |
|||
* [[Hepatitis-B-Virus DNA-Polymerase]] |
|||
* [[HBx]] |
|||
Das [[Hepatitis-D-Virus]] benötigt HBcAg, um [[pathogen]] zu werden.<ref name="pmid18524834">{{cite journal |author=Chai N, Chang HE, Nicolas E, Han Z, Jarnik M, Taylor J |title=Properties of subviral particles of hepatitis B virus |journal=J. Virol. |volume=82 |issue=16 |pages=7812–7 |year=2008 |month=August |pmid=18524834 |pmc=2519590 |doi=10.1128/JVI.00561-08 |url=http://jvi.asm.org/cgi/pmidlookup?view=long&pmid=18524834}}</ref> |
|||
== Evolution == |
|||
Die genaue Bestimmung der frühen Evolution des HBV ist problematisch. Die Aufspaltung in der Hepadnaviren in Ortho- und Avihepadnaviren erfolgte vor ungefähr 125.000 Jahren (95% Intervall 78,297–313,500).<ref name=van_Hemert2011>{{cite journal |author=van Hemert FJ, van de Klundert MA, Lukashov VV, Kootstra NA, Berkhout B, Zaaijer HL |title=Protein X of hepatitis B virus: origin and structure similarity with the central domain of DNA glycosylase |journal=PLoS ONE |volume=6 |issue=8 |page=e23392 |year=2011 |pmid=21850270 |pmc=3153941 |doi=10.1371/journal.pone.0023392 |url=http://dx.plos.org/10.1371/journal.pone.0023392}}</ref> Sowohl Avi- als auch Orthohepadnaviren begannen etwa vor 25.000 Jahren, sich stärker zu verändern.<ref name=van_Hemert2011>van Hemert FJ, van de Klundert MA, Lukashov VV, Kootstra NA, Berkhout B, Zaaijer HL (2011) Protein x of hepatitis B virus: origin and structure similarity with the central domain of DNA glycosylase. PLoS One. 2011;6(8):e23392.</ref> Zu dieser Zeit erfolgte die Aufspaltung in die acht Genotype. Humane Hepadnaviren trennten sich etwa vor 7.000 (95% Intervall: 5,287–9,270) bis 10.000 Jahren (95% Intervall: 6,305–16,681). Den Avihepadnaviren fehlt das X-Protein, jedoch sind noch Reste im Entenhepadnavirus zu finden.<ref name=Lin2000>{{cite journal |author=Lin B, Anderson DA |title=A vestigial X open reading frame in duck hepatitis B virus |journal=Intervirology |volume=43 |issue=3 |pages=185–90 |year=2000 |pmid=11044813 |url=http://content.karger.com/produktedb/produkte.asp?DOI=25037&typ=pdf}}</ref> Möglicherweise entstammt das X-Protein einer [[DNA-Glykosylase]]. Die Rate nicht-synonymer Mutationen des HBV wurde auf etwa 2×10<sup>−5</sup> Aminosäuresubstitutionen pro Stelle pro Jahr geschätzt.<ref name=Osiowy2006>{{cite journal |author=Osiowy C, Giles E, Tanaka Y, Mizokami M, Minuk GY |title=Molecular evolution of hepatitis B virus over 25 years |journal=J. Virol. |volume=80 |issue=21 |pages=10307–14 |year=2006 |month=November |pmid=17041211 |pmc=1641782 |doi=10.1128/JVI.00996-06 |url=http://jvi.asm.org/cgi/pmidlookup?view=long&pmid=17041211}}</ref> Die mittlere Häufigkeit von Nukleotidsubstitutionen pro Stelle pro Jahr wurde mit etwa 7.9 x 10<sup>-5</sup> beziffert. Eine andere Schätzung vermutet den letzten gemeinsamen Vorfahren der humanen HBV-Stämme vor 1.500 Jahren und eine Trennung von den Avihepadnaviren vor 6.000 Jahren bei einer Mutationsrate von etwa 10<sup>-6</sup> Substitutionen pro Stelle pro Jahr.<ref name=Zhou2007>Zhou Y, Holmes EC (2007) Bayesian estimates of the evolutionary rate and age of hepatitis B virus. J Mol Evol 65(2):197-205</ref> |
|||
== Genom == |
|||
[[Datei:HBV_Genome.svg|miniatur|Das Genom des HBV. Die Gene überlappen.]] |
|||
Das [[Genom]] des HBV besteht aus zirkulär-geschlossener [[DNA]], jedoch ist die DNA nicht vollständig doppelsträngig und ein Strang durchgängig. Ein Ende des durchgängigen Strangs von circa 3020-3320 [[Nukleotid]]e bindet die virale DNA-Polymerase, während der kürzere Strang mit 1700-2800 Nukleotiden deutlich kürzer ist.<ref>{{cite journal |author=Kay A, Zoulim F |title=Hepatitis B virus genetic variability and evolution |journal=Virus Res. |volume=127 |issue=2 |pages=164–76 |year=2007 |pmid=17383765 |doi=10.1016/j.virusres.2007.02.021}}</ref> |
|||
Der durchgängige [[Polarität (Virologie)|(-)-Strang]] (nicht-codierend) ist revers-komplementär zur viralen [[mRNA]]. Die virale DNA ist bereits kurz nach der Infektion im [[Zellkern]] nachweisbar. Die teilweise doppelsträngige DNA wird am (+)-Strang vervollständigt und eine [[RNA]]-Sequenz am (+)-Strang entfernt. Zuletzt werden überhängende Nukleotide entfernt und der (-)-Strang zum Ring geschlossen. |
|||
Die vier Gene des HBV werden als C (''core''), X, P (''polymerase'') und S (''surface'') bezeichnet. Das ''core''-Protein HbcAg wird aus einem [[Präprotein]] durch [[Proteolyse]] erzeugt, wodurch auch das HBeAg (''excretory'') entsteht. Das Gen S des Proteins HBsAg enthält drei [[Leseraster]]-konforme Start-Codons (AUG), wodurch drei Proteine entstehen, pre-S1, pre-S2 und S.<ref name="pmid17206754">{{cite journal |author=Beck J, Nassal M |title=Hepatitis B virus replication |journal=World J. Gastroenterol. |volume=13 |issue=1 |pages=48–64 |year=2007 |pmid=17206754 |doi=}}</ref> Über die Funktion des X-Proteins ist bisher wenig bekannt.<ref name="pmid19208517">{{cite journal |author=Guo GH, Tan DM, Zhu PA, Liu F |title=Hepatitis B virus X protein promotes proliferation and upregulates TGF-beta1 and CTGF in human hepatic stellate cell line, LX-2 |journal=Hbpd Int |volume=8 |issue=1 |pages=59–64 |year=2009 |month=February |pmid=19208517 |url=http://www.hbpdint.com/text.asp?id=1196}}</ref><ref name="pmid15542625">{{cite journal |author=Bouchard MJ, Schneider RJ |title=The enigmatic X gene of hepatitis B virus |journal=J. Virol. |volume=78 |issue=23 |pages=12725–34 |year=2004 |pmid=15542625 |doi=10.1128/JVI.78.23.12725-12734.2004 |pmc=524990}}</ref> |
|||
Verschiedene nicht-codierende RNA wurden im Genom des HBV identifiziert. Diese werden als ''Hepatitis B virus PRE alpha'', ''Hepatitis B virus PRE beta'' und ''HBV RNA encapsidation signal epsilon'' bezeichnet.<ref>{{cite journal| doi= 10.1093/nar/26.21.4818| last1= Smith Gj| first1= 3rd| last2= Donello| first2= JE| last3= Lück| first3= R| last4= Steger| first4= G| last5= Hope| first5= TJ| title= The hepatitis B virus post-transcriptional regulatory element contains two conserved RNA stem-loops which are required for function| journal= Nucleic Acids Research| volume= 26| issue= 21| pages= 4818–27| year= 1998 | pmid = 9776740| pmc= 147918}}</ref><ref>{{cite journal |author=Flodell S, Schleucher J, Cromsigt J, Ippel H, Kidd-Ljunggren K, Wijmenga S |title=The apical stem-loop of the hepatitis B virus encapsidation signal folds into a stable tri-loop with two underlying pyrimidine bulges |journal=Nucleic Acids Res. |volume=30 |issue=21 |pages=4803–11 |year=2002 |month=November |pmid=12409471 |pmc=135823 |doi= 10.1093/nar/gkf603|url=http://nar.oxfordjournals.org/cgi/pmidlookup?view=long&pmid=12409471}}</ref> |
|||
==== Genotypen ==== |
|||
[[Datei:HBV prevalence 2005.png|miniatur|Globale Verteilung des HBV im Menschen]] |
|||
Die acht Genotype des HBV werden A bis H benannt.<ref name="pmid15752827">{{cite journal |author=Kramvis A, Kew M, François G |title=Hepatitis B virus genotypes |journal=Vaccine |volume=23 |issue=19 |pages=2409–23 |year=2005 |month=March |pmid=15752827 |doi=10.1016/j.vaccine.2004.10.045 |url=http://linkinghub.elsevier.com/retrieve/pii/S0264-410X(04)00849-7}}</ref> Möglicherweise existiert auch ein Genotyp I,<ref name="pmid18976569">{{cite journal |author=Olinger CM, Jutavijittum P, Hübschen JM, ''et al.'' |title=Possible new hepatitis B virus genotype, southeast Asia |journal=Emerging Infect. Dis. |volume=14 |issue=11 |pages=1777–80 |year=2008 |month=November |pmid=18976569 |pmc=2630741 |doi= 10.3201/eid1411.080437|url=http://www.cdc.gov/eid/content/14/11/1777.htm}}</ref> was jedoch nicht einhellig akzeptiert wird.<ref name="pmid18663008">{{cite journal |author=Kurbanov F, Tanaka Y, Kramvis A, Simmonds P, Mizokami M |title=When should "I" consider a new hepatitis B virus genotype? |journal=J. Virol. |volume=82 |issue=16 |pages=8241–2 |year=2008 |month=August |pmid=18663008 |pmc=2519592 |doi=10.1128/JVI.00793-08 |url=http://jvi.asm.org/cgi/pmidlookup?view=long&pmid=18663008}}</ref> Die unterschiedlichen Genotypen sprechen unterschiedlich auf Therapien an.<ref name="pmid17515708">{{cite journal |author=Palumbo E |title=Hepatitis B genotypes and response to antiviral therapy: a review |journal=Am J Ther |volume=14 |issue=3 |pages=306–9 |year=2007 |pmid=17515708 |doi=10.1097/01.pap.0000249927.67907.eb |url=http://meta.wkhealth.com/pt/pt-core/template-journal/lwwgateway/media/landingpage.htm?an=00045391-200705000-00016}}</ref><ref name="pmid18842489">{{cite journal |author=Mahtab MA, Rahman S, Khan M, Karim F |title=Hepatitis B virus genotypes: an overview |journal=Hbpd Int |volume=7 |issue=5 |pages=457–64 |year=2008 |month=October |pmid=18842489 |url=http://www.hbpdint.com/text.asp?id=1142}}</ref> |
|||
Die Genotypen unterscheiden sich mindestens um acht Prozent auf der Genomebene und haben unterschiedliche geographische Häufigkeiten. Der Genotyp F unterscheidet sich von den anderen am stärksten, mit vierzehn Prozent Unterschied. Der Genotyp A ist [[Prävalenz|prävalent]] in [[Europa]], [[Afrika]] and [[Südasien]], einschließlich der [[Philippinen]]. Die Genotypen B und C sind vorherrschend in [[Asien]]; der Genotyp D tritt im [[Mittelmeerraum]], dem [[Mittlerer Osten|mittleren Osten]] und [[Indien]] auf. Der Genotyp E existiert in [[Afrika]] südlich der Sahara. Der Genotyp F (oder H) kommt in Zentral- und [[Südamerika]] vor. Der Genotyp G wurde in [[Frankreich]] und [[Deutschland]] gefunden. Die Genotypen A, D und F dominieren in [[Brasilien]] und alle Genotypen kommen mit ethnischen Häufungen in den [[Vereinigten Staaten]] vor. |
|||
Innerhalb der Genotypen sind 24 Subtypen beschrieben, die in ihrer DNA-Sequenz untereinander um vier bis acht Prozent abweichen. |
|||
* Genotyp A hat zwei Subtypen: Aa (A1) in Afrika/Asien und Ae (A2) in Europa/USA. |
|||
* Genotyp B besitzt zwei geographisch getrennte Subtypen: Bj/B1 ('j' — Japan) und Ba/B2 ('a' — Asien). Der Genotyp Ba wird weiter in vier ''clades'' unterteilt (B2 — B4). |
|||
* Genotyp C hat ebenso zwei geographisch getrennte Subtypen: Cs (C1) in Südostasien und Ce (C2) in Ostasien. Der C-Subtyp wird in fünf ''clades'' unterteilt, während ein möglicher Sechster (C6) und ein Siebter (C7) bisher nur in einem Isolat in den Philippinen und auf West-Papua gefunden wurden.<ref name="Cavinta2009">{{cite journal |author=Cavinta L, Sun J, May A, ''et al.'' |title=A new isolate of hepatitis B virus from the Philippines possibly representing a new subgenotype C6 |journal=J. Med. Virol. |volume=81 |issue=6 |pages=983–7 |year=2009 |month=June |pmid=19382274 |doi=10.1002/jmv.21475 }}</ref><ref name="Lusida2008">{{cite journal | author = Lusida M.I., Nugrahaputra V.E., Soetjipto Handajani R., Nagano-Fujii M., Sasayama M., Utsumi T., Hotta H. | year = 2008 | title = Novel subgenotypes of hepatitis B virus genotypes C and D in Papua, Indonesia | journal = J. Clin. Microbiol | volume = 46 | issue = 7| pages = 2160–2166 | doi = 10.1128/JCM.01681-07 | pmid = 18463220 | pmc = 2446895}}</ref> Der Genotyp C1 kommt in [[Vietnam]], [[Myanmar]] und [[Thailand]] vor. Der Genotyp C2 tritt in [[Japan]], [[Korea]] und [[China]] auf. Der Genotyp C3 wird in [[Neukaledonien]] und [[Polynesien]] gefunden. Der Genotyp C4 kommt in [[Australien]] vor und C5 auf den [[Philippinen]]. |
|||
* Genotyp D wird in sieben Subtypen unterteilt (D1 — D7). |
|||
* Genotyp E hat nur einen Subtyp. |
|||
* Genotyp F ist in vier Subtypen unterteilt (F1 — F4). Der Subtyp F1 wird weiter in die ''clades'' 1a und 1b getrennt. In [[Venezuela]] werden die Subtypen F1, F2 und F3 im Westen und Osten gefunden, während F3 im Süden alleinig auftritt. Der Subtyp Ia kommt in Mittelamerika, der Subtyp III im nördlichen Südamerika und IV im südlichen Südamerika vor. Das ''clade'' Ib wird auf beinahe dem ganzen amerikanischen Kontinent (außer dem nördlichen Sudamerika) und das ''clade'' II wird in Zentral- und Südamerika gefunden. |
|||
== Replikationszyklus == |
|||
[[Datei:HBV replication.png|miniatur|Replikation des Hepatitis-B-Virus.]] |
|||
Der [[Replikation|Replikationszyklus]] des HBV: |
|||
* [[Adsorption|Anheftung]] und anschließende [[Endozytose]] über einen Rezeptor aus der Klasse der Ovalbumin-[[Serpin]]e.<ref name="RichardHuntUSC" /> |
|||
* Penetration durch eine induzierte Fusion der Virushülle und der [[Zellmembran]], wodurch das ''core'' mit der DNA des Virus ins [[Zytosol]] freigesetzt wird. |
|||
* Entpacken und Transport in den [[Zellkern]] über [[Chaperon]]e, weil eine DNA-Polymerase des Wirts zur eigenen Replikation genutzt wird. Dadurch erfolgt die Freisetzung der DNA aus dem ''core'' und anschließend das Vervollständigen des (-)-Strangs und der Ringschluss zur [[cccDNA]], welche als Vorlage für die Transkription der vier viralen mRNA dient. |
|||
* [[Replikation]], bei der die längste mRNA (länger als das virale Genom) als Vorlage für die Synthese des DNA-Genoms dient, sowie zur [[Proteinbiosynthese]] des Capsids und der viralen Polymerase. |
|||
* Zusammenbau anhand einer der vier viralen mRNA, auf die eine Abschnürung aus der Zelle oder ein Rücktransport zum Zellkern zur weiteren Genomsynthese erfolgt.<ref name="pmid17206754"/><ref name="pmid17206755">{{cite journal |author=Bruss V |title=Hepatitis B virus morphogenesis |journal=World J. Gastroenterol. |volume=13 |issue=1 |pages=65–73 |year=2007 |pmid=17206755 |doi=}}</ref> |
|||
* Freisetzung der Tochtervirionen bei gleichzeitiger Synthese des DNA-Genoms im Zytoplasma aus der längsten mRNA durch die RT-Aktivität der viralen Polymerase. |
|||
== Literatur == |
|||
* S. Modrow, [[Dietrich Falke]], U. Truyen: Molekulare Virologie. 2. Auflage, Spektrum, Heidelberg 2003, ISBN 3-8274-1086-X. |
|||
* D. M. Knipe, [[Peter M. Howley]] (Hrsg.): ''Fields Virology''. 5. Auflage, Philadelphia 2007. ISBN 0-7817-6060-7. |
|||
== Einzelnachweise == |
|||
<references /> |
|||
[[Kategorie:Virusspezies]] |
|||
[[Kategorie:Hepatologie]] |
|||
[[Kategorie: Meldepflichtiger Erreger]] |
|||
[[ar:التهاب الكبد الفيروسي ب]] |
|||
[[ca:Virus de l'hepatitis B]] |
|||
[[cs:Virus hepatitidy B]] |
|||
[[en:Hepatitis B virus]] |
|||
[[es:Virus de la hepatitis B]] |
|||
[[fr:Virus de l'hépatite B]] |
|||
[[it:Hepatitis B virus]] |
|||
[[kn:ಹೆಪಟೈಟಿಸ್ ಬಿ ವೈರಾಣು]] |
|||
[[ht:Viris epatit B]] |
|||
[[mn:Элэгний B вирус]] |
|||
[[ja:B型肝炎ウイルス]] |
|||
[[no:Hepatitt B-virus]] |
|||
[[pl:Wirus zapalenia wątroby typu B]] |
|||
[[pt:Vírus da hepatite B]] |
|||
[[ru:Вирус гепатита B]] |
|||
[[sl:Virus hepatitisa B]] |
|||
[[ta:ஈரலழற்சி பி தீநுண்மம்]] |
|||
[[tr:Hepatit B virüsü]] |
|||
[[vi:Virus viêm gan siêu vi B]] |
|||
[[zh:乙型肝炎病毒]] |
|||
Version vom 1. Juni 2012, 14:44 Uhr
| Hepatitis-B-Virus | ||||||||
|---|---|---|---|---|---|---|---|---|
Hepatitis-B-Virus | ||||||||
| Systematik | ||||||||
| ||||||||
| Taxonomische Merkmale | ||||||||
| ||||||||
| Wissenschaftlicher Name | ||||||||
| Hepatitis B virus | ||||||||
| Kurzbezeichnung | ||||||||
| HBV |
Das Hepatitis-B-Virus (HBV) ist ein behülltes DNA-Virus mit überwiegend doppelsträngigem Genom und der Auslöser der Hepatitis B.[1] Das Hepatitis-B-Virus entstammt der Gattung der Orthohepadnaviren innerhalb der Familie der Hepadnaviridae.[2]
Erkrankung
Zusätzlich zur Hepatitis B verursacht HBV in chronischen Verläufen eine Leberzirrhose und Leberkrebs.[3] Möglicherweise existiert auch ein Zusammenhang bei Pankreaskrebs.[1]
Klassifizierung
HBV ist ein typischer Vertreter der Orthohepadnaviren, die drei weitere Vertreter aufweist: das Ground squirrel hepatitis virus, das Woodchuck hepatitis virus, und das Woolly monkey hepatitis B virus. Die Familie der Hepadnaviridae enthält noch zwei weitere Gattungen, die Avihepadnaviren und eine noch zu Benennende. Die Familie ist noch keiner Ordnung zugeordnet.[4] HBV-ähnliche Viren wurden bereits in allen Altweltaffen gefunden (Orang-Utan, Gibbons, Gorillas and Schimpansen) und auch in Neuweltaffen wie dem Wollaffen nachgewiesen, was einen älteren Ursprung dieses Virus in Primaten nahelegt.
HBV werden in vier Serotypen (adr, adw, ayr, ayw) eingeteilt, je nach den antigenen Epitopen auf ihren Hüllproteinen, genetisch werden sie, je nach ihren Mutationen, acht Genotypen (A-H) zugeordnet. Die Genotype haben eine unterschiedliche geografische Verteilung und werden zur epidemiologischen Bestimmung der Transmission und der viralen Evolution herangezogen. Die unterschiedlichen Genotype weisen auch Unterschiede in den Krankheitsverläufen, der Häufgikeit von Komplikationen sowie in der Therapie- und der Impfstoffwahl.[5][6]
Aufbau
Virion
Das Virion besitzt eine Virushülle und ein ikosahedrales Capsid. Das Capsid umschließt die virale DNA und eine DNA-Polymerase mit einer Reverse Transkriptase-Aktivität (RT), ähnlich einer retroviralen Polymerase.[7] Die Virushülle enthält Membranproteine zur Bindung an die Wirtszelle. Mit einem Durchmesser von 42 nm gehört das sphärische HBV zu den kleineren Tierviren, es existieren aber auch nicht-infektiöse filamentöse und sphärische Formen ohne Capsid. Diese virusartigen Partikel bestehen aus HBsAg in einer Lipiddoppelschicht.[8]
Bestandteile
HBV besteht aus:
- HBsAg
- HBcAg (HBeAg ist eine Spleißvariante)
- Hepatitis-B-Virus DNA-Polymerase
- HBx
Das Hepatitis-D-Virus benötigt HBcAg, um pathogen zu werden.[9]
Evolution
Die genaue Bestimmung der frühen Evolution des HBV ist problematisch. Die Aufspaltung in der Hepadnaviren in Ortho- und Avihepadnaviren erfolgte vor ungefähr 125.000 Jahren (95% Intervall 78,297–313,500).[10] Sowohl Avi- als auch Orthohepadnaviren begannen etwa vor 25.000 Jahren, sich stärker zu verändern.[10] Zu dieser Zeit erfolgte die Aufspaltung in die acht Genotype. Humane Hepadnaviren trennten sich etwa vor 7.000 (95% Intervall: 5,287–9,270) bis 10.000 Jahren (95% Intervall: 6,305–16,681). Den Avihepadnaviren fehlt das X-Protein, jedoch sind noch Reste im Entenhepadnavirus zu finden.[11] Möglicherweise entstammt das X-Protein einer DNA-Glykosylase. Die Rate nicht-synonymer Mutationen des HBV wurde auf etwa 2×10−5 Aminosäuresubstitutionen pro Stelle pro Jahr geschätzt.[12] Die mittlere Häufigkeit von Nukleotidsubstitutionen pro Stelle pro Jahr wurde mit etwa 7.9 x 10-5 beziffert. Eine andere Schätzung vermutet den letzten gemeinsamen Vorfahren der humanen HBV-Stämme vor 1.500 Jahren und eine Trennung von den Avihepadnaviren vor 6.000 Jahren bei einer Mutationsrate von etwa 10-6 Substitutionen pro Stelle pro Jahr.[13]
Genom
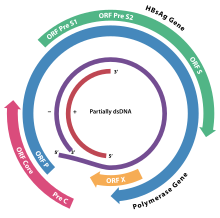
Das Genom des HBV besteht aus zirkulär-geschlossener DNA, jedoch ist die DNA nicht vollständig doppelsträngig und ein Strang durchgängig. Ein Ende des durchgängigen Strangs von circa 3020-3320 Nukleotide bindet die virale DNA-Polymerase, während der kürzere Strang mit 1700-2800 Nukleotiden deutlich kürzer ist.[14]
Der durchgängige (-)-Strang (nicht-codierend) ist revers-komplementär zur viralen mRNA. Die virale DNA ist bereits kurz nach der Infektion im Zellkern nachweisbar. Die teilweise doppelsträngige DNA wird am (+)-Strang vervollständigt und eine RNA-Sequenz am (+)-Strang entfernt. Zuletzt werden überhängende Nukleotide entfernt und der (-)-Strang zum Ring geschlossen.
Die vier Gene des HBV werden als C (core), X, P (polymerase) und S (surface) bezeichnet. Das core-Protein HbcAg wird aus einem Präprotein durch Proteolyse erzeugt, wodurch auch das HBeAg (excretory) entsteht. Das Gen S des Proteins HBsAg enthält drei Leseraster-konforme Start-Codons (AUG), wodurch drei Proteine entstehen, pre-S1, pre-S2 und S.[15] Über die Funktion des X-Proteins ist bisher wenig bekannt.[16][17]
Verschiedene nicht-codierende RNA wurden im Genom des HBV identifiziert. Diese werden als Hepatitis B virus PRE alpha, Hepatitis B virus PRE beta und HBV RNA encapsidation signal epsilon bezeichnet.[18][19]
Genotypen
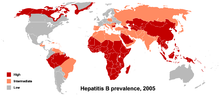
Die acht Genotype des HBV werden A bis H benannt.[5] Möglicherweise existiert auch ein Genotyp I,[20] was jedoch nicht einhellig akzeptiert wird.[21] Die unterschiedlichen Genotypen sprechen unterschiedlich auf Therapien an.[22][23]
Die Genotypen unterscheiden sich mindestens um acht Prozent auf der Genomebene und haben unterschiedliche geographische Häufigkeiten. Der Genotyp F unterscheidet sich von den anderen am stärksten, mit vierzehn Prozent Unterschied. Der Genotyp A ist prävalent in Europa, Afrika and Südasien, einschließlich der Philippinen. Die Genotypen B und C sind vorherrschend in Asien; der Genotyp D tritt im Mittelmeerraum, dem mittleren Osten und Indien auf. Der Genotyp E existiert in Afrika südlich der Sahara. Der Genotyp F (oder H) kommt in Zentral- und Südamerika vor. Der Genotyp G wurde in Frankreich und Deutschland gefunden. Die Genotypen A, D und F dominieren in Brasilien und alle Genotypen kommen mit ethnischen Häufungen in den Vereinigten Staaten vor.
Innerhalb der Genotypen sind 24 Subtypen beschrieben, die in ihrer DNA-Sequenz untereinander um vier bis acht Prozent abweichen.
- Genotyp A hat zwei Subtypen: Aa (A1) in Afrika/Asien und Ae (A2) in Europa/USA.
- Genotyp B besitzt zwei geographisch getrennte Subtypen: Bj/B1 ('j' — Japan) und Ba/B2 ('a' — Asien). Der Genotyp Ba wird weiter in vier clades unterteilt (B2 — B4).
- Genotyp C hat ebenso zwei geographisch getrennte Subtypen: Cs (C1) in Südostasien und Ce (C2) in Ostasien. Der C-Subtyp wird in fünf clades unterteilt, während ein möglicher Sechster (C6) und ein Siebter (C7) bisher nur in einem Isolat in den Philippinen und auf West-Papua gefunden wurden.[24][25] Der Genotyp C1 kommt in Vietnam, Myanmar und Thailand vor. Der Genotyp C2 tritt in Japan, Korea und China auf. Der Genotyp C3 wird in Neukaledonien und Polynesien gefunden. Der Genotyp C4 kommt in Australien vor und C5 auf den Philippinen.
- Genotyp D wird in sieben Subtypen unterteilt (D1 — D7).
- Genotyp E hat nur einen Subtyp.
- Genotyp F ist in vier Subtypen unterteilt (F1 — F4). Der Subtyp F1 wird weiter in die clades 1a und 1b getrennt. In Venezuela werden die Subtypen F1, F2 und F3 im Westen und Osten gefunden, während F3 im Süden alleinig auftritt. Der Subtyp Ia kommt in Mittelamerika, der Subtyp III im nördlichen Südamerika und IV im südlichen Südamerika vor. Das clade Ib wird auf beinahe dem ganzen amerikanischen Kontinent (außer dem nördlichen Sudamerika) und das clade II wird in Zentral- und Südamerika gefunden.
Replikationszyklus
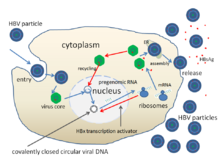
Der Replikationszyklus des HBV:
- Anheftung und anschließende Endozytose über einen Rezeptor aus der Klasse der Ovalbumin-Serpine.[2]
- Penetration durch eine induzierte Fusion der Virushülle und der Zellmembran, wodurch das core mit der DNA des Virus ins Zytosol freigesetzt wird.
- Entpacken und Transport in den Zellkern über Chaperone, weil eine DNA-Polymerase des Wirts zur eigenen Replikation genutzt wird. Dadurch erfolgt die Freisetzung der DNA aus dem core und anschließend das Vervollständigen des (-)-Strangs und der Ringschluss zur cccDNA, welche als Vorlage für die Transkription der vier viralen mRNA dient.
- Replikation, bei der die längste mRNA (länger als das virale Genom) als Vorlage für die Synthese des DNA-Genoms dient, sowie zur Proteinbiosynthese des Capsids und der viralen Polymerase.
- Zusammenbau anhand einer der vier viralen mRNA, auf die eine Abschnürung aus der Zelle oder ein Rücktransport zum Zellkern zur weiteren Genomsynthese erfolgt.[15][26]
- Freisetzung der Tochtervirionen bei gleichzeitiger Synthese des DNA-Genoms im Zytoplasma aus der längsten mRNA durch die RT-Aktivität der viralen Polymerase.
Literatur
- S. Modrow, Dietrich Falke, U. Truyen: Molekulare Virologie. 2. Auflage, Spektrum, Heidelberg 2003, ISBN 3-8274-1086-X.
- D. M. Knipe, Peter M. Howley (Hrsg.): Fields Virology. 5. Auflage, Philadelphia 2007. ISBN 0-7817-6060-7.
Einzelnachweise
- ↑ a b Hassan MM, Li D, El-Deeb AS, et al.: Association between hepatitis B virus and pancreatic cancer. In: J. Clin. Oncol. 26. Jahrgang, Nr. 28, Oktober 2008, S. 4557–62, doi:10.1200/JCO.2008.17.3526, PMID 18824707, PMC 2562875 (freier Volltext) – (jco.org).
- ↑ a b Richard Hunt: Hepatitis viruses. University of Southern California, Department of Pathology and Microbiology, 21. November 2007, abgerufen am 13. März 2008.
- ↑ Schwalbe M, Ohlenschläger O, Marchanka A, et al.: Solution structure of stem-loop alpha of the hepatitis B virus post-transcriptional regulatory element. In: Nucleic Acids Res. 36. Jahrgang, Nr. 5, März 2008, S. 1681–9, doi:10.1093/nar/gkn006, PMID 18263618, PMC 2275152 (freier Volltext) – (oxfordjournals.org).
- ↑ Mason, W.S., et al.: 00.030. Hepadnaviridae. In: ICTVdB Index of Viruses. International Committee on Taxonomy of Viruses, 8. Juli 2008, abgerufen am 13. März 2009.
- ↑ a b Kramvis A, Kew M, François G: Hepatitis B virus genotypes. In: Vaccine. 23. Jahrgang, Nr. 19, 2005, S. 2409–23, doi:10.1016/j.vaccine.2004.10.045, PMID 15752827. Referenzfehler: Ungültiges
<ref>-Tag. Der Name „pmid15752827“ wurde mehrere Male mit einem unterschiedlichen Inhalt definiert. - ↑ Magnius LO, Norder H: Subtypes, genotypes and molecular epidemiology of the hepatitis B virus as reflected by sequence variability of the S-gene. In: Intervirology. 38. Jahrgang, Nr. 1–2, 1995, S. 24–34, PMID 8666521.
- ↑ Locarnini S: Molecular virology of hepatitis B virus. In: Semin. Liver Dis. 24. Jahrgang, Suppl 1, 2004, S. 3–10, doi:10.1055/s-2004-828672, PMID 15192795.
- ↑ Howard CR: The biology of hepadnaviruses. In: J. Gen. Virol. 67. Jahrgang, Nr. 7, 1986, S. 1215–35, doi:10.1099/0022-1317-67-7-1215, PMID 3014045.
- ↑ Chai N, Chang HE, Nicolas E, Han Z, Jarnik M, Taylor J: Properties of subviral particles of hepatitis B virus. In: J. Virol. 82. Jahrgang, Nr. 16, August 2008, S. 7812–7, doi:10.1128/JVI.00561-08, PMID 18524834, PMC 2519590 (freier Volltext) – (asm.org).
- ↑ a b van Hemert FJ, van de Klundert MA, Lukashov VV, Kootstra NA, Berkhout B, Zaaijer HL: Protein X of hepatitis B virus: origin and structure similarity with the central domain of DNA glycosylase. In: PLoS ONE. 6. Jahrgang, Nr. 8, 2011, S. e23392, doi:10.1371/journal.pone.0023392, PMID 21850270, PMC 3153941 (freier Volltext) – (plos.org). Referenzfehler: Ungültiges
<ref>-Tag. Der Name „van_Hemert2011“ wurde mehrere Male mit einem unterschiedlichen Inhalt definiert. - ↑ Lin B, Anderson DA: A vestigial X open reading frame in duck hepatitis B virus. In: Intervirology. 43. Jahrgang, Nr. 3, 2000, S. 185–90, PMID 11044813 (karger.com [PDF]).
- ↑ Osiowy C, Giles E, Tanaka Y, Mizokami M, Minuk GY: Molecular evolution of hepatitis B virus over 25 years. In: J. Virol. 80. Jahrgang, Nr. 21, November 2006, S. 10307–14, doi:10.1128/JVI.00996-06, PMID 17041211, PMC 1641782 (freier Volltext) – (asm.org).
- ↑ Zhou Y, Holmes EC (2007) Bayesian estimates of the evolutionary rate and age of hepatitis B virus. J Mol Evol 65(2):197-205
- ↑ Kay A, Zoulim F: Hepatitis B virus genetic variability and evolution. In: Virus Res. 127. Jahrgang, Nr. 2, 2007, S. 164–76, doi:10.1016/j.virusres.2007.02.021, PMID 17383765.
- ↑ a b Beck J, Nassal M: Hepatitis B virus replication. In: World J. Gastroenterol. 13. Jahrgang, Nr. 1, 2007, S. 48–64, PMID 17206754.
- ↑ Guo GH, Tan DM, Zhu PA, Liu F: Hepatitis B virus X protein promotes proliferation and upregulates TGF-beta1 and CTGF in human hepatic stellate cell line, LX-2. In: Hbpd Int. 8. Jahrgang, Nr. 1, Februar 2009, S. 59–64, PMID 19208517 (hbpdint.com).
- ↑ Bouchard MJ, Schneider RJ: The enigmatic X gene of hepatitis B virus. In: J. Virol. 78. Jahrgang, Nr. 23, 2004, S. 12725–34, doi:10.1128/JVI.78.23.12725-12734.2004, PMID 15542625, PMC 524990 (freier Volltext).
- ↑ 3rd Smith Gj, JE Donello, R Lück, G Steger, TJ Hope: The hepatitis B virus post-transcriptional regulatory element contains two conserved RNA stem-loops which are required for function. In: Nucleic Acids Research. 26. Jahrgang, Nr. 21, 1998, S. 4818–27, doi:10.1093/nar/26.21.4818, PMID 9776740, PMC 147918 (freier Volltext).
- ↑ Flodell S, Schleucher J, Cromsigt J, Ippel H, Kidd-Ljunggren K, Wijmenga S: The apical stem-loop of the hepatitis B virus encapsidation signal folds into a stable tri-loop with two underlying pyrimidine bulges. In: Nucleic Acids Res. 30. Jahrgang, Nr. 21, November 2002, S. 4803–11, doi:10.1093/nar/gkf603, PMID 12409471, PMC 135823 (freier Volltext) – (oxfordjournals.org).
- ↑ Olinger CM, Jutavijittum P, Hübschen JM, et al.: Possible new hepatitis B virus genotype, southeast Asia. In: Emerging Infect. Dis. 14. Jahrgang, Nr. 11, November 2008, S. 1777–80, doi:10.3201/eid1411.080437, PMID 18976569, PMC 2630741 (freier Volltext) – (cdc.gov).
- ↑ Kurbanov F, Tanaka Y, Kramvis A, Simmonds P, Mizokami M: When should "I" consider a new hepatitis B virus genotype? In: J. Virol. 82. Jahrgang, Nr. 16, August 2008, S. 8241–2, doi:10.1128/JVI.00793-08, PMID 18663008, PMC 2519592 (freier Volltext) – (asm.org).
- ↑ Palumbo E: Hepatitis B genotypes and response to antiviral therapy: a review. In: Am J Ther. 14. Jahrgang, Nr. 3, 2007, S. 306–9, doi:10.1097/01.pap.0000249927.67907.eb, PMID 17515708 (wkhealth.com).
- ↑ Mahtab MA, Rahman S, Khan M, Karim F: Hepatitis B virus genotypes: an overview. In: Hbpd Int. 7. Jahrgang, Nr. 5, Oktober 2008, S. 457–64, PMID 18842489 (hbpdint.com).
- ↑ Cavinta L, Sun J, May A, et al.: A new isolate of hepatitis B virus from the Philippines possibly representing a new subgenotype C6. In: J. Med. Virol. 81. Jahrgang, Nr. 6, Juni 2009, S. 983–7, doi:10.1002/jmv.21475, PMID 19382274.
- ↑ Lusida M.I., Nugrahaputra V.E., Soetjipto Handajani R., Nagano-Fujii M., Sasayama M., Utsumi T., Hotta H.: Novel subgenotypes of hepatitis B virus genotypes C and D in Papua, Indonesia. In: J. Clin. Microbiol. 46. Jahrgang, Nr. 7, 2008, S. 2160–2166, doi:10.1128/JCM.01681-07, PMID 18463220, PMC 2446895 (freier Volltext).
- ↑ Bruss V: Hepatitis B virus morphogenesis. In: World J. Gastroenterol. 13. Jahrgang, Nr. 1, 2007, S. 65–73, PMID 17206755.