„Cressdnaviricota“ – Versionsunterschied
| [gesichtete Version] | [gesichtete Version] |
Aka (Diskussion | Beiträge) K →Systematik: Halbgeviertstrich, Kleinkram |
K PMID-fix |
||
| Zeile 9: | Zeile 9: | ||
| Reich = [[Shotokuvirae]]<ref name="ICTV_MSL#35" /> |
| Reich = [[Shotokuvirae]]<ref name="ICTV_MSL#35" /> |
||
| Phylum = Cressdnaviricota |
| Phylum = Cressdnaviricota |
||
| Klasse = |
| Klasse = |
||
| Ordnung = |
| Ordnung = |
||
| Familie = |
| Familie = |
||
| Subfamilie = |
| Subfamilie = |
||
| Gattung = |
| Gattung = |
||
| Spezies = |
| Spezies = |
||
| Subspezies = |
| Subspezies = |
||
| Zeile 20: | Zeile 20: | ||
| Kapsid = |
| Kapsid = |
||
| Virushülle = |
| Virushülle = |
||
| NCBI_Tax = 2732416 |
| NCBI_Tax = 2732416 |
||
| NCBI_Ref = |
| NCBI_Ref = |
||
| ViralZone = |
| ViralZone = |
||
| ICTV = |
| ICTV = |
||
| Zeile 39: | Zeile 39: | ||
[[Datei:Geminiviruses drawing.png|mini|hochkant=0.9|Schemazeichnung: ''Geminiviridae''-[[Virion|Partikel]]]] |
[[Datei:Geminiviruses drawing.png|mini|hochkant=0.9|Schemazeichnung: ''Geminiviridae''-[[Virion|Partikel]]]] |
||
'''''Cressdnaviricota''''' (informell '''CRESS-DNA-Viren''') ist die Bezeichnung für ein [[Phylum]] von [[Viren]] im [[Reich (Biologie)|Reich]] ''[[Shotokuvirae]]'' [[:en:Shotokuvirae|<nowiki>[en]</nowiki>]], [[Realm (Virologie)|Realm]] ''[[Monodnaviria]]'', deren [[Genom]] aus Einzelstrang-[[DNA]] (ssDNA) besteht und deren natürliche [[Wirt (Biologie)|Wirte]] [[Eukaryoten]] (evtl. auch [[Archaeen]]) sind. |
'''''Cressdnaviricota''''' (informell '''CRESS-DNA-Viren''') ist die Bezeichnung für ein [[Phylum]] von [[Viren]] im [[Reich (Biologie)|Reich]] ''[[Shotokuvirae]]'' [[:en:Shotokuvirae|<nowiki>[en]</nowiki>]], [[Realm (Virologie)|Realm]] ''[[Monodnaviria]]'', deren [[Genom]] aus Einzelstrang-[[DNA]] (ssDNA) besteht und deren natürliche [[Wirt (Biologie)|Wirte]] [[Eukaryoten]] (evtl. auch [[Archaeen]]) sind. |
||
Sie [[Replikation|replizieren]] ihr Genom per [[Replikation#Rolling-Circle-Replikation|Rolling-Circle-Replikation]] ({{enS|rolling circle replication}} [[:en:rolling circle replication|<nowiki>[en]</nowiki>]], RCR), gestartet durch ein viruseigenes [[Protein]] mit der Bezeichnung [[#Genom|REP]].<ref name="ssdna_rcr" |
Sie [[Replikation|replizieren]] ihr Genom per [[Replikation#Rolling-Circle-Replikation|Rolling-Circle-Replikation]] ({{enS|rolling circle replication}} [[:en:rolling circle replication|<nowiki>[en]</nowiki>]], RCR), gestartet durch ein viruseigenes [[Protein]] mit der Bezeichnung [[#Genom|REP]].<ref name="ssdna_rcr">{{cite web|title=ssDNA Rolling circle|url=https://viralzone.expasy.org/1941|website=ViralZone|publisher=Swiss Institute of Bioinformatics|access-date=2021-03-29}}</ref><ref>{{cite web|url=https://talk.ictvonline.org/taxonomy/|title=Virus Taxonomy: 2019 Release|website=talk.ictvonline.org|publisher=International Committee on Taxonomy of Viruses|access-date=2020-04-25}}</ref> |
||
Wegen dieser Replikationsmethode stellen die ''Cressdnaviricota'' die „prototypischen“ Vertreter des Realms ''Monodnaviria'' dar. |
Wegen dieser Replikationsmethode stellen die ''Cressdnaviricota'' die „prototypischen“ Vertreter des Realms ''Monodnaviria'' dar. |
||
Mit der Einrichtung dieses Phylums hat das {{lang|en|[[International Committee on Taxonomy of Viruses]] (ICTV)}} die zuvor informell als Supergruppe der CRESS-DNA-Viren (en. ''{{lang|en|CRESS DNA viruses}}'') als offizielles [[Taxon]] anerkannt. Sie umspannen derzeit sieben [[Familie (Biologie)|Familien]].<ref name=Krupovic2019 /><ref name="proposal" /> |
Mit der Einrichtung dieses Phylums hat das {{lang|en|[[International Committee on Taxonomy of Viruses]] (ICTV)}} die zuvor informell als Supergruppe der CRESS-DNA-Viren (en. ''{{lang|en|CRESS DNA viruses}}'') als offizielles [[Taxon]] anerkannt. Sie umspannen derzeit sieben [[Familie (Biologie)|Familien]].<ref name="Krupovic2019" /><ref name="proposal" /> |
||
Die CRESS-DNA-Viren werden mit einer Vielzahl von Krankheiten in Verbindung gebracht. Darunter sind Krankheiten bei wirtschaftlich wichtigen Nutzpflanzen sowie eine Vielzahl von Krankheiten bei Tieren einschließlich des Menschen.<ref name="proposal" /> |
Die CRESS-DNA-Viren werden mit einer Vielzahl von Krankheiten in Verbindung gebracht. Darunter sind Krankheiten bei wirtschaftlich wichtigen Nutzpflanzen sowie eine Vielzahl von Krankheiten bei Tieren einschließlich des Menschen.<ref name="proposal" /> |
||
Die Bezeichnung ''Cressdnaviricota'' setzt sich zusammen aus dem Akronym CRESS ({{lang|en|circular REP-encoding single-stranded|de=zirkulär, REP-kodierend, einzelsträngig}}), DNA, und der Endung -viricota für Virusphyla.<ref name=Krupovic2019 /><ref name="ICTV_Prop">Mart Krupovic, Arvind Varsani, Jens H. Kuhn, D. Kazlauskas, M. Breitbart, E. Delwart, K. Rosario, N. Yutin, Y. I. Wolf, B. Harrach, F. M. Zerbini, V. V. Dolja, E. V. Koonin: [https://talk.ictvonline.org/ictv/proposals/2019.012D.zip Create 1 new phylum (Cressdnaviricota) including 2 classes and 6 orders for classification of CRESS-DNA viruses], Vorschlag 2019.012DA.v1.Cressdnaviricota (ZIP von docx und xlsx); April 2019</ref> Dieses ICTV-bestätigte Phylum von DNA-Viren mit zirkulärem '''Einzelstrang'''-DNA-Genom ist nicht zu verwechseln mit der vorgeschlagenen Ordnung „''[[Crassvirales|Cr'''a'''ssvirales]]''“ mit zirkuläem '''Doppelstrang'''-DNA-Genom, deren Wirte [[Bakterien]] sind. |
Die Bezeichnung ''Cressdnaviricota'' setzt sich zusammen aus dem Akronym CRESS ({{lang|en|circular REP-encoding single-stranded|de=zirkulär, REP-kodierend, einzelsträngig}}), DNA, und der Endung -viricota für Virusphyla.<ref name="Krupovic2019" /><ref name="ICTV_Prop">Mart Krupovic, Arvind Varsani, Jens H. Kuhn, D. Kazlauskas, M. Breitbart, E. Delwart, K. Rosario, N. Yutin, Y. I. Wolf, B. Harrach, F. M. Zerbini, V. V. Dolja, E. V. Koonin: [https://talk.ictvonline.org/ictv/proposals/2019.012D.zip Create 1 new phylum (Cressdnaviricota) including 2 classes and 6 orders for classification of CRESS-DNA viruses], Vorschlag 2019.012DA.v1.Cressdnaviricota (ZIP von docx und xlsx); April 2019</ref> Dieses ICTV-bestätigte Phylum von DNA-Viren mit zirkulärem '''Einzelstrang'''-DNA-Genom ist nicht zu verwechseln mit der vorgeschlagenen Ordnung „''[[Crassvirales|Cr'''a'''ssvirales]]''“ mit zirkuläem '''Doppelstrang'''-DNA-Genom, deren Wirte [[Bakterien]] sind. |
||
== Beschreibung == |
== Beschreibung == |
||
| Zeile 51: | Zeile 51: | ||
[[Datei:Circovirus genome.jpg|mini|hochkant=0.9|Genomkarte von der Spezies ''[[Porcines Circovirus-1]]'' (PCV-1), ''[[Circoviridae]]'']] |
[[Datei:Circovirus genome.jpg|mini|hochkant=0.9|Genomkarte von der Spezies ''[[Porcines Circovirus-1]]'' (PCV-1), ''[[Circoviridae]]'']] |
||
[[Datei:PCVmeltingpot.jpg|mini|hochkant=1.5|Die [[Haarnadelstruktur]] (''{{lang|en|stem-loop}}'') des Replikations­ursprungs: Replikations­start per ''{{lang|en|melting pot}}''-Mechanismus]] |
[[Datei:PCVmeltingpot.jpg|mini|hochkant=1.5|Die [[Haarnadelstruktur]] (''{{lang|en|stem-loop}}'') des Replikations­ursprungs: Replikations­start per ''{{lang|en|melting pot}}''-Mechanismus]] |
||
Das Genom der ''Cress­dna­viricota'' [[Genetischer Code|kodiert]] für zwei wesentliche Proteine:<ref name=Krupovic2019 /> |
Das Genom der ''Cress­dna­viricota'' [[Genetischer Code|kodiert]] für zwei wesentliche Proteine:<ref name="Krupovic2019" /> |
||
* das REP (oder Rep) – dieses startet (initiiert) die [[Replikation#Rolling-Circle-Replikation|Rolling-Circle-Replikation]] ({{enS|rolling circle replication}} [[:en:rolling circle replication|<nowiki>[en]</nowiki>]], RCR) des zirkulären Virus[[genom]]s. Sie ist im Phylum [[Konservierung#Evolution|konserviert]], d. h. wenig unterschiedlich, da sie sich im Lauf der Evolution nicht wesentlich verändert hat. Rep ist eine [[Endonuklease]] der [[HUH-Endonuklease|HUH-Superfamilie]] [[:en:HUH-tag|<nowiki>[en]</nowiki>]]. Endonukleasen sind [[Enzym]]e, die [[Phosphodiester]]­bindungen innerhalb einer [[Polynukleotid]]­kette spalten können. HUH- oder HuH-Endonukleasen sind spezielle Endonukleasen, die |
* das REP (oder Rep) – dieses startet (initiiert) die [[Replikation#Rolling-Circle-Replikation|Rolling-Circle-Replikation]] ({{enS|rolling circle replication}} [[:en:rolling circle replication|<nowiki>[en]</nowiki>]], RCR) des zirkulären Virus[[genom]]s. Sie ist im Phylum [[Konservierung#Evolution|konserviert]], d. h. wenig unterschiedlich, da sie sich im Lauf der Evolution nicht wesentlich verändert hat. Rep ist eine [[Endonuklease]] der [[HUH-Endonuklease|HUH-Superfamilie]] [[:en:HUH-tag|<nowiki>[en]</nowiki>]]. Endonukleasen sind [[Enzym]]e, die [[Phosphodiester]]­bindungen innerhalb einer [[Polynukleotid]]­kette spalten können. HUH- oder HuH-Endonukleasen sind spezielle Endonukleasen, die |
||
:* ein HUH-[[Sequenzmotiv|Motiv]] enthalten, das aus zwei [[Histidin|'''H'''istidinresten]] besteht, die durch einen sperrigen [[hydrophob]]en Rest getrennt sind, |
:* ein HUH-[[Sequenzmotiv|Motiv]] enthalten, das aus zwei [[Histidin|'''H'''istidinresten]] besteht, die durch einen sperrigen [[hydrophob]]en Rest getrennt sind, |
||
:* und ein Y-Motiv, das einen oder zwei [[Tyrosin]]reste enthält. |
:* und ein Y-Motiv, das einen oder zwei [[Tyrosin]]reste enthält. |
||
: HUH-Endonukleasen werden grob in zwei Kategorien von Enzymen eingeteilt: '''Re'''plikationsinitiations'''p'''roteine (Rep) und Relaxase-/Mobilisierungsproteine. Die HUH-Endonuklease der ssDNA-Viren gehört zur ersten Gruppe (Rep), weil die von ihr vermittelte Spaltung des (zirkularen) viralen Genoms die Replikation einleitet.<ref name=proposal /><ref name=chandler /> |
: HUH-Endonukleasen werden grob in zwei Kategorien von Enzymen eingeteilt: '''Re'''plikationsinitiations'''p'''roteine (Rep) und Relaxase-/Mobilisierungsproteine. Die HUH-Endonuklease der ssDNA-Viren gehört zur ersten Gruppe (Rep), weil die von ihr vermittelte Spaltung des (zirkularen) viralen Genoms die Replikation einleitet.<ref name="proposal" /><ref name="chandler" /> |
||
* das CP (auch Cp, CAP oder Cap) – dieses bezeichnet ein [[Kapsomer|Kapsidprotein]], also ein [[Protein]], das das [[Kapsid]] der [[Virion|Virusteilchen]] (Virionen) aufbaut. |
* das CP (auch Cp, CAP oder Cap) – dieses bezeichnet ein [[Kapsomer|Kapsidprotein]], also ein [[Protein]], das das [[Kapsid]] der [[Virion|Virusteilchen]] (Virionen) aufbaut. |
||
Im Gegensatz zu den REP-Proteinen sind die Kapsidproteine der CRESS-DNA-Viren nicht [[ortholog]], sondern wurden mehrfach unabhängig voneinander von [[RNA]]-Viren übernommen. Daher ist das Gen für das REP-Protein das (alleinige) Kriterium, um über die Zugehörigkeit zum Phylum Aufschluss zu geben.<ref name=Krupovic2019 /> |
Im Gegensatz zu den REP-Proteinen sind die Kapsidproteine der CRESS-DNA-Viren nicht [[ortholog]], sondern wurden mehrfach unabhängig voneinander von [[RNA]]-Viren übernommen. Daher ist das Gen für das REP-Protein das (alleinige) Kriterium, um über die Zugehörigkeit zum Phylum Aufschluss zu geben.<ref name="Krupovic2019" /> |
||
=== Wirte === |
=== Wirte === |
||
| Zeile 67: | Zeile 67: | ||
* [[Tiere]]n (''[[Circoviridae]]'') und |
* [[Tiere]]n (''[[Circoviridae]]'') und |
||
* [[Algen]] (''[[Bacilladnaviridae]]''). |
* [[Algen]] (''[[Bacilladnaviridae]]''). |
||
Mitglieder der beiden Familien ''[[Smacoviridae]]'' und ''[[Redondoviridae]]'' wurden durch [[Metagenomik]] entdeckt wurden infizieren vermutlich ebenfalls Tiere, obwohl auch eine Assoziation von Smacoviren mit methanogenen [[Archaeen]] vorgeschlagen wurde.<ref name=Krupovic2019 /> |
Mitglieder der beiden Familien ''[[Smacoviridae]]'' und ''[[Redondoviridae]]'' wurden durch [[Metagenomik]] entdeckt wurden infizieren vermutlich ebenfalls Tiere, obwohl auch eine Assoziation von Smacoviren mit methanogenen [[Archaeen]] vorgeschlagen wurde.<ref name="Krupovic2019" /> |
||
== Systematik == |
== Systematik == |
||
[[Datei:Viruses-10-00187-Figure-S1.png|mini|hochkant=1.5|Ungewurzelter [[Phylogenetischer Baum]] der CRESS-DNA-Viren nach Kazlauskas, Varsani und Krupovic (2018, Supplement).<ref name=Kazlauskas2018 /> Der nach rechts zeigende Ast entspricht den ''Geplafuvirales''.]] |
[[Datei:Viruses-10-00187-Figure-S1.png|mini|hochkant=1.5|Ungewurzelter [[Phylogenetischer Baum]] der CRESS-DNA-Viren nach Kazlauskas, Varsani und Krupovic (2018, Supplement).<ref name="Kazlauskas2018" /> Der nach rechts zeigende Ast entspricht den ''Geplafuvirales''.]] |
||
Die Systematik der CRESS-DNA-Viren ist nach ICTV (Master Species List #35) und Vorschlag 2021.010F folgende (Stand 1. April 2022):<ref name="ICTV_MSL#37"/><ref>Arvind Varsani, Mart Krupovic: [https://talk.ictvonline.org/ictv/proposals/2021.010F.R.Cressdna_2neword_3newfam_21newgen.zip Create two new orders, three new families and 21 new genera in the class ''Arfiviricetes'' (''Cressdnaviricota'')] (ZIP: pdf, xlsx). Proposal 021.010F, Oktober 2020. Siehe insbes. Fig. 1.</ref> |
Die Systematik der CRESS-DNA-Viren ist nach ICTV (Master Species List #35) und Vorschlag 2021.010F folgende (Stand 1. April 2022):<ref name="ICTV_MSL#37" /><ref>Arvind Varsani, Mart Krupovic: [https://talk.ictvonline.org/ictv/proposals/2021.010F.R.Cressdna_2neword_3newfam_21newgen.zip Create two new orders, three new families and 21 new genera in the class ''Arfiviricetes'' (''Cressdnaviricota'')] (ZIP: pdf, xlsx). Proposal 021.010F, Oktober 2020. Siehe insbes. Fig. 1.</ref> |
||
[[Realm (Virologie)|Realm]] ''[[Monodnaviria]]'' |
[[Realm (Virologie)|Realm]] ''[[Monodnaviria]]'' |
||
| Zeile 80: | Zeile 80: | ||
:::* Ordnung ''Cirlivirales'' – Familien ''[[Circoviridae]]'' und ''[[Vilyaviridae]]'' (letztere 12 Gattungen), sowie „CRESSV1“ (alias „CRESS1“) und „CRESSV3“ (alias „CRESS3“) |
:::* Ordnung ''Cirlivirales'' – Familien ''[[Circoviridae]]'' und ''[[Vilyaviridae]]'' (letztere 12 Gattungen), sowie „CRESSV1“ (alias „CRESS1“) und „CRESSV3“ (alias „CRESS3“) |
||
:::* Ordnung ''Cremevirales'' – Familie ''[[Smacoviridae]]'' |
:::* Ordnung ''Cremevirales'' – Familie ''[[Smacoviridae]]'' |
||
:::* Ordnung ''[[Mulpavirales]]'' – Familien ''[[Metaxyviridae]]''<!-- (mit Spezies ''Coconut foliar decay virus'', CFDV)--><ref name=Gronenborn2020>Bruno Gronenborn, Arvind Varsani, J. W. Randles, H. J. Vetten, J. E. Thomas: [https://talk.ictvonline.org/ictv/proposals/2020.001P.R.Petromoalphasatellitinae_nsf.zip Create a new subfamily (''Petromoalphasatellitinae'') with four new genera and six new species (''Alphasatellitidae'')] (ZIP: docx, xlsx), Vorschlag 2020.001P.R.Petromoalphasatellitinae_nsf and das ICTV (angenommen)</ref><ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=12474 Coconut foliar decay virus] (species)</ref> und ''[[Nanoviridae]]'', sowie „CRESSV4“ (alias „CRESS4“) und „CRESSV4“ (alias „CRESS4“) |
:::* Ordnung ''[[Mulpavirales]]'' – Familien ''[[Metaxyviridae]]''<!-- (mit Spezies ''Coconut foliar decay virus'', CFDV)--><ref name="Gronenborn2020">Bruno Gronenborn, Arvind Varsani, J. W. Randles, H. J. Vetten, J. E. Thomas: [https://talk.ictvonline.org/ictv/proposals/2020.001P.R.Petromoalphasatellitinae_nsf.zip Create a new subfamily (''Petromoalphasatellitinae'') with four new genera and six new species (''Alphasatellitidae'')] (ZIP: docx, xlsx), Vorschlag 2020.001P.R.Petromoalphasatellitinae_nsf and das ICTV (angenommen)</ref><ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=12474 Coconut foliar decay virus] (species)</ref> und ''[[Nanoviridae]]'', sowie „CRESSV4“ (alias „CRESS4“) und „CRESSV4“ (alias „CRESS4“) |
||
:::* Ordnung ''Recrevirales'' – Familie ''[[Redondoviridae]]'' |
:::* Ordnung ''Recrevirales'' – Familie ''[[Redondoviridae]]'' |
||
| Zeile 87: | Zeile 87: | ||
::* Klasse ''Repensiviricetes'' |
::* Klasse ''Repensiviricetes'' |
||
:::* Ordnung ''[[Geplafuvirales]]'' – Familien ''[[Geminiviridae]]'' und ''[[Genomoviridae]]'', evtl. auch ''[[Alphasatellitidae]]'' und Kandidaten-Gattung/Klade „''[[Geplafuvirales#Systematik|Nepavirus]]''“ |
:::* Ordnung ''[[Geplafuvirales]]'' – Familien ''[[Geminiviridae]]'' und ''[[Genomoviridae]]'', evtl. auch ''[[Alphasatellitidae]]'' und Kandidaten-Gattung/Klade „''[[Geplafuvirales#Systematik|Nepavirus]]''“ |
||
::* vorgeschlagene Familien (außer den oben genannten CRESSV1, CRESSV2, …):<ref |
::* vorgeschlagene Familien (außer den oben genannten CRESSV1, CRESSV2, …):<ref |
||
name=Kazlauskas2018>Darius Kazlauskas, Arvind Varsani, Mart Krupovic: [https://www.mdpi.com/1999-4915/10/4/187/htm Pervasive Chimerism in the Replication-Associated Proteins of Uncultured Single-Stranded DNA Viruses], in: MDPI Viruses, Band 10, Nr. 4, Special Issue Viral Recombination: Ecology, Evolution and Pathogenesis, 10. April 2018, S. 187; [[doi:10.3390/v10040187]], PMID 29642587; siehe insbes. Fig. S1 in [https://www.mdpi.com/1999-4915/10/4/187/s1 Supplement S1] (ZIP: pdf, xlsx)</ref><ref |
name=Kazlauskas2018>Darius Kazlauskas, Arvind Varsani, Mart Krupovic: [https://www.mdpi.com/1999-4915/10/4/187/htm Pervasive Chimerism in the Replication-Associated Proteins of Uncultured Single-Stranded DNA Viruses], in: MDPI Viruses, Band 10, Nr. 4, Special Issue Viral Recombination: Ecology, Evolution and Pathogenesis, 10. April 2018, S. 187; [[doi:10.3390/v10040187]], PMID 29642587; siehe insbes. Fig. S1 in [https://www.mdpi.com/1999-4915/10/4/187/s1 Supplement S1] (ZIP: pdf, xlsx)</ref><ref |
||
name=Kazlauskas2019>Darius Kazlauskas, Arvind Varsani, [[Eugene V. Koonin]], Mart Krupovic: [https://www.nature.com/articles/s41467-019-11433-0 Multiple origins of prokaryotic and eukaryotic single-stranded DNA viruses from bacterial and archaeal plasmids]. In: [[Nature]] Communicastions, Band 10, Nr. 3425, 31. Juli 2019, [[doi:10.1038/s41467-019-11433-0]], PMID |
name="Kazlauskas2019">Darius Kazlauskas, Arvind Varsani, [[Eugene V. Koonin]], Mart Krupovic: [https://www.nature.com/articles/s41467-019-11433-0 Multiple origins of prokaryotic and eukaryotic single-stranded DNA viruses from bacterial and archaeal plasmids]. In: [[Nature]] Communicastions, Band 10, Nr. 3425, 31. Juli 2019, [[doi:10.1038/s41467-019-11433-0]], PMID 31366885.</ref><ref |
||
name=Zhao2019>Lele Zhao, Karyna Rosario, Mya Breitbart, Siobain Duffy: [https://www.sciencedirect.com/science/article/pii/S0065352718300575 Chapter Three - Eukaryotic Circular Rep-Encoding Single-Stranded DNA (CRESS DNA) Viruses: Ubiquitous Viruses With Small Genomes and a Diverse Host Range], in: Advances in Virus Research, Band 103, 2019, S. 71–133, [[doi:10.1016/bs.aivir.2018.10.001]], Epub 5. Dezember 2018. Insbes. siehe [https://ars.els-cdn.com/content/image/1-s2.0-S0065352718300575-f03-04-9780128177228.jpg Fig. 3]</ref><ref |
name=Zhao2019>Lele Zhao, Karyna Rosario, Mya Breitbart, Siobain Duffy: [https://www.sciencedirect.com/science/article/pii/S0065352718300575 Chapter Three - Eukaryotic Circular Rep-Encoding Single-Stranded DNA (CRESS DNA) Viruses: Ubiquitous Viruses With Small Genomes and a Diverse Host Range], in: Advances in Virus Research, Band 103, 2019, S. 71–133, [[doi:10.1016/bs.aivir.2018.10.001]], Epub 5. Dezember 2018. Insbes. siehe [https://ars.els-cdn.com/content/image/1-s2.0-S0065352718300575-f03-04-9780128177228.jpg Fig. 3]</ref><ref |
||
name=Kaneko2020>Hiroto Kaneko, Morgan Gaia, Rodrigo Hernández-Velázquez, [[Patrick Forterre]], Curtis A. Suttle, Hiroyuki Ogata ''et al.'': [https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7811142/ Eukaryotic virus composition can predict the efficiency of carbon export in the global ocean], in: iScience, Band 24, Nr. 1, 22. Januar 2021, 102002, Epub 29. Dezember 2020, [[doi: |
name=Kaneko2020>Hiroto Kaneko, Morgan Gaia, Rodrigo Hernández-Velázquez, [[Patrick Forterre]], Curtis A. Suttle, Hiroyuki Ogata ''et al.'': [https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7811142/ Eukaryotic virus composition can predict the efficiency of carbon export in the global ocean], in: iScience, Band 24, Nr. 1, 22. Januar 2021, 102002, Epub 29. Dezember 2020, [[doi:10.1016/j.isci.2020.102002]], {{PMC|7811142}}, PMID 33490910; siehe [https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7811142/figure/fig1/?report=objectonly Fig. 1]<nowiki />(C)</ref> |
||
:::* Familie „CRESS1“ (alias „CRESSV1“) – zu ''Cirlivirales'', d. h. nahe ''Circoviridae'' |
:::* Familie „CRESS1“ (alias „CRESSV1“) – zu ''Cirlivirales'', d. h. nahe ''Circoviridae'' |
||
:::* Familie „CRESS1-Rec1“ – nahe ''Bacilladnaviridae'' oder ''Circoviridae'' |
:::* Familie „CRESS1-Rec1“ – nahe ''Bacilladnaviridae'' oder ''Circoviridae'' |
||
:::* Familie „CRESS2“ (alias „CRESSV2“) – zu ''Rohanvirales'' |
:::* Familie „CRESS2“ (alias „CRESSV2“) – zu ''Rohanvirales'' |
||
::::::* {{Anker|Circularisvirus}}Gattung(?) „''Circularisvirus''“<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=2502017&lvl=3&srchmode=3 Circularisvirus] (clade)</ref> (zusammen mit DfCirV zu „CRESS2“) |
::::::* {{Anker|Circularisvirus}}Gattung(?) „''Circularisvirus''“<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=2502017&lvl=3&srchmode=3 Circularisvirus] (clade)</ref> (zusammen mit DfCirV zu „CRESS2“) |
||
:::::::* Spezies: „''[[Anisoptera|Dragonfly]] circularisvirus''“ (DfCirV)<ref name=Rosario2012Tbl2>Karyna Rosario, Anisha Dayaram, Milen Marinov, Jessica Ware, Simona Kraberger, Daisy Stainton, Mya Breitbart, Arvind Varsani: [https://www.microbiologyresearch.org/content/journal/jgv/10.1099/vir.0.045948-0 Diverse circular ssDNA viruses discovered in dragonflies (Odonata: Epiprocta)], in: Journal of General Virology Band 93, Nr. 12, 1. Dezember 2012, [[doi:10.1099/vir.0.045948-0]]. Hier insbes. [https://www.microbiologyresearch.org/content/jgv/10.1099/vir.0.045948-0.t2?fmt=ahah&fullscreen=true Tbl. 2]</ref> (zu „CRESS2“<ref name=Kazlauskas2018xls>Darius Kazlauskas, Arvind Varsani, Mart Krupovic: [https://www.mdpi.com/1999-4915/10/4/187/htm Pervasive Chimerism in the Replication-Associated Proteins of Uncultured Single-Stranded DNA Viruses], in: MDPI Viruses, Band 10, Nr. 4, Special Issue Viral Recombination: Ecology, Evolution and Pathogenesis, 10. April 2018, 187, [[doi:10.3390/v10040187]]; siehe Excel-Datei in [https://www.mdpi.com/1999-4915/10/4/187/s1 Supplement S1] (ZIP: pdf, xlsx)</ref>) |
:::::::* Spezies: „''[[Anisoptera|Dragonfly]] circularisvirus''“ (DfCirV)<ref name="Rosario2012Tbl2">Karyna Rosario, Anisha Dayaram, Milen Marinov, Jessica Ware, Simona Kraberger, Daisy Stainton, Mya Breitbart, Arvind Varsani: [https://www.microbiologyresearch.org/content/journal/jgv/10.1099/vir.0.045948-0 Diverse circular ssDNA viruses discovered in dragonflies (Odonata: Epiprocta)], in: Journal of General Virology Band 93, Nr. 12, 1. Dezember 2012, [[doi:10.1099/vir.0.045948-0]]. Hier insbes. [https://www.microbiologyresearch.org/content/jgv/10.1099/vir.0.045948-0.t2?fmt=ahah&fullscreen=true Tbl. 2]</ref> (zu „CRESS2“<ref name="Kazlauskas2018xls">Darius Kazlauskas, Arvind Varsani, Mart Krupovic: [https://www.mdpi.com/1999-4915/10/4/187/htm Pervasive Chimerism in the Replication-Associated Proteins of Uncultured Single-Stranded DNA Viruses], in: MDPI Viruses, Band 10, Nr. 4, Special Issue Viral Recombination: Ecology, Evolution and Pathogenesis, 10. April 2018, 187, [[doi:10.3390/v10040187]]; siehe Excel-Datei in [https://www.mdpi.com/1999-4915/10/4/187/s1 Supplement S1] (ZIP: pdf, xlsx)</ref>) |
||
:::::::* Spezies: „''[[Entelegynae#Systematik|Cybaeus]] spider associated circular virus 1''“ (CySACV-1), in Spinnen der Gattung ''Cybaeus''<ref name=Rosario2017>Karyna Rosario, Kaitlin Mettel, Bayleigh Benner, Ryan Johnson, Catherine Scott, Sohath Yusseff-Vanegas, Christopher Baker, Deby Cassill, Caroline Storer, Arvind Varsani, Mya Breitbart: [https://scholarcommons.usf.edu/cgi/viewcontent.cgi?article=1671&context=msc_facpub Virus Discovery in All Three Major Lineages of Terrestrial Arthropods Highlights the Diversity of Single-stranded DNA Viruses Associated with Invertebrates], University of South Florida, College of Marine Science: Scholar Commons — Marine Science Faculty Publications, 2017, [https://scholarcommons.usf.edu/msc_facpub/652 652]. Part of the Life Sciences Commons. 11. Oktober 2018 [https://peerj.com/articles/5761/ PeerJ], PMID 30324030, {{PMC|6186406}}, [[doi:10.7717/peerj.5761]]</ref> |
:::::::* Spezies: „''[[Entelegynae#Systematik|Cybaeus]] spider associated circular virus 1''“ (CySACV-1), in Spinnen der Gattung ''Cybaeus''<ref name="Rosario2017">Karyna Rosario, Kaitlin Mettel, Bayleigh Benner, Ryan Johnson, Catherine Scott, Sohath Yusseff-Vanegas, Christopher Baker, Deby Cassill, Caroline Storer, Arvind Varsani, Mya Breitbart: [https://scholarcommons.usf.edu/cgi/viewcontent.cgi?article=1671&context=msc_facpub Virus Discovery in All Three Major Lineages of Terrestrial Arthropods Highlights the Diversity of Single-stranded DNA Viruses Associated with Invertebrates], University of South Florida, College of Marine Science: Scholar Commons — Marine Science Faculty Publications, 2017, [https://scholarcommons.usf.edu/msc_facpub/652 652]. Part of the Life Sciences Commons. 11. Oktober 2018 [https://peerj.com/articles/5761/ PeerJ], PMID 30324030, {{PMC|6186406}}, [[doi:10.7717/peerj.5761]]</ref> |
||
:::::::* Spezies: „''[[Seidenspinnen (Gattung)|Golden silk orbweaver]] associated circular virus 1''“ (GoSOrbACV-1), in Spinnen der Gattung ''Nephila''<ref name=Rosario2017 /> |
:::::::* Spezies: „''[[Seidenspinnen (Gattung)|Golden silk orbweaver]] associated circular virus 1''“ (GoSOrbACV-1), in Spinnen der Gattung ''Nephila''<ref name="Rosario2017" /> |
||
:::::::* Spezies: „''[[Streckerspinnen|Longjawed orbweaver]] circular virus 1''“ (LjOrbCV-1), in Spinnen der Gattung ''Leucauge''<ref name=Rosario2017 /> |
:::::::* Spezies: „''[[Streckerspinnen|Longjawed orbweaver]] circular virus 1''“ (LjOrbCV-1), in Spinnen der Gattung ''Leucauge''<ref name="Rosario2017" /> |
||
:::::::* Spezies: „''[[Gasteracantha cancriformis|Spinybacked orbweaver]] circular virus 1''“ (SpOrbCV-1), in Spinnen der Gattung ''Gasteracantha''<ref name=Rosario2017 /> |
:::::::* Spezies: „''[[Gasteracantha cancriformis|Spinybacked orbweaver]] circular virus 1''“ (SpOrbCV-1), in Spinnen der Gattung ''Gasteracantha''<ref name="Rosario2017" /> |
||
:::* Familie „CRESS3“ (alias „CRESSV3“) – zu ''Cirlivirales'' |
:::* Familie „CRESS3“ (alias „CRESSV3“) – zu ''Cirlivirales'' |
||
:::::::* {{Anker|Cyclicusvirus}}Spezies: „''Dragonfly cyclicusvirus''“ (DfCyclV)<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1234878 Dragonfly cyclicusvirus] (species)</ref><ref name=Rosario2012Tbl2 /> (zu „CRESS3“<ref name=Kazlauskas2018xls />) |
:::::::* {{Anker|Cyclicusvirus}}Spezies: „''Dragonfly cyclicusvirus''“ (DfCyclV)<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1234878 Dragonfly cyclicusvirus] (species)</ref><ref name="Rosario2012Tbl2" /> (zu „CRESS3“<ref name="Kazlauskas2018xls" />) |
||
:::* Familien „CRESS4“ (alias „CRESSV4“) und „CRESS5“ (alias „CRESSV5“) – zu ''Mulpavirales'', d. h. nahe ''Nanoviridae'' |
:::* Familien „CRESS4“ (alias „CRESSV4“) und „CRESS5“ (alias „CRESSV5“) – zu ''Mulpavirales'', d. h. nahe ''Nanoviridae'' |
||
:::* Familien oder [[Klade]]n „CRESS6“ (alias „CRESSV6“<ref name="ICTV_Prop" />) und „CRESS-Rec2“ – nahe ''Geplafuvirales'' |
:::* Familien oder [[Klade]]n „CRESS6“ (alias „CRESSV6“<ref name="ICTV_Prop" />) und „CRESS-Rec2“ – nahe ''Geplafuvirales'' |
||
:::* Familie oder Klade „{{lang|en|GasCSV-like viruses}}“ mit Spezies „''Gastropod associated circular ssDNA virus''“ (GasCSV)<ref>Anisha Dayaram, Sharyn Goldstien, Peyman Zawar-Reza, Christopher Gomez, Jon S. Harding, Arvind Varsani: [https://pubmed.ncbi.nlm.nih.gov/23364192/ Novel ssDNA virus recovered from estuarine Mollusc (Amphibola crenata) whose replication associated protein (Rep) shares similarities with Rep-like sequences of bacterial origin]. In: J. Gen. Virol., Band 94, Nr. Pt 5, Mai 2013, S. 1104–1110; [[doi:10.1099/vir.0.050088-0]], PMID 23364192, Epub 30. Januar 2013.</ref><ref>NCBI Taxonomy Browser: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1296573 Gastropod associated circular ssDNA virus] (species; acronym: GaCSV)</ref> |
:::* Familie oder Klade „{{lang|en|GasCSV-like viruses}}“ mit Spezies „''Gastropod associated circular ssDNA virus''“ (GasCSV)<ref>Anisha Dayaram, Sharyn Goldstien, Peyman Zawar-Reza, Christopher Gomez, Jon S. Harding, Arvind Varsani: [https://pubmed.ncbi.nlm.nih.gov/23364192/ Novel ssDNA virus recovered from estuarine Mollusc (Amphibola crenata) whose replication associated protein (Rep) shares similarities with Rep-like sequences of bacterial origin]. In: J. Gen. Virol., Band 94, Nr. Pt 5, Mai 2013, S. 1104–1110; [[doi:10.1099/vir.0.050088-0]], PMID 23364192, Epub 30. Januar 2013.</ref><ref>NCBI Taxonomy Browser: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1296573 Gastropod associated circular ssDNA virus] (species; acronym: GaCSV)</ref> |
||
:::* Familie „''[[Cruciviridae]]''“<ref name="NCBI_Cruci">NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=2058757 Cruciviridae] (family)</ref><ref name=HigueraNacho /> ohne nähere Zuordnung, mit „''[[Boiling Springs Lake RNA-DNA hybrid virus]]''“ (BSL-RDHV) und „''[[Idotea-Virus IWaV278]]''“<ref>Geoffrey S. Diemer, Kenneth M. Stedman:[https://biologydirect.biomedcentral.com/articles/10.1186/1745-6150-7-13 A novel virus genome discovered in an extreme environment suggests recombination between unrelated groups of RNA and DNA viruses]. In: Biology Direct, Band 7, Nr. 13, 11. Juni 2012; [[10.1186/1745-6150-7-13]], PMID 22515485, {{PMC|3372434}}.</ref> |
:::* Familie „''[[Cruciviridae]]''“<ref name="NCBI_Cruci">NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=2058757 Cruciviridae] (family)</ref><ref name="HigueraNacho" /> ohne nähere Zuordnung, mit „''[[Boiling Springs Lake RNA-DNA hybrid virus]]''“ (BSL-RDHV) und „''[[Idotea-Virus IWaV278]]''“<ref>Geoffrey S. Diemer, Kenneth M. Stedman:[https://biologydirect.biomedcentral.com/articles/10.1186/1745-6150-7-13 A novel virus genome discovered in an extreme environment suggests recombination between unrelated groups of RNA and DNA viruses]. In: Biology Direct, Band 7, Nr. 13, 11. Juni 2012; [[10.1186/1745-6150-7-13]], PMID 22515485, {{PMC|3372434}}.</ref> |
||
::::::* {{Anker|Volvovirus}}Gattung oder Klade „''Volvovirus''“<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=2502017&lvl=3&srchmode=3 Volvovirus] (clade)</ref> |
::::::* {{Anker|Volvovirus}}Gattung oder Klade „''Volvovirus''“<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=2502017&lvl=3&srchmode=3 Volvovirus] (clade)</ref> |
||
:::::::* Spezies: „''[[Heimchen|Acheta domesticus]] volvovirus''“ (AdVVV), in [[Heimchen]]<ref>Hanh T. Pham, Max Bergoin, Peter Tijssen: [https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3623006/ ''Acheta domesticus'' Volvovirus, a Novel Single-Stranded Circular DNA Virus of the House Cricket], in: Genome Announc. 1(2), März-April 2013, e00079-13. Epub 14. März 2013, [[doi: |
:::::::* Spezies: „''[[Heimchen|Acheta domesticus]] volvovirus''“ (AdVVV), in [[Heimchen]]<ref>Hanh T. Pham, Max Bergoin, Peter Tijssen: [https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3623006/ ''Acheta domesticus'' Volvovirus, a Novel Single-Stranded Circular DNA Virus of the House Cricket], in: Genome Announc. 1(2), März-April 2013, e00079-13. Epub 14. März 2013, [[doi:10.1128/genomeA.00079-13]], {{PMC|3623006}}, PMID 23516206</ref><ref>Hanh T. Pham, Hajime Iwao, Max Bergoin, Peter Tijssen: [https://journals.asm.org/doi/full/10.1128/genomeA.00328-13 New Volvovirus Isolates from ''Acheta domesticus'' (Japan) and ''Gryllus assimilis'' (United States)], in: ASM Journals: Genome Announcements, Band 1, Nr. 3, 6. Juni 2013, [[doi:10.1128/genomeA.00328-13]]</ref> |
||
:::::::* Spezies: „''[[Langfühlerschrecken#Grillen – Grylloidea|Cricket]] associated circular virus 1''“ (CrACV-1), in [[Langfühlerschrecken#Grillen – Grylloidea|Feldgrillen]]<ref name=Rosario2017 /> (zu „nicht-klassifizierten CRESS“<ref name=Kazlauskas2018xls />) |
:::::::* Spezies: „''[[Langfühlerschrecken#Grillen – Grylloidea|Cricket]] associated circular virus 1''“ (CrACV-1), in [[Langfühlerschrecken#Grillen – Grylloidea|Feldgrillen]]<ref name="Rosario2017" /> (zu „nicht-klassifizierten CRESS“<ref name="Kazlauskas2018xls" />) |
||
::* „{{lang|en|bacteria-tropic branch of the CRESS virus supergroup}}“ (aus [[Metagenomik]])<ref name=Tisza2020 /><!-- Zitat Tisza2020: ctbb30 and ctbc27 (may) represent a previously undescribed bacteria-tropic branch of the CRESS virus supergroup. |
::* „{{lang|en|bacteria-tropic branch of the CRESS virus supergroup}}“ (aus [[Metagenomik]])<ref name="Tisza2020" /><!-- Zitat Tisza2020: ctbb30 and ctbc27 (may) represent a previously undescribed bacteria-tropic branch of the CRESS virus supergroup. |
||
-- Eventuelle Zugehörigkeit zu einer der CRESS-Familien muss noch geklärt werden--> |
-- Eventuelle Zugehörigkeit zu einer der CRESS-Familien muss noch geklärt werden--> |
||
:::::::* [[Contig]] „ctbb30“ |
:::::::* [[Contig]] „ctbb30“ |
||
:::::::* Contig „ctbc27“ |
:::::::* Contig „ctbc27“ |
||
::* keinem höheren Rang zugeordnete vorgeschlagene Spezies: |
::* keinem höheren Rang zugeordnete vorgeschlagene Spezies: |
||
:::::::* Spezies: „''Dragonfly orbiculatusvirus''“ (DfOrV)<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1234874 Dragonfly orbiculatusvirus] (species)</ref><ref name=Rosario2012Tbl2 /> (zu „nicht-klassifizierten CRESS“<ref name=Kazlauskas2018xls />) |
:::::::* Spezies: „''Dragonfly orbiculatusvirus''“ (DfOrV)<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1234874 Dragonfly orbiculatusvirus] (species)</ref><ref name="Rosario2012Tbl2" /> (zu „nicht-klassifizierten CRESS“<ref name="Kazlauskas2018xls" />) |
||
== RNA-DNA-Hybridviren == |
== RNA-DNA-Hybridviren == |
||
Mehrere der bis heute entdeckten CRESS-DNA-Viren haben ein Genom, das signifikant größer ist, als das bei diesem Phylum normalerweise anzutreffen ist. |
Mehrere der bis heute entdeckten CRESS-DNA-Viren haben ein Genom, das signifikant größer ist, als das bei diesem Phylum normalerweise anzutreffen ist. |
||
Denkbar wäre, dass dies das Ergebnis von Duplikationsereignissen ist. |
Denkbar wäre, dass dies das Ergebnis von Duplikationsereignissen ist. |
||
Darüber hinaus wurde eine Akquisition von Kapsiden mit einer Symmetrie [[Triangulationszahl|T=3]] von den ssRNA-Familien ''[[Nodaviridae]]'' [[:en:Nodaviridae|<nowiki>[en]</nowiki>]] und ''[[Tombusviridae]]'' als Ursprung der Familie ''[[Bacilladnaviridae]]''<ref name=Kazlauskas2017 /> beziehungsweise der vorgeschlagenen Familie „''[[Cruciviridae]]''“<ref name=Steel2016 /><ref name=Dayaram2016 /> vermutet. Offenbar gab es noch weitere Rekombinationsereignisse von ssRNA-Viren oder ssDNA-[[Bakteriophagen]] mit CRESS-DNA-Viren.<ref name=Tisza2020 /> |
Darüber hinaus wurde eine Akquisition von Kapsiden mit einer Symmetrie [[Triangulationszahl|T=3]] von den ssRNA-Familien ''[[Nodaviridae]]'' [[:en:Nodaviridae|<nowiki>[en]</nowiki>]] und ''[[Tombusviridae]]'' als Ursprung der Familie ''[[Bacilladnaviridae]]''<ref name="Kazlauskas2017" /> beziehungsweise der vorgeschlagenen Familie „''[[Cruciviridae]]''“<ref name="Steel2016" /><ref name="Dayaram2016" /> vermutet. Offenbar gab es noch weitere Rekombinationsereignisse von ssRNA-Viren oder ssDNA-[[Bakteriophagen]] mit CRESS-DNA-Viren.<ref name="Tisza2020" /> |
||
Bei seit 2001 durchgeführten [[Metagenomik|Metagenomanalysen]] in den [[Geothermalgebiet]]en des [[Lassen-Volcanic-Nationalpark]]s (''{{lang|en|[[:en:Geothermal areas in Lassen Volcanic National Park|Geothermal areas in Lassen Volcanic National Park]]}}'', {{enS}}) in [[Nordkalifornien]] wurde eine ssDNA-Genomsequenz ([[Contig]]) gefunden, in der sich einerseits in einem mutmaßlichen REP-Gen [[Homologie (Genetik)|Homologien]] zu dem von ''[[Porcines Circovirus-2]]'' (PCV-2), Fam. ''Circoviridae'', zeigten. Andererseits gab es in einem mutmaßlichen Kapsidprotein-Gen Homologien zu denen der Familie ''[[Tombusviridae]]'' (sowie den vorgeschlagenen Spezies „''[[Peronosporaceae#Systematik|Sclerophthora]] macrospora virus A''“, SmV-A<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=191289 Sclerophthora macrospora virus A] (species)</ref> und |
Bei seit 2001 durchgeführten [[Metagenomik|Metagenomanalysen]] in den [[Geothermalgebiet]]en des [[Lassen-Volcanic-Nationalpark]]s (''{{lang|en|[[:en:Geothermal areas in Lassen Volcanic National Park|Geothermal areas in Lassen Volcanic National Park]]}}'', {{enS}}) in [[Nordkalifornien]] wurde eine ssDNA-Genomsequenz ([[Contig]]) gefunden, in der sich einerseits in einem mutmaßlichen REP-Gen [[Homologie (Genetik)|Homologien]] zu dem von ''[[Porcines Circovirus-2]]'' (PCV-2), Fam. ''Circoviridae'', zeigten. Andererseits gab es in einem mutmaßlichen Kapsidprotein-Gen Homologien zu denen der Familie ''[[Tombusviridae]]'' (sowie den vorgeschlagenen Spezies „''[[Peronosporaceae#Systematik|Sclerophthora]] macrospora virus A''“, SmV-A<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=191289 Sclerophthora macrospora virus A] (species)</ref> und |
||
„''[[Peronosporaceae#Systematik|Plasmopara]] halstedii virus A''“<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=468928 Plasmopara halstedii virus A] (species)</ref> – beide infizieren [[Eipilze]] der Familie [[Peronosporaceae]].<ref name=HigueraNacho /><ref name=Roux2013 /> |
„''[[Peronosporaceae#Systematik|Plasmopara]] halstedii virus A''“<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=468928 Plasmopara halstedii virus A] (species)</ref> – beide infizieren [[Eipilze]] der Familie [[Peronosporaceae]].<ref name="HigueraNacho" /><ref name="Roux2013" /> |
||
Diese gefundenen chimären Contigs wurde einem neuen Virus zugeordnet, für das nach dem dortigen Fundort ({{lang|en|Boiling Springs Lake}} {{Coordinate|NS=40.435528|EW=-121.397093|type=waterbody|region=US-CA|text=DEC|name=Boiling Springs Lake}}) die Bezeichnung „''[[Boiling Springs Lake RNA-DNA Hybrid Virus]]''“ (BSL-RDHV) vorgeschlagen wurde.<ref name=Diemer2012 /><ref name=HigueraNacho /><ref name=Harth2020 /> |
Diese gefundenen chimären Contigs wurde einem neuen Virus zugeordnet, für das nach dem dortigen Fundort ({{lang|en|Boiling Springs Lake}} {{Coordinate|NS=40.435528|EW=-121.397093|type=waterbody|region=US-CA|text=DEC|name=Boiling Springs Lake}}) die Bezeichnung „''[[Boiling Springs Lake RNA-DNA Hybrid Virus]]''“ (BSL-RDHV) vorgeschlagen wurde.<ref name="Diemer2012" /><ref name="HigueraNacho" /><ref name="Harth2020" /> |
||
Weitere Metagenomanalysen an verschiedenen Orten lieferten immer mehr an hybriden Contigs. Insgesamt sind von diesen mit Stand Oktober 2020 nicht wenige als 461 Kandidaten bekannt.<ref name=HigueraNacho /><ref name=Harth2020 /> |
Weitere Metagenomanalysen an verschiedenen Orten lieferten immer mehr an hybriden Contigs. Insgesamt sind von diesen mit Stand Oktober 2020 nicht wenige als 461 Kandidaten bekannt.<ref name="HigueraNacho" /><ref name="Harth2020" /> |
||
Zunächst schienen alle im CAP dem BSL-RDHV homolog zu sein; das REP-Gen scheint dagegen mehrmals von anderen CRESS-DNA-Familien (''Circoviridae'', ''Nanoviridae'', ''Geminiviridae'') per [[Horizontaler Gentransfer|horizontalem Gentransfer]] (HGT) übernommen worden zu sein.<ref name=Roux2013 /> |
Zunächst schienen alle im CAP dem BSL-RDHV homolog zu sein; das REP-Gen scheint dagegen mehrmals von anderen CRESS-DNA-Familien (''Circoviridae'', ''Nanoviridae'', ''Geminiviridae'') per [[Horizontaler Gentransfer|horizontalem Gentransfer]] (HGT) übernommen worden zu sein.<ref name="Roux2013" /> |
||
Im Laufe der Zeit fanden sich jedoch auch RNA-DNA-Hybridviren (RDHVs), die augenscheinlich nicht der Verwandtschaft von BSL-RDHV angehören: Ein bei Untersuchung der Vielfalt von mit Libellen assoziierten ssDNA-Viren gefundenes Contig mit der Bezeichnung DfCyclV („''Dragonfly cyclicusvirus''“)<ref name="NCBI_DfCyclV" /> zeigte eine schwache, aber signifikante Homologie eines mutmaßlichen Proteins zum CAP des ''[[Tabak-Mosaik-Satelliten-Virus|Satelliten-Tabaknekrosevirus]]'' (en. ''{{lang|en|Satellite tobacco necrosis virus}}'', STNV oder STMV).<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=12881 Satellite tobacco mosaic virus, equivalent Satellite tobacco necrosis virus] (no rank), Gattung ''[[Virtovirus]]'' vs. NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?name=Satellite+tobacco+necrosis+virus+?*&srchmode=2 Satellite tobacco necrosis virus ?*] (list), Gattung ''[[Albetovirus]]'' [[:en:Albetovirus|<nowiki>[en]</nowiki>]] |
Im Laufe der Zeit fanden sich jedoch auch RNA-DNA-Hybridviren (RDHVs), die augenscheinlich nicht der Verwandtschaft von BSL-RDHV angehören: Ein bei Untersuchung der Vielfalt von mit Libellen assoziierten ssDNA-Viren gefundenes Contig mit der Bezeichnung DfCyclV („''Dragonfly cyclicusvirus''“)<ref name="NCBI_DfCyclV" /> zeigte eine schwache, aber signifikante Homologie eines mutmaßlichen Proteins zum CAP des ''[[Tabak-Mosaik-Satelliten-Virus|Satelliten-Tabaknekrosevirus]]'' (en. ''{{lang|en|Satellite tobacco necrosis virus}}'', STNV oder STMV).<ref>NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=12881 Satellite tobacco mosaic virus, equivalent Satellite tobacco necrosis virus] (no rank), Gattung ''[[Virtovirus]]'' vs. NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?name=Satellite+tobacco+necrosis+virus+?*&srchmode=2 Satellite tobacco necrosis virus ?*] (list), Gattung ''[[Albetovirus]]'' [[:en:Albetovirus|<nowiki>[en]</nowiki>]]</ref><ref name="Rosario2012" /> |
||
</ref><ref name=Rosario2012 /> |
|||
Das CAP des Satelliten-Tabaknekrosevirus unterscheidet sich jedoch in Sequenz und Struktur grundlegend von denen der ''Tombusviridae'' und ähnelt am ehesten den CAPs der ''[[Geminiviridae]]''. |
Das CAP des Satelliten-Tabaknekrosevirus unterscheidet sich jedoch in Sequenz und Struktur grundlegend von denen der ''Tombusviridae'' und ähnelt am ehesten den CAPs der ''[[Geminiviridae]]''. |
||
Daher ist vermutlich DfCyclV kein Mitglied der [[Klade]] der BSL-RDHV-ähnlichen Viren.<ref name=Roux2013 /> |
Daher ist vermutlich DfCyclV kein Mitglied der [[Klade]] der BSL-RDHV-ähnlichen Viren.<ref name="Roux2013" /> |
||
Über den Fund von RDHVs weiteren Typs berichteten zudem Koonin und Dolja (2012);<ref name=Koonin2012 /> |
Über den Fund von RDHVs weiteren Typs berichteten zudem Koonin und Dolja (2012);<ref name="Koonin2012" /> |
||
und die ssDNA-Familie ''[[Bidnaviridae]]'' <!--[[:en:Bidnaviridae|<nowiki>[en]</nowiki>]] -- obsolet -->hat offenbar auch eine „turbulente“ evolutionäre Vergangenheit u. a. mit einer mutmaßlichen Gen-Übernahme von den dsRNA-Viren der ''[[Reoviridae]]''<ref name=Krupovic2014 /> |
und die ssDNA-Familie ''[[Bidnaviridae]]'' <!--[[:en:Bidnaviridae|<nowiki>[en]</nowiki>]] -- obsolet -->hat offenbar auch eine „turbulente“ evolutionäre Vergangenheit u. a. mit einer mutmaßlichen Gen-Übernahme von den dsRNA-Viren der ''[[Reoviridae]]''<ref name="Krupovic2014" /> |
||
Die Entdeckung weiterer Contigs (Genome aus Metagenomanalysen), die Ähnlichkeiten sowohl mit DNA- als auch mit RNA-Viren aufweisen, deutet darauf hin, dass RDHVs vermutlich weiter verbreitet sind als anfangs angenommen.<ref name=Rosario2012 /> |
Die Entdeckung weiterer Contigs (Genome aus Metagenomanalysen), die Ähnlichkeiten sowohl mit DNA- als auch mit RNA-Viren aufweisen, deutet darauf hin, dass RDHVs vermutlich weiter verbreitet sind als anfangs angenommen.<ref name="Rosario2012" /> |
||
Für die [[Klade]] (Verwandtschaftsgruppe) der BSL-RDHV-ähnlichen chimären Viren<ref group="Anm.">Der Begriff „chimäres Virus“ wird je nach Autor unterschiedlich eng ausgelegt: Es kann ein Virus (bzw. Contig) aus der Klade von BSL-RDHV (d. h. ein Mitglied der ''Cruciviridae'') gemeint sein, ein beliebiges RDHV, oder eine beliebige [[Rekombination (Genetik)#Rekombination bei Viren|Rekombination]] (etwa [[Reassortment]] bei ''[[Influenzavirus#Influenzavirus A|Influenzavirus A]]'' mit segmentiertem Genom).</ref> |
Für die [[Klade]] (Verwandtschaftsgruppe) der BSL-RDHV-ähnlichen chimären Viren<ref group="Anm.">Der Begriff „chimäres Virus“ wird je nach Autor unterschiedlich eng ausgelegt: Es kann ein Virus (bzw. Contig) aus der Klade von BSL-RDHV (d. h. ein Mitglied der ''Cruciviridae'') gemeint sein, ein beliebiges RDHV, oder eine beliebige [[Rekombination (Genetik)#Rekombination bei Viren|Rekombination]] (etwa [[Reassortment]] bei ''[[Influenzavirus#Influenzavirus A|Influenzavirus A]]'' mit segmentiertem Genom).</ref> |
||
innerhalb der Supergruppe der CRESS-DNA-Viren — dem heutigen Phylum ''Cressdnaviricota'' — wurde die informelle Bezeichnung „'''Cruciviren'''“ (en. {{lang|en|'''cruciviruses'''}}, CruVs),<ref name=HigueraNacho /><ref name=Harth2020 /> und darüber hinaus der taxonomischen Rang einer Familie mit Namen '''''Cruciviridae''''' vorgeschlagen.<ref name=HigueraNacho /><ref name="NCBI_Cruci" /> |
innerhalb der Supergruppe der CRESS-DNA-Viren — dem heutigen Phylum ''Cressdnaviricota'' — wurde die informelle Bezeichnung „'''Cruciviren'''“ (en. {{lang|en|'''cruciviruses'''}}, CruVs),<ref name="HigueraNacho" /><ref name="Harth2020" /> und darüber hinaus der taxonomischen Rang einer Familie mit Namen '''''Cruciviridae''''' vorgeschlagen.<ref name="HigueraNacho" /><ref name="NCBI_Cruci" /> |
||
== Anmerkungen == |
== Anmerkungen == |
||
| Zeile 152: | Zeile 151: | ||
== Einzelnachweise == |
== Einzelnachweise == |
||
<references responsive> |
<references responsive> |
||
<ref name="ICTV_MSL#37">ICTV: [https://talk.ictvonline.org/files/master-species-lists/m/msl/13425 ICTV Master Species List 2021.v1], New MSL including all taxa updates since the 2020 release, March 2022 (MSL #37)</ref> |
<ref name="ICTV_MSL#37"> |
||
ICTV: [https://talk.ictvonline.org/files/master-species-lists/m/msl/13425 ICTV Master Species List 2021.v1], New MSL including all taxa updates since the 2020 release, March 2022 (MSL #37) |
|||
</ref> |
|||
<ref name="NCBI_DfCyclV"> |
<ref name="NCBI_DfCyclV"> |
||
NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1234878 Dragonfly cyclicusvirus] (species, unclassified ssDNA) |
NCBI: [https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=1234878 Dragonfly cyclicusvirus] (species, unclassified ssDNA) |
||
</ref> |
</ref> |
||
<ref name=chandler |
<ref name="chandler"> |
||
{{cite journal |author=M. Chandler, F. de la Cruz, F. Dyda, A. B. Hickman, G. Moncalian, B. Ton-Hoang |date=2013-08 |title=Breaking and Joining Single-Stranded DNA: The HUH Endonuclease Superfamily |journal=Nat Rev Microbiol |volume=11 |issue=8 |pages=525–538 |doi=10.1038/nrmicro3067 |pmc=6493337 |pmid=23832240}} |
{{cite journal |author=M. Chandler, F. de la Cruz, F. Dyda, A. B. Hickman, G. Moncalian, B. Ton-Hoang |date=2013-08 |title=Breaking and Joining Single-Stranded DNA: The HUH Endonuclease Superfamily |journal=Nat Rev Microbiol |volume=11 |issue=8 |pages=525–538 |doi=10.1038/nrmicro3067 |pmc=6493337 |pmid=23832240}} |
||
</ref> |
</ref> |
||
<ref name=Dayaram2016> |
<ref name="Dayaram2016"> |
||
Anisha Dayarama, Mark L. Galatowitsch, Gerardo R. Argüello-Astorg, Katherine van Bysterveldt, Simona Kraberger, Daisy Stainton, Jon S. Harding, Philippe Roumagnac, Darren P. Martin, Pierre Lefeuvre, Arvind Varsani: [https://www.sciencedirect.com/science/article/abs/pii/S1567134816300429 Diverse circular replication-associated protein encoding viruses circulating in invertebrates within a lake ecosystem], in: Infection, Genetics and Evolution 39, April 2016, Epub 10. Februar 2016, S. 304–316. [[doi:10.1016/j.meegid.2016.02.011]] |
Anisha Dayarama, Mark L. Galatowitsch, Gerardo R. Argüello-Astorg, Katherine van Bysterveldt, Simona Kraberger, Daisy Stainton, Jon S. Harding, Philippe Roumagnac, Darren P. Martin, Pierre Lefeuvre, Arvind Varsani: [https://www.sciencedirect.com/science/article/abs/pii/S1567134816300429 Diverse circular replication-associated protein encoding viruses circulating in invertebrates within a lake ecosystem], in: Infection, Genetics and Evolution 39, April 2016, Epub 10. Februar 2016, S. 304–316. [[doi:10.1016/j.meegid.2016.02.011]] |
||
</ref> |
</ref> |
||
<ref name=Diemer2012> |
<ref name="Diemer2012"> |
||
Geoffrey S. Diemer, Kenneth M. Stedman: [https://pdxscholar.library.pdx.edu/bio_fac/30/ A novel virus genome discovered in an extreme environment suggests recombination between unrelated groups of RNA and DNA viruses], auf: PDXScholar, University Library, Portland State University, 6. Dezember 2012. Ebenso in: Biol Direct. Band 7, Nr. 13, 11. Juni 2012, |
Geoffrey S. Diemer, Kenneth M. Stedman: [https://pdxscholar.library.pdx.edu/bio_fac/30/ A novel virus genome discovered in an extreme environment suggests recombination between unrelated groups of RNA and DNA viruses], auf: PDXScholar, University Library, Portland State University, 6. Dezember 2012. Ebenso in: Biol Direct. Band 7, Nr. 13, 11. Juni 2012, [[doi:10.1186/1745-6150-7-13]], PMID 22515485, {{PMC|3372434}} |
||
</ref> |
</ref> |
||
<ref name=Harth2020> |
<ref name="Harth2020"> |
||
Richard Harth: [https://biodesign.asu.edu/news/criss-crossing-viruses-give-rise-peculiar-hybrid-variants Criss-crossing viruses give rise to peculiar hybrid variants], Arizona State University, Biodesign Institute, 29. Oktober 2020. Siehe insbes. [https://biodesign.asu.edu/sites/default/files/news/biodesign_cruciviruses_arvind_102920-01.jpg Graphik] |
Richard Harth: [https://biodesign.asu.edu/news/criss-crossing-viruses-give-rise-peculiar-hybrid-variants Criss-crossing viruses give rise to peculiar hybrid variants], Arizona State University, Biodesign Institute, 29. Oktober 2020. Siehe insbes. [https://biodesign.asu.edu/sites/default/files/news/biodesign_cruciviruses_arvind_102920-01.jpg Graphik] |
||
</ref> |
</ref> |
||
<ref name=HigueraNacho> |
<ref name="HigueraNacho"> |
||
Ignacio (Nacho) de la Higuera, Ellis Torrance, Amber Maluenda, George Kasun, Max Larson; Rita Clare: [https://www.extremeviruses.org/crucivirusesrdhvs Cruciviruses / RNA-DNA Hybrid Viruses], Extreme Virus Lab (Stedman Lab), 2018 |
Ignacio (Nacho) de la Higuera, Ellis Torrance, Amber Maluenda, George Kasun, Max Larson; Rita Clare: [https://www.extremeviruses.org/crucivirusesrdhvs Cruciviruses / RNA-DNA Hybrid Viruses], Extreme Virus Lab (Stedman Lab), 2018 |
||
</ref> |
</ref> |
||
<ref name=Kazlauskas2017> |
<ref name="Kazlauskas2017"> |
||
{{cite journal |url= https://www.sciencedirect.com/science/article/pii/S0042682217300338 |author=Darius Kazlauskas, Anisha Dayaram, Simona Kraberger, Sharyn Goldstien, Arvind Varsani, Mart Krupovic |title=Evolutionary history of ssDNA bacilladnaviruses features horizontal acquisition of the capsid gene from ssRNA nodaviruses |journal=Virology |date=2017 |volume=504 |pages=114–121 |doi=10.1016/j.virol.2017.02.001 |pmid=28189969<!--|doi-access=free-->}} |
{{cite journal |url= https://www.sciencedirect.com/science/article/pii/S0042682217300338 |author=Darius Kazlauskas, Anisha Dayaram, Simona Kraberger, Sharyn Goldstien, Arvind Varsani, Mart Krupovic |title=Evolutionary history of ssDNA bacilladnaviruses features horizontal acquisition of the capsid gene from ssRNA nodaviruses |journal=Virology |date=2017 |volume=504 |pages=114–121 |doi=10.1016/j.virol.2017.02.001 |pmid=28189969<!--|doi-access=free-->}} |
||
</ref> |
</ref> |
||
<ref name=Koonin2012> |
<ref name="Koonin2012"> |
||
Eugene V. Koonin, Valerian V. Dolja: [https://bmcbiol.biomedcentral.com/articles/10.1186/1741-7007-10-54 Expanding networks of RNA virus evolution], In: BMC Biology Band 10, Nr. 54, 20. Juni 2012, |
Eugene V. Koonin, Valerian V. Dolja: [https://bmcbiol.biomedcentral.com/articles/10.1186/1741-7007-10-54 Expanding networks of RNA virus evolution], In: BMC Biology Band 10, Nr. 54, 20. Juni 2012, [[doi:10.1186/1741-7007-10-54]] |
||
</ref> |
</ref> |
||
<ref name="proposal"> |
<ref name="proposal"> |
||
{{cite web| authors=Eugene V. Koonin, Valerian V. Dolja, Mart Krupovic, Arvind Varsani, Y. I. Wolf, N. Yutin, M. Zerbini, J. H. Kuhn| title=Create a megataxonomic framework, filling all principal taxonomic ranks, for ssDNA viruses| url=https://talk.ictvonline.org/ictv/proposals/2019.005G.zip| website=International Committee on Taxonomy of Viruses| access-date=2020-05-27| language=en| format=docx| date=2019-10-18}} |
{{cite web| authors=Eugene V. Koonin, Valerian V. Dolja, Mart Krupovic, Arvind Varsani, Y. I. Wolf, N. Yutin, M. Zerbini, J. H. Kuhn| title=Create a megataxonomic framework, filling all principal taxonomic ranks, for ssDNA viruses| url=https://talk.ictvonline.org/ictv/proposals/2019.005G.zip| website=International Committee on Taxonomy of Viruses| access-date=2020-05-27| language=en| format=docx| date=2019-10-18}} |
||
</ref> |
</ref> |
||
<ref name=Krupovic2014> |
<ref name="Krupovic2014"> |
||
Mart Krupovic, Eugene V. Koonin: [https://www.nature.com/articles/srep05347 Evolution of eukaryotic single-stranded DNA viruses of the ''Bidnaviridae'' family from genes of four other groups of widely different viruses], in: [[Nature]] Sci Rep Band 4, Nr. 5347, 18. Juni 2014, |
Mart Krupovic, Eugene V. Koonin: [https://www.nature.com/articles/srep05347 Evolution of eukaryotic single-stranded DNA viruses of the ''Bidnaviridae'' family from genes of four other groups of widely different viruses], in: [[Nature]] Sci Rep Band 4, Nr. 5347, 18. Juni 2014, [[doi:10.1038/srep05347]]. Siehe insbes. [https://onlinelibrary.wiley.com/cms/asset/1174a347-1055-4b1c-9259-abc5907e7da6/mfig001.jpg Fig. 1] |
||
</ref> |
</ref> |
||
<ref name=Krupovic2019> |
<ref name="Krupovic2019"> |
||
Krupovic M, Varsani A, Kuhn J, Kazlauskas D, Breitbart M, Delwart E, Rosario K, Yutin N, Wolf YI, Harrach B, Zerbini FM, Dolja VV, Koonin EV: [https://talk.ictvonline.org/files/ictv_official_taxonomy_updates_since_the_8th_report/m/animal-dna-viruses-and-retroviruses/9405/download 2019.012D.R.Cressdnaviricota], Proposal to ICTV (accepted) |
Krupovic M, Varsani A, Kuhn J, Kazlauskas D, Breitbart M, Delwart E, Rosario K, Yutin N, Wolf YI, Harrach B, Zerbini FM, Dolja VV, Koonin EV: [https://talk.ictvonline.org/files/ictv_official_taxonomy_updates_since_the_8th_report/m/animal-dna-viruses-and-retroviruses/9405/download 2019.012D.R.Cressdnaviricota], Proposal to ICTV (accepted) |
||
</ref> |
</ref> |
||
<ref name=Rosario2012> |
<ref name="Rosario2012"> |
||
Karyna Rosario, Anisha Dayaram, Milen Marinov, Jessica Ware, Simona Kraberger, Daisy Stainton, Mya Breitbart, Arvind Varsani: [https://www.microbiologyresearch.org/content/journal/jgv/10.1099/vir.0.045948-0 Diverse circular ssDNA viruses discovered in dragonflies (Odonata: Epiprocta)], in: Journal of General Virology Band 93, Nr. 12, 1. Dezember 2012, |
Karyna Rosario, Anisha Dayaram, Milen Marinov, Jessica Ware, Simona Kraberger, Daisy Stainton, Mya Breitbart, Arvind Varsani: [https://www.microbiologyresearch.org/content/journal/jgv/10.1099/vir.0.045948-0 Diverse circular ssDNA viruses discovered in dragonflies (Odonata: Epiprocta)], in: Journal of General Virology Band 93, Nr. 12, 1. Dezember 2012, [[doi:10.1099/vir.0.045948-0]] |
||
</ref> |
</ref> |
||
<ref name=Roux2013> |
<ref name="Roux2013"> |
||
Simon Roux, François Enault, Gisèle Bronner, Daniel Vaulot, [[Patrick Forterre]], Mart Krupovic: [https://www.nature.com/articles/ncomms3700 Chimeric viruses blur the borders between the major groups of eukaryotic single-stranded DNA viruses], in: Nat Commun 4:2700, 6. November 2013, |
Simon Roux, François Enault, Gisèle Bronner, Daniel Vaulot, [[Patrick Forterre]], Mart Krupovic: [https://www.nature.com/articles/ncomms3700 Chimeric viruses blur the borders between the major groups of eukaryotic single-stranded DNA viruses], in: Nat Commun 4:2700, 6. November 2013, [[doi:10.1038/ncomms3700]]. Siehe insbesondere [https://static-content.springer.com/esm/art%3A10.1038%2Fncomms3700/MediaObjects/41467_2013_BFncomms3700_MOESM611_ESM.xls Supplement 2] (xls). |
||
</ref> |
</ref> |
||
<ref name=Steel2016> |
<ref name="Steel2016"> |
||
Olivia Steel, Simona Kraberger, Alyssa Sikorski, Laura M.Young, Ryan J. Catchpole, Aaron J. Stevens, Jenny J. Ladley, Dorien S. Coraya, Daisy Stainton, Anisha Dayaram, Laurel Julian, Katherine van Bysterveldt, Arvind Varsani: [https://www.sciencedirect.com/science/article/abs/pii/S1567134816301794 Circular replication-associated protein encoding DNA viruses identified in the faecal matter of various animals in New Zealand], in: Genetics and Evolution 43, September 2016, S. 151–164, [[doi:10.1016/j.meegid.2016.05.008]] |
Olivia Steel, Simona Kraberger, Alyssa Sikorski, Laura M.Young, Ryan J. Catchpole, Aaron J. Stevens, Jenny J. Ladley, Dorien S. Coraya, Daisy Stainton, Anisha Dayaram, Laurel Julian, Katherine van Bysterveldt, Arvind Varsani: [https://www.sciencedirect.com/science/article/abs/pii/S1567134816301794 Circular replication-associated protein encoding DNA viruses identified in the faecal matter of various animals in New Zealand], in: Genetics and Evolution 43, September 2016, S. 151–164, [[doi:10.1016/j.meegid.2016.05.008]] |
||
</ref> |
</ref> |
||
<ref name=Tisza2020> |
<ref name="Tisza2020"> |
||
Michael J. Tisza, Diana V. Pastrana, Nicole L. Welch, Brittany Stewart, Alberto Peretti, Gabriel J. Starrett, Yuk-Ying S. Pang, Siddharth R. Krishnamurthy, Patricia A. Pesavento, David H. McDermott, Philip M. Murphy, Jessica L. Whited, Bess Miller, Jason Brenchley, Stephan P. Rosshart, Barbara Rehermann, John Doorbar, Blake A. Ta'ala, Olga Pletnikova, Juan C. Troncoso, Susan M. Resnick, Ben Bolduc, Matthew B. Sullivan, Arvind Varsani, Anca M. Segall, Christopher B. Buck: [https://elifesciences.org/articles/51971#content Discovery of several thousand highly diverse circular DNA viruses], in: eLife 9, 4. Februar 2020, e51971; [[doi:10.7554/eLife.51971]]. |
Michael J. Tisza, Diana V. Pastrana, Nicole L. Welch, Brittany Stewart, Alberto Peretti, Gabriel J. Starrett, Yuk-Ying S. Pang, Siddharth R. Krishnamurthy, Patricia A. Pesavento, David H. McDermott, Philip M. Murphy, Jessica L. Whited, Bess Miller, Jason Brenchley, Stephan P. Rosshart, Barbara Rehermann, John Doorbar, Blake A. Ta'ala, Olga Pletnikova, Juan C. Troncoso, Susan M. Resnick, Ben Bolduc, Matthew B. Sullivan, Arvind Varsani, Anca M. Segall, Christopher B. Buck: [https://elifesciences.org/articles/51971#content Discovery of several thousand highly diverse circular DNA viruses], in: eLife 9, 4. Februar 2020, e51971; [[doi:10.7554/eLife.51971]]. |
||
</ref> |
</ref> |
||
Version vom 30. April 2022, 10:14 Uhr
| Cressdnaviricota | ||||||||
|---|---|---|---|---|---|---|---|---|

Gereinigtes Maize streak virus (MSV), | ||||||||
| Systematik | ||||||||
| ||||||||
| Taxonomische Merkmale | ||||||||
| ||||||||
| Wissenschaftlicher Name | ||||||||
| Cressdnaviricota | ||||||||
| Kurzbezeichnung | ||||||||
| CRESS | ||||||||
| Links | ||||||||
|

Cressdnaviricota (informell CRESS-DNA-Viren) ist die Bezeichnung für ein Phylum von Viren im Reich Shotokuvirae [en], Realm Monodnaviria, deren Genom aus Einzelstrang-DNA (ssDNA) besteht und deren natürliche Wirte Eukaryoten (evtl. auch Archaeen) sind. Sie replizieren ihr Genom per Rolling-Circle-Replikation (englisch rolling circle replication [en], RCR), gestartet durch ein viruseigenes Protein mit der Bezeichnung REP.[2][3] Wegen dieser Replikationsmethode stellen die Cressdnaviricota die „prototypischen“ Vertreter des Realms Monodnaviria dar. Mit der Einrichtung dieses Phylums hat das International Committee on Taxonomy of Viruses (ICTV) die zuvor informell als Supergruppe der CRESS-DNA-Viren (en. CRESS DNA viruses) als offizielles Taxon anerkannt. Sie umspannen derzeit sieben Familien.[4][5]
Die CRESS-DNA-Viren werden mit einer Vielzahl von Krankheiten in Verbindung gebracht. Darunter sind Krankheiten bei wirtschaftlich wichtigen Nutzpflanzen sowie eine Vielzahl von Krankheiten bei Tieren einschließlich des Menschen.[5]
Die Bezeichnung Cressdnaviricota setzt sich zusammen aus dem Akronym CRESS (circular REP-encoding single-stranded ‚zirkulär, REP-kodierend, einzelsträngig‘), DNA, und der Endung -viricota für Virusphyla.[4][6] Dieses ICTV-bestätigte Phylum von DNA-Viren mit zirkulärem Einzelstrang-DNA-Genom ist nicht zu verwechseln mit der vorgeschlagenen Ordnung „Crassvirales“ mit zirkuläem Doppelstrang-DNA-Genom, deren Wirte Bakterien sind.
Beschreibung
Genom

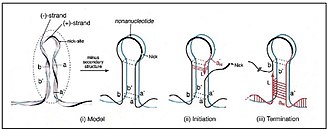
Das Genom der Cressdnaviricota kodiert für zwei wesentliche Proteine:[4]
- das REP (oder Rep) – dieses startet (initiiert) die Rolling-Circle-Replikation (englisch rolling circle replication [en], RCR) des zirkulären Virusgenoms. Sie ist im Phylum konserviert, d. h. wenig unterschiedlich, da sie sich im Lauf der Evolution nicht wesentlich verändert hat. Rep ist eine Endonuklease der HUH-Superfamilie [en]. Endonukleasen sind Enzyme, die Phosphodiesterbindungen innerhalb einer Polynukleotidkette spalten können. HUH- oder HuH-Endonukleasen sind spezielle Endonukleasen, die
- ein HUH-Motiv enthalten, das aus zwei Histidinresten besteht, die durch einen sperrigen hydrophoben Rest getrennt sind,
- und ein Y-Motiv, das einen oder zwei Tyrosinreste enthält.
- HUH-Endonukleasen werden grob in zwei Kategorien von Enzymen eingeteilt: Replikationsinitiationsproteine (Rep) und Relaxase-/Mobilisierungsproteine. Die HUH-Endonuklease der ssDNA-Viren gehört zur ersten Gruppe (Rep), weil die von ihr vermittelte Spaltung des (zirkularen) viralen Genoms die Replikation einleitet.[5][7]
- das CP (auch Cp, CAP oder Cap) – dieses bezeichnet ein Kapsidprotein, also ein Protein, das das Kapsid der Virusteilchen (Virionen) aufbaut.
Im Gegensatz zu den REP-Proteinen sind die Kapsidproteine der CRESS-DNA-Viren nicht ortholog, sondern wurden mehrfach unabhängig voneinander von RNA-Viren übernommen. Daher ist das Gen für das REP-Protein das (alleinige) Kriterium, um über die Zugehörigkeit zum Phylum Aufschluss zu geben.[4]
Wirte
Die Familien der Cressdnaviricota parasitieren Wirte in der gesamten Domäne der Eukaryoten, mit:
- Pflanzen (Nanoviridae, Geminiviridae),
- Pilzen (Genomoviridae),
- Tieren (Circoviridae) und
- Algen (Bacilladnaviridae).
Mitglieder der beiden Familien Smacoviridae und Redondoviridae wurden durch Metagenomik entdeckt wurden infizieren vermutlich ebenfalls Tiere, obwohl auch eine Assoziation von Smacoviren mit methanogenen Archaeen vorgeschlagen wurde.[4]
Systematik

Die Systematik der CRESS-DNA-Viren ist nach ICTV (Master Species List #35) und Vorschlag 2021.010F folgende (Stand 1. April 2022):[9][10]
- Phylum Cressdnaviricota
- Klasse Arfiviricetes [en] (Ar von Arginin; fi von Finger; Eigenschaft des Rep-Proteins, konserviert unter den Mitgliedern)
- Ordnung Baphyvirales – Familie Bacilladnaviridae
- Ordnung Cirlivirales – Familien Circoviridae und Vilyaviridae (letztere 12 Gattungen), sowie „CRESSV1“ (alias „CRESS1“) und „CRESSV3“ (alias „CRESS3“)
- Ordnung Cremevirales – Familie Smacoviridae
- Ordnung Mulpavirales – Familien Metaxyviridae[11][12] und Nanoviridae, sowie „CRESSV4“ (alias „CRESS4“) und „CRESSV4“ (alias „CRESS4“)
- Ordnung Recrevirales – Familie Redondoviridae
- Ordnung Rivendellvirales – Familie Naryaviridae (4 Gattungen, Wirt: Entamoeba)
- Ordnung Rohanvirales – Familie Nenyaviridae (5 Gattungen, Wirt: Giardia), sowie „CRESSV2“ (alias „CRESS2“)
- Klasse Repensiviricetes
- Ordnung Geplafuvirales – Familien Geminiviridae und Genomoviridae, evtl. auch Alphasatellitidae und Kandidaten-Gattung/Klade „Nepavirus“
- Familie „CRESS1“ (alias „CRESSV1“) – zu Cirlivirales, d. h. nahe Circoviridae
- Familie „CRESS1-Rec1“ – nahe Bacilladnaviridae oder Circoviridae
- Familie „CRESS2“ (alias „CRESSV2“) – zu Rohanvirales
- Gattung(?) „Circularisvirus“[16] (zusammen mit DfCirV zu „CRESS2“)
- Spezies: „Dragonfly circularisvirus“ (DfCirV)[17] (zu „CRESS2“[18])
- Spezies: „Cybaeus spider associated circular virus 1“ (CySACV-1), in Spinnen der Gattung Cybaeus[19]
- Spezies: „Golden silk orbweaver associated circular virus 1“ (GoSOrbACV-1), in Spinnen der Gattung Nephila[19]
- Spezies: „Longjawed orbweaver circular virus 1“ (LjOrbCV-1), in Spinnen der Gattung Leucauge[19]
- Spezies: „Spinybacked orbweaver circular virus 1“ (SpOrbCV-1), in Spinnen der Gattung Gasteracantha[19]
- Familie „CRESS3“ (alias „CRESSV3“) – zu Cirlivirales
- Familien „CRESS4“ (alias „CRESSV4“) und „CRESS5“ (alias „CRESSV5“) – zu Mulpavirales, d. h. nahe Nanoviridae
- Familien oder Kladen „CRESS6“ (alias „CRESSV6“[6]) und „CRESS-Rec2“ – nahe Geplafuvirales
- Familie oder Klade „GasCSV-like viruses“ mit Spezies „Gastropod associated circular ssDNA virus“ (GasCSV)[21][22]
- Familie „Cruciviridae“[23][24] ohne nähere Zuordnung, mit „Boiling Springs Lake RNA-DNA hybrid virus“ (BSL-RDHV) und „Idotea-Virus IWaV278“[25]
- Gattung oder Klade „Volvovirus“[26]
- Spezies: „Acheta domesticus volvovirus“ (AdVVV), in Heimchen[27][28]
- Spezies: „Cricket associated circular virus 1“ (CrACV-1), in Feldgrillen[19] (zu „nicht-klassifizierten CRESS“[18])
- „bacteria-tropic branch of the CRESS virus supergroup“ (aus Metagenomik)[29]
- Contig „ctbb30“
- Contig „ctbc27“
- keinem höheren Rang zugeordnete vorgeschlagene Spezies:
RNA-DNA-Hybridviren
Mehrere der bis heute entdeckten CRESS-DNA-Viren haben ein Genom, das signifikant größer ist, als das bei diesem Phylum normalerweise anzutreffen ist. Denkbar wäre, dass dies das Ergebnis von Duplikationsereignissen ist. Darüber hinaus wurde eine Akquisition von Kapsiden mit einer Symmetrie T=3 von den ssRNA-Familien Nodaviridae [en] und Tombusviridae als Ursprung der Familie Bacilladnaviridae[31] beziehungsweise der vorgeschlagenen Familie „Cruciviridae“[32][33] vermutet. Offenbar gab es noch weitere Rekombinationsereignisse von ssRNA-Viren oder ssDNA-Bakteriophagen mit CRESS-DNA-Viren.[29]
Bei seit 2001 durchgeführten Metagenomanalysen in den Geothermalgebieten des Lassen-Volcanic-Nationalparks (Geothermal areas in Lassen Volcanic National Park, englisch) in Nordkalifornien wurde eine ssDNA-Genomsequenz (Contig) gefunden, in der sich einerseits in einem mutmaßlichen REP-Gen Homologien zu dem von Porcines Circovirus-2 (PCV-2), Fam. Circoviridae, zeigten. Andererseits gab es in einem mutmaßlichen Kapsidprotein-Gen Homologien zu denen der Familie Tombusviridae (sowie den vorgeschlagenen Spezies „Sclerophthora macrospora virus A“, SmV-A[34] und „Plasmopara halstedii virus A“[35] – beide infizieren Eipilze der Familie Peronosporaceae.[24][36]
Diese gefundenen chimären Contigs wurde einem neuen Virus zugeordnet, für das nach dem dortigen Fundort (Boiling Springs Lake 40,4355° N, 121,3971° W) die Bezeichnung „Boiling Springs Lake RNA-DNA Hybrid Virus“ (BSL-RDHV) vorgeschlagen wurde.[37][24][38]
Weitere Metagenomanalysen an verschiedenen Orten lieferten immer mehr an hybriden Contigs. Insgesamt sind von diesen mit Stand Oktober 2020 nicht wenige als 461 Kandidaten bekannt.[24][38]
Zunächst schienen alle im CAP dem BSL-RDHV homolog zu sein; das REP-Gen scheint dagegen mehrmals von anderen CRESS-DNA-Familien (Circoviridae, Nanoviridae, Geminiviridae) per horizontalem Gentransfer (HGT) übernommen worden zu sein.[36]
Im Laufe der Zeit fanden sich jedoch auch RNA-DNA-Hybridviren (RDHVs), die augenscheinlich nicht der Verwandtschaft von BSL-RDHV angehören: Ein bei Untersuchung der Vielfalt von mit Libellen assoziierten ssDNA-Viren gefundenes Contig mit der Bezeichnung DfCyclV („Dragonfly cyclicusvirus“)[39] zeigte eine schwache, aber signifikante Homologie eines mutmaßlichen Proteins zum CAP des Satelliten-Tabaknekrosevirus (en. Satellite tobacco necrosis virus, STNV oder STMV).[40][41] Das CAP des Satelliten-Tabaknekrosevirus unterscheidet sich jedoch in Sequenz und Struktur grundlegend von denen der Tombusviridae und ähnelt am ehesten den CAPs der Geminiviridae. Daher ist vermutlich DfCyclV kein Mitglied der Klade der BSL-RDHV-ähnlichen Viren.[36]
Über den Fund von RDHVs weiteren Typs berichteten zudem Koonin und Dolja (2012);[42] und die ssDNA-Familie Bidnaviridae hat offenbar auch eine „turbulente“ evolutionäre Vergangenheit u. a. mit einer mutmaßlichen Gen-Übernahme von den dsRNA-Viren der Reoviridae[43]
Die Entdeckung weiterer Contigs (Genome aus Metagenomanalysen), die Ähnlichkeiten sowohl mit DNA- als auch mit RNA-Viren aufweisen, deutet darauf hin, dass RDHVs vermutlich weiter verbreitet sind als anfangs angenommen.[41]
Für die Klade (Verwandtschaftsgruppe) der BSL-RDHV-ähnlichen chimären Viren[Anm. 1] innerhalb der Supergruppe der CRESS-DNA-Viren — dem heutigen Phylum Cressdnaviricota — wurde die informelle Bezeichnung „Cruciviren“ (en. cruciviruses, CruVs),[24][38] und darüber hinaus der taxonomischen Rang einer Familie mit Namen Cruciviridae vorgeschlagen.[24][23]
Anmerkungen
- ↑ Der Begriff „chimäres Virus“ wird je nach Autor unterschiedlich eng ausgelegt: Es kann ein Virus (bzw. Contig) aus der Klade von BSL-RDHV (d. h. ein Mitglied der Cruciviridae) gemeint sein, ein beliebiges RDHV, oder eine beliebige Rekombination (etwa Reassortment bei Influenzavirus A mit segmentiertem Genom).
Einzelnachweise
- ↑ a b ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- ↑ ssDNA Rolling circle. In: ViralZone. Swiss Institute of Bioinformatics, abgerufen am 29. März 2021.
- ↑ Virus Taxonomy: 2019 Release. In: talk.ictvonline.org. International Committee on Taxonomy of Viruses, abgerufen am 25. April 2020.
- ↑ a b c d e Krupovic M, Varsani A, Kuhn J, Kazlauskas D, Breitbart M, Delwart E, Rosario K, Yutin N, Wolf YI, Harrach B, Zerbini FM, Dolja VV, Koonin EV: 2019.012D.R.Cressdnaviricota, Proposal to ICTV (accepted)
- ↑ a b c Eugene V. Koonin, Valerian V. Dolja, Mart Krupovic, Arvind Varsani, Y. I. Wolf, N. Yutin, M. Zerbini, J. H. Kuhn: Create a megataxonomic framework, filling all principal taxonomic ranks, for ssDNA viruses. (docx) In: International Committee on Taxonomy of Viruses. 18. Oktober 2019, abgerufen am 27. Mai 2020 (englisch).
- ↑ a b Mart Krupovic, Arvind Varsani, Jens H. Kuhn, D. Kazlauskas, M. Breitbart, E. Delwart, K. Rosario, N. Yutin, Y. I. Wolf, B. Harrach, F. M. Zerbini, V. V. Dolja, E. V. Koonin: Create 1 new phylum (Cressdnaviricota) including 2 classes and 6 orders for classification of CRESS-DNA viruses, Vorschlag 2019.012DA.v1.Cressdnaviricota (ZIP von docx und xlsx); April 2019
- ↑ M. Chandler, F. de la Cruz, F. Dyda, A. B. Hickman, G. Moncalian, B. Ton-Hoang: Breaking and Joining Single-Stranded DNA: The HUH Endonuclease Superfamily. In: Nat Rev Microbiol. 11. Jahrgang, Nr. 8, August 2013, S. 525–538, doi:10.1038/nrmicro3067, PMID 23832240, PMC 6493337 (freier Volltext).
- ↑ a b Darius Kazlauskas, Arvind Varsani, Mart Krupovic: Pervasive Chimerism in the Replication-Associated Proteins of Uncultured Single-Stranded DNA Viruses, in: MDPI Viruses, Band 10, Nr. 4, Special Issue Viral Recombination: Ecology, Evolution and Pathogenesis, 10. April 2018, S. 187; doi:10.3390/v10040187, PMID 29642587; siehe insbes. Fig. S1 in Supplement S1 (ZIP: pdf, xlsx)
- ↑ ICTV: ICTV Master Species List 2021.v1, New MSL including all taxa updates since the 2020 release, March 2022 (MSL #37)
- ↑ Arvind Varsani, Mart Krupovic: Create two new orders, three new families and 21 new genera in the class Arfiviricetes (Cressdnaviricota) (ZIP: pdf, xlsx). Proposal 021.010F, Oktober 2020. Siehe insbes. Fig. 1.
- ↑ Bruno Gronenborn, Arvind Varsani, J. W. Randles, H. J. Vetten, J. E. Thomas: Create a new subfamily (Petromoalphasatellitinae) with four new genera and six new species (Alphasatellitidae) (ZIP: docx, xlsx), Vorschlag 2020.001P.R.Petromoalphasatellitinae_nsf and das ICTV (angenommen)
- ↑ NCBI: Coconut foliar decay virus (species)
- ↑ Darius Kazlauskas, Arvind Varsani, Eugene V. Koonin, Mart Krupovic: Multiple origins of prokaryotic and eukaryotic single-stranded DNA viruses from bacterial and archaeal plasmids. In: Nature Communicastions, Band 10, Nr. 3425, 31. Juli 2019, doi:10.1038/s41467-019-11433-0, PMID 31366885.
- ↑ Lele Zhao, Karyna Rosario, Mya Breitbart, Siobain Duffy: Chapter Three - Eukaryotic Circular Rep-Encoding Single-Stranded DNA (CRESS DNA) Viruses: Ubiquitous Viruses With Small Genomes and a Diverse Host Range, in: Advances in Virus Research, Band 103, 2019, S. 71–133, doi:10.1016/bs.aivir.2018.10.001, Epub 5. Dezember 2018. Insbes. siehe Fig. 3
- ↑ Hiroto Kaneko, Morgan Gaia, Rodrigo Hernández-Velázquez, Patrick Forterre, Curtis A. Suttle, Hiroyuki Ogata et al.: Eukaryotic virus composition can predict the efficiency of carbon export in the global ocean, in: iScience, Band 24, Nr. 1, 22. Januar 2021, 102002, Epub 29. Dezember 2020, doi:10.1016/j.isci.2020.102002, PMC 7811142 (freier Volltext), PMID 33490910; siehe Fig. 1(C)
- ↑ NCBI: Circularisvirus (clade)
- ↑ a b c Karyna Rosario, Anisha Dayaram, Milen Marinov, Jessica Ware, Simona Kraberger, Daisy Stainton, Mya Breitbart, Arvind Varsani: Diverse circular ssDNA viruses discovered in dragonflies (Odonata: Epiprocta), in: Journal of General Virology Band 93, Nr. 12, 1. Dezember 2012, doi:10.1099/vir.0.045948-0. Hier insbes. Tbl. 2
- ↑ a b c d Darius Kazlauskas, Arvind Varsani, Mart Krupovic: Pervasive Chimerism in the Replication-Associated Proteins of Uncultured Single-Stranded DNA Viruses, in: MDPI Viruses, Band 10, Nr. 4, Special Issue Viral Recombination: Ecology, Evolution and Pathogenesis, 10. April 2018, 187, doi:10.3390/v10040187; siehe Excel-Datei in Supplement S1 (ZIP: pdf, xlsx)
- ↑ a b c d e Karyna Rosario, Kaitlin Mettel, Bayleigh Benner, Ryan Johnson, Catherine Scott, Sohath Yusseff-Vanegas, Christopher Baker, Deby Cassill, Caroline Storer, Arvind Varsani, Mya Breitbart: Virus Discovery in All Three Major Lineages of Terrestrial Arthropods Highlights the Diversity of Single-stranded DNA Viruses Associated with Invertebrates, University of South Florida, College of Marine Science: Scholar Commons — Marine Science Faculty Publications, 2017, 652. Part of the Life Sciences Commons. 11. Oktober 2018 PeerJ, PMID 30324030, PMC 6186406 (freier Volltext), doi:10.7717/peerj.5761
- ↑ NCBI: Dragonfly cyclicusvirus (species)
- ↑ Anisha Dayaram, Sharyn Goldstien, Peyman Zawar-Reza, Christopher Gomez, Jon S. Harding, Arvind Varsani: Novel ssDNA virus recovered from estuarine Mollusc (Amphibola crenata) whose replication associated protein (Rep) shares similarities with Rep-like sequences of bacterial origin. In: J. Gen. Virol., Band 94, Nr. Pt 5, Mai 2013, S. 1104–1110; doi:10.1099/vir.0.050088-0, PMID 23364192, Epub 30. Januar 2013.
- ↑ NCBI Taxonomy Browser: Gastropod associated circular ssDNA virus (species; acronym: GaCSV)
- ↑ a b NCBI: Cruciviridae (family)
- ↑ a b c d e f Ignacio (Nacho) de la Higuera, Ellis Torrance, Amber Maluenda, George Kasun, Max Larson; Rita Clare: Cruciviruses / RNA-DNA Hybrid Viruses, Extreme Virus Lab (Stedman Lab), 2018
- ↑ Geoffrey S. Diemer, Kenneth M. Stedman:A novel virus genome discovered in an extreme environment suggests recombination between unrelated groups of RNA and DNA viruses. In: Biology Direct, Band 7, Nr. 13, 11. Juni 2012; 10.1186/1745-6150-7-13, PMID 22515485, PMC 3372434 (freier Volltext).
- ↑ NCBI: Volvovirus (clade)
- ↑ Hanh T. Pham, Max Bergoin, Peter Tijssen: Acheta domesticus Volvovirus, a Novel Single-Stranded Circular DNA Virus of the House Cricket, in: Genome Announc. 1(2), März-April 2013, e00079-13. Epub 14. März 2013, doi:10.1128/genomeA.00079-13, PMC 3623006 (freier Volltext), PMID 23516206
- ↑ Hanh T. Pham, Hajime Iwao, Max Bergoin, Peter Tijssen: New Volvovirus Isolates from Acheta domesticus (Japan) and Gryllus assimilis (United States), in: ASM Journals: Genome Announcements, Band 1, Nr. 3, 6. Juni 2013, doi:10.1128/genomeA.00328-13
- ↑ a b Michael J. Tisza, Diana V. Pastrana, Nicole L. Welch, Brittany Stewart, Alberto Peretti, Gabriel J. Starrett, Yuk-Ying S. Pang, Siddharth R. Krishnamurthy, Patricia A. Pesavento, David H. McDermott, Philip M. Murphy, Jessica L. Whited, Bess Miller, Jason Brenchley, Stephan P. Rosshart, Barbara Rehermann, John Doorbar, Blake A. Ta'ala, Olga Pletnikova, Juan C. Troncoso, Susan M. Resnick, Ben Bolduc, Matthew B. Sullivan, Arvind Varsani, Anca M. Segall, Christopher B. Buck: Discovery of several thousand highly diverse circular DNA viruses, in: eLife 9, 4. Februar 2020, e51971; doi:10.7554/eLife.51971.
- ↑ NCBI: Dragonfly orbiculatusvirus (species)
- ↑ Darius Kazlauskas, Anisha Dayaram, Simona Kraberger, Sharyn Goldstien, Arvind Varsani, Mart Krupovic: Evolutionary history of ssDNA bacilladnaviruses features horizontal acquisition of the capsid gene from ssRNA nodaviruses. In: Virology. 504. Jahrgang, 2017, S. 114–121, doi:10.1016/j.virol.2017.02.001, PMID 28189969 (sciencedirect.com).
- ↑ Olivia Steel, Simona Kraberger, Alyssa Sikorski, Laura M.Young, Ryan J. Catchpole, Aaron J. Stevens, Jenny J. Ladley, Dorien S. Coraya, Daisy Stainton, Anisha Dayaram, Laurel Julian, Katherine van Bysterveldt, Arvind Varsani: Circular replication-associated protein encoding DNA viruses identified in the faecal matter of various animals in New Zealand, in: Genetics and Evolution 43, September 2016, S. 151–164, doi:10.1016/j.meegid.2016.05.008
- ↑ Anisha Dayarama, Mark L. Galatowitsch, Gerardo R. Argüello-Astorg, Katherine van Bysterveldt, Simona Kraberger, Daisy Stainton, Jon S. Harding, Philippe Roumagnac, Darren P. Martin, Pierre Lefeuvre, Arvind Varsani: Diverse circular replication-associated protein encoding viruses circulating in invertebrates within a lake ecosystem, in: Infection, Genetics and Evolution 39, April 2016, Epub 10. Februar 2016, S. 304–316. doi:10.1016/j.meegid.2016.02.011
- ↑ NCBI: Sclerophthora macrospora virus A (species)
- ↑ NCBI: Plasmopara halstedii virus A (species)
- ↑ a b c Simon Roux, François Enault, Gisèle Bronner, Daniel Vaulot, Patrick Forterre, Mart Krupovic: Chimeric viruses blur the borders between the major groups of eukaryotic single-stranded DNA viruses, in: Nat Commun 4:2700, 6. November 2013, doi:10.1038/ncomms3700. Siehe insbesondere Supplement 2 (xls).
- ↑ Geoffrey S. Diemer, Kenneth M. Stedman: A novel virus genome discovered in an extreme environment suggests recombination between unrelated groups of RNA and DNA viruses, auf: PDXScholar, University Library, Portland State University, 6. Dezember 2012. Ebenso in: Biol Direct. Band 7, Nr. 13, 11. Juni 2012, doi:10.1186/1745-6150-7-13, PMID 22515485, PMC 3372434 (freier Volltext)
- ↑ a b c Richard Harth: Criss-crossing viruses give rise to peculiar hybrid variants, Arizona State University, Biodesign Institute, 29. Oktober 2020. Siehe insbes. Graphik
- ↑ NCBI: Dragonfly cyclicusvirus (species, unclassified ssDNA)
- ↑ NCBI: Satellite tobacco mosaic virus, equivalent Satellite tobacco necrosis virus (no rank), Gattung Virtovirus vs. NCBI: Satellite tobacco necrosis virus ?* (list), Gattung Albetovirus [en]
- ↑ a b Karyna Rosario, Anisha Dayaram, Milen Marinov, Jessica Ware, Simona Kraberger, Daisy Stainton, Mya Breitbart, Arvind Varsani: Diverse circular ssDNA viruses discovered in dragonflies (Odonata: Epiprocta), in: Journal of General Virology Band 93, Nr. 12, 1. Dezember 2012, doi:10.1099/vir.0.045948-0
- ↑ Eugene V. Koonin, Valerian V. Dolja: Expanding networks of RNA virus evolution, In: BMC Biology Band 10, Nr. 54, 20. Juni 2012, doi:10.1186/1741-7007-10-54
- ↑ Mart Krupovic, Eugene V. Koonin: Evolution of eukaryotic single-stranded DNA viruses of the Bidnaviridae family from genes of four other groups of widely different viruses, in: Nature Sci Rep Band 4, Nr. 5347, 18. Juni 2014, doi:10.1038/srep05347. Siehe insbes. Fig. 1
Weblinks
- Mya Breitbart, Karyna Rosario, Hector G. Loyola Irizarry, Siobain Duffy, Erik Lavington, Lele Zhao: CRESSdna.org
- Anisha Dayaram; Arvind Varsani et al.: Discovery of novel circular replication-associated protein encoding single-stranded DNA viruses in ecosystems using viral metagenomic approaches, Dissertation, University of Canterbury, Neuseeland, 12. Dezember 2014
- Caroline Tochetto, Ana Paula Muterle Varela, Diane Alves de Lima, Márcia Regina Loiko, Camila Mengue Scheffer, Willian Pinto Paim, Cristine Cerva, Candice Schmidt, Samuel Paulo Cibulski, Lucía Cano Ortiz, Sidia Maria Callegari Jacques, Ana Cláudia Franco, Fabiana Quoos Mayer, Paulo Michel Roehe: Viral DNA genomes in sera of farrowing sows with or without stillbirths, in: PLoS ONE 15(3), e0230714, 26. März 2020, doi:10.1371/journal.pone.0230714
 Genomkarten (auf Commons)
Genomkarten (auf Commons)