Pyrophosphatasen
| Pyrophosphatasen | ||
|---|---|---|

| ||
| Abb. 1: Links: Anorganische Pyrophosphatase (PPi). Rechts: Diphosphatase ("organische Pyrophosphatase", oP), R1 = organischer Rest, R2 = organischer Rest oder H | ||
| Enzymklassifikation | ||
| EC, Kategorie | 3.6.1.-, Hydrolasen | |
| Reaktionsart | Exergonische Hydrolyse | |
| Substrat | Anionen oder organische Derivate der Diphosphorsäure | |
| Produkte | Anionen oder organische Derivate der Phosphorsäure | |
| Vorkommen | ||
| Übergeordnetes Taxon | Lebewesen | |
Als Pyrophosphatasen werden meist Enzyme bezeichnet, die Anionen der Diphosphorsäure (Pyrophosphate) zu Phosphat aufspalten. Die aktuell gültige Bezeichnung für diese Pyrophosphatasen ist anorganische Diphosphatasen.[1] PPase ist die verbreitete Abkürzung, die im Folgenden verwendet wird.
Daneben werden auch noch (organische) Diphosphatasen, die (organische) Derivate der Pyrophosphorsäure unter Wasseraufnahme spalten, als Pyrophosphatasen bezeichnet.
Anorganische Diphosphatasen (PPasen)
[Bearbeiten | Quelltext bearbeiten]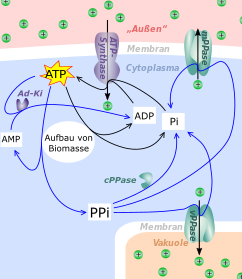
Bei vielen anabolischen Reaktionen werden diese beiden Ausgangsstoffe zurück geliefert (schwarze Pfeile).
Dagegen entsteht bei einer Reihe anderer Prozesse Pyrophosphat (PPi) und AMP. Letzteres wird durch eine Adenylat-Kinase unter ATP-Verbrauch zu ADP umgesetzt. Zur Regeneration von Phosphat können verschieden platzierte Diphosphatasen (cPPase, vPPase und mPPase) dienen.
Anorganisches Diphosphat (Pyrophosphat, PPi) entsteht unter anderem bei allen[2] Biosynthesen von Makromolekülen (Proteine, DNA, RNA, Zellwände, Cellulose etc.).
Seine Spaltung zu Phosphat ist nötig, um dieses Substrat der ATP-Synthase zu regenerieren. Zudem muss die Diphosphat-Konzentration im Cytosol gering gehalten werden, damit die Reaktionen, bei denen es entsteht, nicht ins Stocken kommen.
Die von PPasen katalysierte Hydrolyse von Pyrophosphat (PPi) zu Phosphat (Pi) ist exergon:[3]
Pyrophosphatasen findet man von der Bakterienzelle bis zum menschlichen Organismus in allen Lebewesen. Es gibt drei unterschiedliche Gruppen von PPasen, die sich in ihrer Struktur, ihrer Evolution und ihrem Reaktionsmechanismus grundlegend unterscheiden.
- Zwei Gruppen von im Cytosol gelösten cPPasen (cPPasen I und cPPasen II) setzen die Energie der Hydrolyse als Wärme frei. Sie dienen lediglich der Kontrolle der intrazellulären PPi-Konzentration.[4]
- Bei der dritten, sehr alte Gruppe von anorganischen PPasen, handelt es sich um Membranproteine, das als Ionenpumpen die Energie der Pyrophosphat-Spaltung durch Aufbau eines Membranpotentials konservieren können.[5] Aus dem Zellinneren durch eine mPPase nach außen gepumpte Ionen können dann chemiosmotisch zur ATP-Regenerierung verwendet werden. Quasi baugleich sind die vPPasen, die ihre Fracht in Acidocalcisomen bzw. in eukaryotische Vakuolen pumpen.[6]
Verglichen mit der langsamen, nicht katalysierten Hydrolyse steigern alle Pyrophosphatasen die Geschwindigkeit der Diphosphatspaltung um viele Größenordnungen. Allerdings sind die in Membrane integrierten mPPasen und vPPasen deutlich langsamer.
Sie alle benötigen zu ihrer Funktion Mg2+-Ionen. Diese bilden im Reaktionszentrum die Bindeglieder zwischen negativ geladenen Aminosäuregruppen des Enzyms und den beiden Substraten Pyrophosphat und Wasser. Weil bei den drei Gruppen die Reaktionszentren ganz verschieden strukturiert sind, reagieren sie unterschiedlich sensibel auf selektive Hemmstoffe. Sie werden vom Pyrophosphat-Analogon AMDP (Aminomethylen-Diphosphonat, PO3-CHNH2-PO34-) bei unterschiedlichen Konzentrationen kompetitiv gehemmt, ebenso durch Fluorid, das sich störend an die H2O-Bindungsstelle anlagern kann.[4]
| cPPasen I | cPPasen II | mPPasen und vPPasen | |
|---|---|---|---|
| Reaktionsgeschwindigkeit kcat (s−1) | 200–400 | 1700–3000 | 3,5–20 |
| ki AMDP-Hemmung (µM) | 11–150 | 1000 | 1,2–1,8 |
| ki Fluorid-Hemmung (µM) | 11–90 | 6 | 3000–4800 |
cPPasen der Gruppe I
[Bearbeiten | Quelltext bearbeiten]
Lösliche cPPasen der Gruppe I finden sich in allen drei Domänen der Lebewesen. Dazu zählen die cPPasen von Escherichia coli (EPPase) und der Bäckerhefe Saccharomyces cerevisiae (YPPase). Beide sind seit langem bekannt und intensiv untersucht.[8] Die eukaryotischen cPPasen leiten sich von prokaryotischen Vorgängern ab und sind größer. Die cPPase aus Escherichia coli bildet ein Hexamer (also sechs Protein-Domänen). eukaryotische cPPasen, z. B. die aus menschlichen Zellen und Hefezellen bilden Dimere.[4]
In Abb. 3 sind Bakterien in roter und Archaeen in lila Schrift dargestellt. Inzwischen hat sich gezeigt, dass die cPPasen der Archaeen und der Bakterien zwei getrennte Gruppen bilden[9] Klar davon abgegrenzt finden sich die pflanzlichen (grün) cPPasen, die eng miteinander verwandt sind. Die zu den Opisthokonta gehörenden Tiere (blau) und der Pilz S. cervisiae (cyan) haben eng verwandte cPPasen.[7]
Bemerkenswert ist, dass Mitochondrien eigene cPPasen der Gruppe I besitzen. Das trifft auch auf die mitochondriale PPase von S. cervisiae (rot dargestellt) zu. In der Abbildung nicht dargestellt sind die mitochondriale und cytosolische cPPase der Maus, die auch beide in die Opisthokonta-Gruppe einzuordnen sind.[10] Bei vielen Pflanzen finden sich cPPasen der Gruppe I nicht im Cytosol, sondern in den Mitochondrien und Plastiden. Die Diphosphat-Konzentration im Cytosol kann Werte von 0,2–0,3 mM annehmen und wird dort von vPPasen energetisch genutzt (siehe unten).[11]
| Anorganische cPPase I | ||
|---|---|---|

| ||
| Abb. 4. Bändermodell der cPPase I (Thermococcus litoralis) | ||
| Bezeichner | ||
| Externe IDs |
| |
| Enzymklassifikation | ||
| EC, Kategorie | 3.6.1.1 | |
Das Bakterium Thermus thermophilus besitzt eine für die Polymerase-Kettenreaktion (PCR) geeignete, temperaturbeständige Pyrophosphatase. Bei der PCR entsteht durch die Polymerisation der Desoxyribonukleosidtriphosphate (dNTP) Pyrophosphat, das inhibierend auf den Prozess wirken kann. Durch den Abbau von Pyrophosphat lässt sich die Effizienz und Ausbeute der PCR steigern.
cPPasen der Gruppe II
[Bearbeiten | Quelltext bearbeiten]Die cPPasen der Gruppe II finden sich nur bei Prokaryoten, insbesondere bei den bakteriellen Firmicutes (Bacilli und Clostridien). Die wenigen Vertreter dieser cPPasen außerhalb dieser Gruppe sind wahrscheinlich durch horizontalen Gentransfer zu den Organismen gelangt. Zu ihrer Funktion benötigen sie Mangan2+ (oder Co2+). Diese Enzyme hydrolysieren Pyrophosphat noch im nanomolaren Bereich. Mn2+ bedingt vermutlich ihre höhere Affinität gegenüber ihren Substraten und ihre höhere Umsatzgeschwindigkeit. Man geht davon aus, dass es die Nukleophilie des hydrolysierenden H2O steigert und auch für die größere Sensibilität gegenüber F- verantwortlich ist.[4]
Die cPPase des pathogenen Bakteriums Staphylococcus aureus gehört zur Gruppe II. Sie wird intensiv als mögliches Ziel für neu zu entwickelnde Antibiotika untersucht.[12]
Membrangebundene Energie konservierende mPPasen und vPPasen
[Bearbeiten | Quelltext bearbeiten]Chemiosmotische Funktion
[Bearbeiten | Quelltext bearbeiten]Die Membran gebundenen Pyrophosphatasen (mPPasen und vPPasen) unterscheiden sich nicht nur strukturell, sondern noch mehr funktionell von den im Cytosol gelösten Pyrophosphat hydrolysierenden cPPasen. Sie koppeln die Hydrolyse von PPi an den Export von H+ oder Na+-Kationen aus dem Cytosol durch eine Membran:
- PPi + Kation+innen + H2O ⇌ 2 Pi + Kation+außen
Die exergone Hydrolyse ist an einen endergonen Vorgang gekoppelt, nämlich an den Transport der Ionen gegen ein Membranpotential. Die darin enthaltene potentielle Energie steigt durch die gekoppelte Hydrolyse. Ein Teil der im Pyrophosphat enthaltenen Energie wird also in dem Membranpotential konserviert.
Die membrangebundenen PPasen dienen nicht nur als Ionenpumpen, denn Prozess ist reversibel. Beim Rückstrom der Kationen wird Pyrophosphat produziert.[4] Diese Rückreaktion ist nach Substratkettenphosphorylierung und ATP-Bildung durch die ATP-Synthasen ein dritter Weg zum Aufbau energiereicher Phosphatbindungen.[13]
Vorkommen
[Bearbeiten | Quelltext bearbeiten]Nachgewiesen wurde die Chemiosmotische Kopplung der Pyrophosphat-Synthese an einen Protonengradienten bereits 1966, als Membranvesikel eines phototrophen Bakteriums der Familie der Rhodospirillaceae Phosphat zu Pyrophosphat umsetzten.[14] Eine durch ein Membranpotential ermöglichte Synthese einer energiereichen Phosphatverbindung war damals nur von der ATP-Synthase bekannt, die allerdings eine völlig andere Struktur als die PPase aufweist.[15] Allerdings gibt es bei Rhodospirillum offenbar noch einen funktionellen Unterschied zur ATP-Synthase, denn seine PPase ist primär nicht wie jene in der Zellmembran, sondern in der Membran der Acidocalcisomen (saure Polyphosphat-haltige Organellen[16]) angesiedelt.[17]
Membranständige PPasen sind bei 25 % aller Prokaryoten nachgewiesen. Bei Pilzen und mehrzelligen Tieren scheinen sie dagegen nicht vor zu kommen.[6] Eine wesentliche Rolle spielen sie als vPPasen bei tierischen Einzellern wie dem Krankheitserreger Trypanosoma brucei, einzelligen Algen und insbesondere bei allen Landpflanzen. Sie sind bei Organismen besonders verbreitet, die regelmäßig unter Energie-, Sauerstoffmangel, Salz- und anderen Stressbedingungen leiden.[4]
Evolution
[Bearbeiten | Quelltext bearbeiten]
Die mPPasen und vPPasen haben einen prinzipiell gleichen Aufbau (vgl. Abb. 5) und eine gemeinsame evolutionäre Wurzel, die wohl bis zum Urvorfahr aller Lebewesen zurückreicht. Es wird auch vermutet, dass sie aus einer Phase der chemischen Evolution stammen, als abiotisch entstandenes Pyrophosphat vielleicht die zentrale Rolle als Energieüberträger für die Bildung von Nukleinsäuren spielte.[3][22][23]
Die erste membranständige PPase hat vermutlich Na+ transportiert.[6][24] Das legt die vergleichende Untersuchung der Aminosäurensequenzen dieser Enzyme nahe. Zur selben Vermutung, nämlich dass Na+-Pumpen den H+-Pumpen vorausgingen, kam man auch bei den ATP-Synthasen.[25] Biomembrane sind generell leichter für H+-Ionen zu durchdringen als für Na+, und so erforderte es eine ganze Zeit der Koevolution von Membranproteinen und Membranen, bis eine H+-ATP-Synthase funktionieren konnte.[26][27] Aus den Na+ PPasen haben sich in ihrer weiteren Evolution verschiedene H+-PPasen entwickelt, die zum Teil K+für ihr Funktionieren benötigen.[24]
Molekulare Struktur und Reaktionsmechanismus
[Bearbeiten | Quelltext bearbeiten]Seit 2012 konnte die genaue Struktur einer Na+-mPPase des Bakteriums Thermotoga maritima und einer davon evolutionär weit entfernten pflanzlichen H+-abhängigen vPPase ermittelt werden. Beiden unterscheiden sich im Wesentlichen nicht (Abb. 5). Sie bestehen beide aus zwei funktionell gleichen Proteinketten. Diese durchdringen die Membran in 16 Segmenten. Ihre reaktiven Zentren befinden sich auf der Cytosol-Seite etwa 20 Å entfernt von der Membran in einer hydrophilen Trichter-förmigen Struktur.[4]
In Abb. 5 ist die bakterielle mPPase und die pflanzliche vPPase schematisch dargestellt. In der Frontansicht sind die dimeren Enzyme 85 Å breit und 75 Å hoch.
Das reaktive Zentrum liegt auf Seiten des Cytosols unterhalb der Membran. Beide Enzyme bieten nach der Cytosolseite hin einen hydrophilen Zugang zu dem Enzym und oben einen hydrophilen Kanal (Pfeil), durch den die Kationen die Membran durchdringen können.
Im reaktiven Zentrum sind die Substrate dargestellt. Pyrophosphat ist mit 4 Mg+-Ionen an beide Proteine gekoppelt. Das zur Hydrolyse dienende Wasser ist durch zwei Asparaginsäure-Gruppen an das Enzym assoziiert[5] (in Abb. 5 nicht dargestellt).

In Abb. 6 findet sich ein Arbeitsmodell für die reversible, Ionen transportierende Hydrolyse. Man kann sich den hydrolytischen Weg (in Abb. 6 im Uhrzeigersinn) folgendermaßen vorstellen:
- Der Pump-Mechanismus wird dadurch vorbereitet, dass ein Pyrophosphat-Ion im Austausch gegen ein Hydrogenphosphat-Ion in das Reaktionszentrum eindringt. In der PP-Phase ist das Enzym zur cytosolischen Seite geschlossen, aber der Transmembrankanal ist kurz geöffnet.
- Synchron mit dem Entweichen des Kations nach außen findet die Hydrolyse statt. Dabei bewegt sich ein zunächst an die Asparaginsäure D287 gebundenes H+ an den Ort D294, an den vorher das Kation gebunden war. Die an D273 zurückbleibende OH--Gruppe ist so nukleophil, dass die nukleophile Substitution[28] zu zwei Hydrogenphosphat-Ionen gelingt (2P in der Abbildung).
- In Phase P ist das Enzym nach Austausch des ersten Phosphat-Ions mit einem Hydroxylion OH- unten geöffnet. Es kann ein zu transportierendes Kation eindringen. Danach ist der Zustand wieder hergestellt, in dem das zweite Phosphation gegen Pyrophosphat ersetzt werden kann und ein neuer Zyklus beginnt.[18]
Jeder dieser Schritte ist reversibel. Wenn kaum Pyrophosphat vorhanden ist, steigt die Wahrscheinlichkeit in der P+-Phase, dass das zweite Phosphat nicht ausgetauscht wird. Nach Entweichen des Kations nach innen kann ein zweites Phosphat (im Austausch gegen OH-) in das Enzym eindringen. Eine hohe Kationenkonzentration außen erhöht dann die Chance, dass die Kondensationsreaktion zu Pyrophosphat gelingt.
Die im Vergleich zu den cPPasen langsame Reaktionsgeschwindigkeit ergibt sich aus der Reversibilität der Teilschritte. Die oben erwähnte schwache Fluorid-Hemmung (erst im millimolaren Bereich) liegt vermutlich an der schwachen Bindung des Substrats H2O.[4]
Organische Diphosphatasen
[Bearbeiten | Quelltext bearbeiten]
Als „Pyrophosphatasen“ werden bisweilen auch Hydrolasen bezeichnet, die organische Substrate spalten. Sie folgen in der Regel dem Schema oben in der nebenstehenden Abbildung.
- Nukleotid-Diphosphatasen (EC 3.6.1.9), alias Dinukleotid-Pyrophosphatase spalten Dinukleotide wie beispielsweise FAD. Dieses wird zu FMN und AMP hydrolysiert. In der Abbildung ist dabei R1 = Flavin und R2 = Adenosin. Diese Enzyme sind meist unspezifisch und setzen eine Reihe anderer Coenzyme hydrolytisch außer Funktion, wie z. B. NAD und CoA. Hydrolysiert wird ebenfalls UDP-Glucose.
- Zu den als Pyrophosphatasen bezeichneten Enzymen gehören die Nukleosid-Diphosphatasen (EC 3.6.1.6). Auch sie dienen dem Abbau von Coenzymen. Zu diesen gehört die sogenannte Thiamin-Pyrophosphatase. (R1 = Thiamin, R2 = H.)[29] Eine ADPase, alias Adenosin-Pyrophosphatase (R1 = Adenosin) baut ADP unter Abspaltung von Phosphat zu AMP ab.
- Die nicht mehr IUBMB-konforme Bezeichnung Dihydroneopterin-Pyrophosphatase bezeichnet eine Dihydroneopterin Triphosphat-Diphosphatase EC 3.6.1.67, die anorganisches Pyrophosphat nicht spaltet, sondern freisetzt (Abbildung unten).
Weblinks
[Bearbeiten | Quelltext bearbeiten]- Pyrophosphate Assay Kit (PDF; 253 kB)
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ Laut IUBMB gilt für (anorganische) Pyrophosphatasen die Bezeichnung (inorganic) diphosphatase [1] [2]
- ↑ Jukka K. Heinonen: Biological role of inorganic pyrophosphate. Springer Science & Business Media (vormals Kluwer Academic Publ), Berlin 2001, ISBN 978-1-4615-1433-6, S. 11 (englisch).
- ↑ a b Alexander A. Baykov, Anssi M. Malinen, Heidi H. Luoto, Reijo Lahti: Pyrophosphate-Fueled Na+ and H+ Transport in Prokaryotes. In: Microbiology and Molecular Biology Reviews. Band 77, Nr. 2, 2013, S. 267–276, doi:10.1128/MMBR.00003-13 (englisch).
- ↑ a b c d e f g h Tommi Kajandera, Juho Kellosalob, Adrian Goldman: Inorganic pyrophosphatases: One substrate, three mechanisms. In: FEBS Letters. Band 587, Nr. 13, 2013, S. 1863–1869, doi:10.1016/j.febslet.2013.05.003 (englisch).
- ↑ a b Shih-Ming Lin, Jia-Yin Tsai, Chwan-Deng Hsiao, Yun-Tzu Huang, Chen-Liang Chiu, Mu-Hsuan Liu, Jung-Yu Tung, Tseng-Huang Liu, Rong-Long Pan, Yuh-Ju Sun: Crystal structure of a membrane-embedded H+-translocating pyrophosphatase. In: Nature. Band 484, Nr. 7394, 2012, S. 399–403, doi:10.1038/nature10963 (englisch).
- ↑ a b c Heidi H. Luoto, Georgiy A. Belogurov, Alexander A. Baykov, Reijo Lahti, Anssi M. Malinen: Na+-translocating Membrane Pyrophosphatases Are Widespread in the Microbial World and Evolutionarily Precede H+-translocating Pyrophosphatases. In: Journal of Biological Chemistry. Band 286, Nr. 11, 2011, S. 21633–21642, doi:10.1074/jbc.M111.244483 (englisch).
- ↑ a b Toni Sivula, Anu Salminen, Alexey N. Parfenyev, Pekka Pohjanjoki, Adrian Goldman, Barry S. Cooperman, Alexander A. Baykov, Reijo Lahti: Evolutionary aspects of inorganic pyrophosphatase. In: FEBS Letters. Band 454, Nr. 1–2, 1999, S. 75–80, doi:10.1016/S0014-5793(99)00779-6 (englisch).
- ↑ A. Kunitz: Crystalline inorganic pyrophosphatase isolated from baker's yeast. In: The Journal of General Physiology. Band 35, Nr. 3, 1952, S. 423–450, doi:10.1085/jgp.35.3.423 (englisch).
- ↑ Lana J. McMillana, Nathaniel L. Hepowit, Julie A. Maupin-Furlow: Archaeal Inorganic Pyrophosphatase Displays Robust Activity under High-Salt Conditions and in Organic Solvents. In: Applied and Environmental Microbiology. Band 82, Nr. 2, 2016, S. 538–548, doi:10.1128/AEM.03055-15 (englisch).
- ↑ María R Gómez‐García, Manuel Losada, Aurelio Serrano: Comparative biochemical and functional studies of family I soluble inorganic pyrophosphatases from photosynthetic bacteria. In: FEBS Journal. Band 274, Nr. 15, 2007, S. 3948–3959, doi:10.1111/j.1742-4658.2007.05927.x (englisch).
- ↑ Marco Zancani, Valentino Casolo, Carlo Peresson, Giorgio Federici, Andrea Urbani, Francesco Macrı̀, Angelo Vianello: The β-subunit of pea stem mitochondrial ATP synthase exhibits PPiase activity. In: Mitochondrion. Band 3, Nr. 2, 2003, S. 111–118, doi:10.1016/S1567-7249(03)00105-3 (englisch).
- ↑ Chathurada S. Gajadeera, Xinyi Zhang, Yinan Wei, Oleg V. Tsodikov: Structure of inorganic pyrophosphatase from Staphylococcus aureus reveals conformational flexibility of the active site. In: Journal of Structural Biology. Band 189, Nr. 2, 2015, S. 81–86, doi:10.1016/j.jsb.2014.12.003 (englisch).
- ↑ Laura M. Barge, Ivria J. Doloboff, Michael J. Russell, David VanderVelde, Lauren M. White, Galen D. Stucky, Marc M. Baum, John Zeytounian, Richard Kidd, Isik Kanik: Pyrophosphate synthesis in iron mineral films and membranes simulating prebiotic submarine hydrothermal precipitates. In: Geochimica et Cosmochimica Acta. Band 128, 2014, S. 42705, doi:10.1016/j.gca.2013.12.006 (englisch).
- ↑ Herrick Baltscheffsky, Lars-Victor von Stedingk, Hans-Walter Heldt, Martin Klingenberg: Inorganic Pyrophosphate: Formation in Bacterial Photophosphorylation (Inorganic pyrophosphate is identified as the major product of photophosphorylation by isolated chromatophores from Rhodospirillum rubrum in the absence of added nucleotides.). In: Science. Band 274, Nr. 3740, 1966, S. 1120–1122, JSTOR:1719164 (englisch).
- ↑ Jennifer Moyle, Roy Mitchell, Peter Mitchell: Proton-translocating pyrophosphatase of Rhodospirillum rubrum. In: FEBS Letters. Band 23, Nr. 2, 1972, S. 233–236, doi:10.1016/0014-5793(72)80349-1 (englisch).
- ↑ Roberto Docampo, Wanderley de Souza, Kildare Miranda, Peter Rohloff, Silvia N. J. Moreno: Acidocalcisomes? conserved from bacteria to man. In: Nature Reviews Microbiology. Band 3, Nr. 3, 2005, S. 251–261, doi:10.1038/nrmicro1097 (englisch).
- ↑ Manfredo Seufferheld, Christopher R. Lea, Mauricio Vieira, Eric Oldfield, Roberto Docampo: The H+-pyrophosphatase of Rhodospirillum rubrum is predominantly located in Polyphosphate-rich Acidocalcisomes. In: Journal of Biological Chemistry. Band 279, 2004, S. 51193–51202, doi:10.1074/jbc.M406099200 (englisch).
- ↑ a b c Juho Kellosalo, Tommi Kajander, Konstantin Kogan, Kisun Pokharel, Adrian Goldman: The Structure and Catalytic Cycle of a Sodium-Pumping Pyrophosphatase. In: Science. Band 337, Nr. 6093, 2012, S. 473–476, doi:10.1126/science.1222505 (englisch).
- ↑ [3]
- ↑ Shih-Ming Lin, Jia-Yin Tsai, Chwan-Deng Hsiao, Yun-Tzu Huang, Chen-Liang Chiu, Mu-Hsuan Liu, Jung-Yu Tung, Tseng-Huang Liu, Rong-Long Pan, Yuh-Ju Sun: Crystal structure of a membrane-embedded H+-translocating pyrophosphatase. In: Nature. Band 484, Nr. 7394, 2012, S. 399–403, doi:10.1038/nature10963 (englisch). Grafik vgl. A working model for proton pumping in VrH+-PPase
- ↑ [4]
- ↑ Herrick Baltscheffsky: Origin and evolution of biological energy conversion. John Wiley & Sons, 1996, Energy conversion leading to the origin and early evolution of life: did inorganic pyrophosphate precede adenosine triphosphate? (englisch).
- ↑ Nils G. Holm, Herrick Baltscheffsky: Links between hydrothermal environments, pyrophosphate, Na+, and early evolution. In: Origins of Life and Evolution of Biospheres. Band 41, Nr. 5, 2011, S. 483–493, doi:10.1007/s11084-011-9235-4 (englisch).
- ↑ a b Tommi Kajandera, Juho Kellosalob, Adrian Goldman: Inorganic pyrophosphatases: One substrate, three mechanisms. In: FEBS Letters. Band 587, Nr. 13, 2013, S. 1866 f., doi:10.1016/j.febslet.2013.05.003 (englisch).
- ↑ Armen Y. Mulkidjanian, Pavel Dibrov, Michael Y. Galperin: The past and present of sodium energetics: May the sodium-motive force be with you. In: Biochimica et Biophysica Acta (BBA) – Bioenergetics. Band 1777, Nr. 7–8, 2008, S. 985–992, doi:10.1016/j.bbabio.2008.04.028 (englisch).
- ↑ Armen Y Mulkidjanian, Michael Y Galperin, Kira S Makarova, Yuri I Wolf, Eugene V Koonin: Evolutionary primacy of sodium bioenergetics. In: Biology Direct. Band 3, Nr. 13, 2008, doi:10.1186/1745-6150-3-13 (englisch).
- ↑ Armen Y Mulkidjanian, Kira S Makarova, Michael Y Galperin, Eugene V Koonin: Inventing the dynamo machine: the evolution of the F-type and V-type ATPases. In: Nature Reviews Microbiology. Band 5, Nr. 11, 2007, S. 892–899, doi:10.1038/nrmicro1767 (englisch, dieser Artikel bei Uni Osnabrück: Perspectives ( vom 31. Oktober 2008 im Internet Archive) [PDF]).
- ↑ Suzanne J. Admiraal, Daniel Herschlag: Mapping the transition state for ATP hydrolysis: implications for enzymatic catalysis. In: Chemistry & Biology. Band 2, Nr. 11, 1995, S. 729–739, doi:10.1016/1074-5521(95)90101-9 (englisch).
- ↑ Lucien Bettendorff, Pierre Wins: Thiamin diphosphate in biological chemistry: new aspects of thiamin metabolism, especially triphosphate derivatives acting other than as cofactors. In: FEBS Journal. Band 276, Nr. 11, 2009, S. 2917–2925, doi:10.1111/j.1742-4658.2009.07019.x (englisch).