Aldolase
| Aldolase | ||
|---|---|---|

| ||
| Bänder-/Oberflächenmodell des ALDOA-Tetramers nach PDB 1ALD | ||
| Masse/Länge Primärstruktur | 39.339 Da / 363 Aminosäuren | |
| Sekundär- bis Quartärstruktur | Homotetramer | |
| Isoformen | A, B, C | |
| Bezeichner | ||
| Gen-Name(n) | ALDOA, ALDOB, ALDOC | |
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 4.1.2.13, Lyase | |
| Substrat | Fructose-1,6-bisphosphat | |
| Produkte | Dihydroxyacetonphosphat + D-Glycerinaldehyd-3-phosphat | |
| Vorkommen | ||
| Homologie-Familie | Hovergen | |
| Übergeordnetes Taxon | Lebewesen[1] | |
Aldolase (ausführlich Fructose-1,6-bisphosphat-Aldolase) ist das Enzym, das die Spaltung von Fructose-1,6-bisphosphat (F-1,6-BP) in die Isomere Dihydroxyacetonphosphat (DHAP) und Glycerinaldehyd-3-phosphat (GAP) katalysiert. Diese Reaktion ist ein Teilschritt der Glykolyse und daher unentbehrlich für die Verwertung von Kohlenhydraten in allen Lebewesen. Drei Isoformen des Enzyms sind bei Wirbeltieren bekannt, die in den Muskeln (A), der Leber (A und B), den Erythrozyten (B) sowie im Zentralen Nervensystem (C) lokalisiert sind und von jeweils eigenen Genen kodiert werden. Mutationen an einem dieser Gene können zu Aldolasemangel führen. Fehlt beispielsweise die Aldolase A, kann dies zu Rhabdomyolyse und einer Form der hämolytischen Anämie führen.[2]
Aldolase B wird ihrem Vorkommen wegen auch Leber-Aldolase genannt, allerdings ist sie auch in der Niere präsent. Sie übernimmt gleichzeitig die Funktion einer Fructose-1-phosphat-Aldolase[3] und ist die entwicklungsgeschichtlich älteste Isoform und diejenige, die in Bakterien und Pflanzen gefunden wird.

Eigenschaften
[Bearbeiten | Quelltext bearbeiten]Je nach ihrem Reaktionsmechanismus werden Aldolasen in zwei Klassen unterteilt. Aldolasen der Klasse I bilden ein protoniertes Schiff-Base-Intermediat, das ein hochkonserviertes Lysin des aktiven Zentrums mit dem DHAP-Carbonylkohlenstoff verbindet. Darüber hinaus sind Tyrosinreste für diesen Mechanismus als stabilisierende Wasserstoffakzeptoren von entscheidender Bedeutung.
Klasse-II-Aldolasen verwenden einen anderen Mechanismus, der die Carbonylgruppe mit einem zweiwertigen Kation wie Zn2+ polarisiert. Das Escherichia coli-Galactitol-Operon-Protein gatY und das N-Acetylgalactosamin-Operon-Protein agaY, die beide Tagatose-Bisphosphat-Aldolasen sind, sind Homologe der Aldolase Klasse II. Es wurde gezeigt, dass zwei Histidinreste in der ersten Hälfte der Sequenz dieser Homologen an der Bindung von Zink beteiligt sind.[4]
Die Proteinuntereinheiten beider Klassen haben jeweils eine α/β-Domäne, die in einen TIM-Fass gefaltet ist, der das aktive Zentrum enthält. Mehrere Untereinheiten werden zu dem vollständigen Protein zusammengesetzt. Die beiden Klassen zeigen kaum Sequenzidentität auf.
Mit wenigen Ausnahmen wurden nur Klasse-I-Aldolasen in Tieren, Pflanzen und Grünalgen[5] und mit wenigen Ausnahmen wurden in Pilzen nur Klasse-II-Aldolasen gefunden. Beide Klassen wurden weit verbreitet in anderen Eukaryoten und in Bakterien gefunden. Die beiden Klassen sind oft gemeinsam im selben Organismus vorhanden. Pflanzen und Algen haben neben der üblichen cytosolischen Aldolase auch eine plastidale Aldolase, das manchmal ein Relikt der Endosymbiose ist. Eine bifunktionelle Aldolase-Phosphatase mit Klasse-I-Mechanismus wurde artübergreifend in Archaeen und einigen Bakterien gefunden.[6] Das aktive Zentrum der Aldolase der Archaeen befindet sich ebenfalls in einem TIM-Fass.
Mechanismus der katalysierten Reaktion
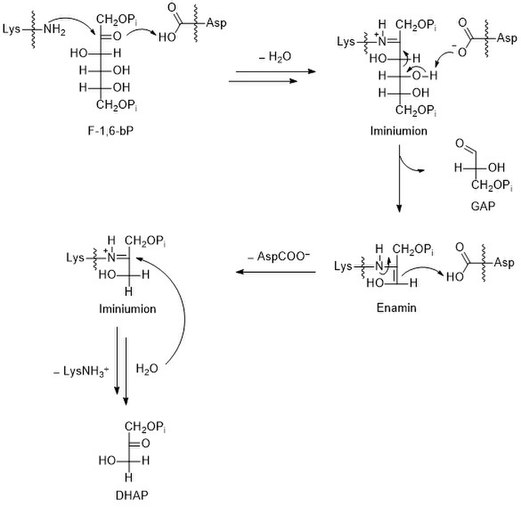
[Bearbeiten | Quelltext bearbeiten]Die durch die Aldolase katalysierte Reaktion entspricht formal einer Aldolspaltung bzw. einer Aldoladdition.[7] Anzumerken ist, dass es sich hierbei um eine Gleichgewichtsreaktion handelt. Für die Beschleunigung der Aldolspaltung von F-1,6-bP wird aus der Carbonylgruppe des Zuckers das carbonylanaloge, aber reaktivere, Iminiumion über eine Kondensation gebildet. Der Protonentransfer durch eine Carboxylatgruppe beschleunigt die Reaktion weiterhin. Für den Abschluss des katalytischen Zyklus erfolgt eine Hydrolyse. Die genauen Mechanismen der Hydrolyse des Iminiumions bzw. der Kondensation der Amino- und der Carbonylgruppe sind nicht dargestellt.
Im aktiven Zentrum der Aldolase liegen die Aminosäureseitenketten von Asparaginsäure und Lysin vor. Die Aminogruppe der Lysinseitenkette greift die Carbonylgruppe von F-1,6-bP nucleophil an. Nach der Eliminierung von Wasser bildet sich ein Iminiumion, was einer Kondensation entspricht. Die Umsetzung der Carbonylgruppe (Kohlenstoff-Doppelbindung-Sauerstoff) zum analogen Iminiumion (Kohlenstoff-Doppelbindung-Stickstoff) folgt der Logik, dass diese stärker elektronenziehend sind als Carbonyle, was die folgende Spaltung der Bindung zwischen C3 und C4 beschleunigt. Die Aldolspaltung führt zur Bildung von GAP und einer Enamin-Zwischenstufe. Diese wird durch die Carboxygruppe der Asparaginsäureseitenkette protoniert und es bildet sich wieder ein Iminiumion. Durch Hydrolyse wird DHAP freigesetzt und die Lysinseitenkette zurückgewonnen. Die Katalyse von Aldoladditionen mit Aminen ist auch aus der Synthesechemie bekannt, beispielsweise in der Hajos-Parrish-Eder-Sauer-Wiechert-Reaktion.
Die Isomerisierung von Glucose-6-phosphat (G6P) zu Fructose-6-phosphat (F6P), früher in der Glykolyse, führt zur Migration der Carbonylgruppe vom C1 zum C2. Die Sinnhaftigkeit dieser Isomerisierung wird in der Aldolase-Reaktion ersichtlich. Eine analoge Aldolspaltung von G6P würde zu einem C2-Körper und einem C4-Körper führen. Die Tatsache, dass jedoch aus F6P (über F-1,6-bP) zwei isomere C3-Körper gewonnen werden, die in einem enzymkatalysierten Gleichgewicht sind, erlaubt die Metabolisierung beider Spaltprodukte über einen linearen Stoffwechselweg. Dies minimiert die Zahl verschiedener Reaktionen und somit die Anzahl an benötigten Enzymen.
Zusammenbau von H+-ATPase
[Bearbeiten | Quelltext bearbeiten]Aldolase B hat eine weitere Funktion, die insbesondere in den Nieren wichtig ist. Dort findet ständig Rückresorption von Blutbestandteilen mittels Endozytose statt. Voraussetzung für die Regulation von Vesikeln und anderen Zellinnenräumen ist deren Ansäuerung mittels V-Typ ATPasen, Transportproteinen in der jeweiligen Membran, die aus mehreren Untereinheiten zusammengebaut werden. Für diesen Zusammenbau ist Aldolase B essentiell; diese Funktion ist von der genannten enzymatischen Funktion völlig unabhängig.[8]
Weblinks
[Bearbeiten | Quelltext bearbeiten]Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ Homologe bei OMA
- ↑ Todd A. Swanson, Sandra I. Kim, Marc J. Glucksman: BRS Biochemistry, Molecular Biology, and Genetics. Lippincott Raven; 5. Auflage 2010, ISBN 978-0-7817-9875-4; S. 65.
- ↑ UniProt P04075
- ↑ S. M. Zgiby, G. J. Thomson, S. Qamar, A. Berry: Exploring substrate binding and discrimination in fructose1, 6-bisphosphate and tagatose 1,6-bisphosphate aldolases. In: European Journal of Biochemistry. Band 267, Nr. 6, März 2000, S. 1858–1868, doi:10.1046/j.1432-1327.2000.01191.x, PMID 10712619.
- ↑ N. J. Patron, M. B. Rogers, P. J. Keeling: Gene replacement of fructose-1,6-bisphosphate aldolase supports the hypothesis of a single photosynthetic ancestor of chromalveolates. In: Eukaryotic Cell. Band 3, Nr. 5, Oktober 2004, S. 1169–1175, doi:10.1128/EC.3.5.1169-1175.2004, PMID 15470245, PMC 522617 (freier Volltext)
- ↑ B. Siebers, H. Brinkmann, C. Dörr, B. Tjaden, H. Lilie, J. van der Oost, C. H. Verhees: Archaeal fructose-1,6-bisphosphate aldolases constitute a new family of archaeal type class I aldolase. In: Journal of Biological Chemistry. Band 276, Nr. 31, August 2001, S. 28710–28718, doi:10.1074/jbc.M103447200, PMID 11387336.
- ↑ Donald Voet, Judith G. Voet, Charlotte W. Pratt: Lehrbuch der Biochemie. 3. Auflage. Wiley-VCH Verlag GmbH & Co. KGaA, 2019, ISBN 978-3-527-34286-0, S. 589.
- ↑ Ming Lu, David Ammar, Harlan Ives, Fred Albrecht, Stephen L. Gluck: Physical Interaction between Aldolase and Vacuolar H+-ATPase Is Essential for the Assembly and Activity of the Proton Pump. In: J. Biol. Chem. 282. Jahrgang, Nr. 34, 2007, S. 24495–24503, doi:10.1074/jbc.M702598200, PMID 17576770.