CRISPR

CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats) sind Abschnitte sich wiederholender DNA (repeats), die im Erbgut vieler Bakterien und Archaeen auftreten. Sie dienen einem Mechanismus, dem CRISPR/Cas-System, der Resistenz gegen das Eindringen fremden Erbguts von Viren oder Plasmiden verschafft, und sind hierdurch ein Teil des Immunsystem-Äquivalents vieler Prokaryoten. Dieses System bildet die Grundlage der gentechnischen CRISPR/Cas-Methode zur Erzeugung gentechnisch veränderter Organismen.
Entdeckung und Eigenschaften[Bearbeiten | Quelltext bearbeiten]
Die Existenz sich wiederholender DNA-Abschnitte, die heute als CRISPR bekannt sind, wurde bereits 1987 im Bakterienstamm Escherichia coli K12 von Yoshizumi Ishino und Kollegen entdeckt. Sie identifizierten eine sich wiederholende Sequenz von 29 Nukleotiden, die von variablen Regionen mit jeweils 32 Nukleotiden unterbrochen wurden.[1] 1993 wurden ähnliche Regionen auch auf der DNA von Mycobacterium tuberculosis entdeckt und als „Direct Variable Repeats“ (DVR) bezeichnet,[2] 1995 erfolgte die Entdeckung dieser Sequenzen auch bei den Meeresbakterien Haloferax volcanii und Haloferax mediterranei durch den spanischen Mikrobiologen Francisco Mojica, der sie als „Tandem Repeats“ (TREPs) bezeichnete.[3] Die Arbeitsgruppe um Mojica identifizierte weitere Bakterien und Archaea mit entsprechenden Sequenzen und wählte für diese sich gleichenden Wiederholungen eine neue Bezeichnung als „Short Regularly Spaced Repeats“ (SRSR).[4] In der Literatur kamen weitere Namen hinzu, die ebenfalls diese Sequenzen bezeichneten, etwa „spacer interspersed and direct repeats“ (SPIDRs) und „long clustered tandem repeats“ (LCTRs).[5] 2002 wurde dann durch Jansen und Kollegen erstmals der Begriff „Clustered Regularly Interspaced Short Palindromic Repeats“, kurz CRISPR, verwendet. Es wurde bekannt, dass ähnliche Strukturen im Genom vieler verschiedener Prokaryoten existieren, und es wurde eine Gruppe von Genen entdeckt, die in allen untersuchten Organismen nahe am Genlokus der CRISPR lagen und daher cas-Gene (CRISPR-associated) genannt wurden.[6] Jansen und Kollegen identifizierten vier verschiedene Cas-Core-Sequenzen (Cas1 bis Cas4), bis 2005 wurden durch Haft und Kollegen insgesamt 41 entsprechende Gene und zwei weitere Cas-Core-Sequenzen (Cas5 und Cas6) und insgesamt acht Subtypen von CRISPR/Cas-Systemen beschrieben.[7][5]
Heute ist bekannt, dass das Genom von etwa 45 % der bislang sequenzierten Bakterien und 83 % der Archaeen mindestens eine CRISPR-Struktur enthält.[8]
Pathogene der Art Francisella verwenden das CRISPR-Cas-System zur Immunevasion.[9] Bei Neisseria meningitidis und Campylobacter jejuni ist das System ein Pathogenitätsfaktor mit bisher unbekanntem Mechanismus.[9]
Struktur[Bearbeiten | Quelltext bearbeiten]

Der CRISPR-Genlocus besteht wesentlich aus zwei Hauptkomponenten: dem cas-Gene enthaltenden cas-Operon und dem CRISPR-Array, der sich aus einer leader-Sequenz und einer Repeat-Spacer-Sequenz (auch Repeat-Spacer-Array genannt) zusammensetzt.[12][13]
Repeat-Spacer-Sequenz[Bearbeiten | Quelltext bearbeiten]
Die Einzelsequenzen des sich wiederholenden Grundmotives (Repeats) haben eine Länge, die zwischen 23 und 47 bp variiert. Die Repeats wechseln sich ab mit Spacern, die eine Länge von 21 bis 72 bp haben. Während innerhalb einer CRISPR-Struktur die sich wiederholende Sequenz erhalten bleibt, variiert die Sequenz der CRISPR in verschiedenen Mikroorganismen stark.[10] Die Sequenz von CRISPR-Repeats der Bakterien ist in der Regel palindromisch (d. h. spiegelverkehrt komplementär), was eine stabile Sekundärstruktur der zugehörigen RNA zur Folge hat, wohingegen die meisten Repeats der Archaeen nicht palindromisch sind.[13]
Die Sequenzen der Spacer-Abschnitte variieren stark, sowohl innerhalb einer CRISPR-Struktur als auch in verschiedenen Prokaryoten. 2005 wurde entdeckt, dass die Spacer-Sequenzen mit Fremd-DNA aus Bakteriophagen und Plasmiden identisch sind.[14][15][16] Dies führte zur Hypothese, dass die Funktion von CRISPR darin besteht, den Organismus gegen Fremd-DNA zu verteidigen.
cas-Operon[Bearbeiten | Quelltext bearbeiten]
Auch zum CRISPR-Genlocus gehörend ist das cas-Operon. Das cas-Operon enthält cas-Gene und die zu codierenden Proteine, die für die adaptive Immunantwort notwendig sind, z. B. Helikasen, Nukleasen, aber auch Proteine mit Eigenschaften zur RNA-Bindung.[13] cas-Gene lassen sich in zwei Module gliedern: dem Effektor- und dem Adaptationsmodul. Unter einem Effektormodul versteht man eine Gruppe von cas-Genen, die zur Identifizierung von genetischem Material dient. Das Adaptationsmodul enthält ebenfalls cas-Gene und trägt mithilfe von Effektorproteinen zur Protospacer-Auswahl bei, die in das bakterielle Genom integriert werden können.[17][18]
leader-Sequenz[Bearbeiten | Quelltext bearbeiten]
In der Nähe der Repeat-Spacer-Sequenz befindet sich eine sogenannte leader-Sequenz (nicht zu verwechseln mit der Leader-Sequenz der mRNA). Die leader-Sequenz ist eine Adenin- und Thymin-reiche Sequenz mit einer Länge von 100–500 bp. Wie bei den Repeats sind leader-Sequenzen innerhalb eines Genoms zu ca. 80 % identisch, aber weisen innerhalb verschiedener Organismen starke Unterschiede auf.[13] Als nichtcodierende Sequenz lässt sich diese in zwei Bereiche aufteilen: einem core leader und einem extended leader. Der core leader ist in mehreren Organismen konserviert und mit einer Länge von 20–300 bp in der Regel kürzer als der extended leader. Außerdem verfügt der core leader über ein Promotorelement, an dem sich Regulatorproteine binden können, um so die Genexpression, genauer die Initiation der CRISPR-Transkription, und die Spacer-Akquirierung kontrollieren zu können.
Der extended leader ist mit einer Länge von 50–500 bp länger als der core leader und enthält ebenfalls in den CRISPR-fernen Regionen konservierte Sequenzen, die vermutlich durch Genduplikation zustande gekommen sind. Die Funktionen des extended leaders sind zurzeit unbekannt. Vermutlich hat der extended leader keine wichtigen Funktionen.[19]
Immunität durch CRISPR[Bearbeiten | Quelltext bearbeiten]

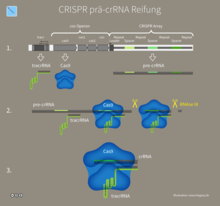
Die Immunität durch CRISPR erfolgt in drei Schritten, wobei die letzten beiden Schritte bei den jeweiligen CRISPR/Cas-Systemtypen unterschiedlich verlaufen.[13]
1. Adaptation: 2007 zeigten Barrangou et al., dass Bakterien, die mit Phagen infiziert werden, Teile der Fremd-DNA als Spacer in die CRISPR-Bereiche ihres Genoms integrieren und hierdurch Immunität gegen die Phagen entwickeln können.[20] Zudem zeigten sie, dass Spacer-Sequenzen, die künstlich in die CRISPR-Bereiche von Bakterien eingefügt werden, diese gegen die zugehörigen Phagen resistent machen. Werden die Spacer-Sequenzen wieder herausgeschnitten, ist auch die Resistenz aufgehoben. Es wurde außerdem gezeigt, dass die cas-Gene eine essentielle Rolle bei der Phagenabwehr spielen: Das Inaktivieren einiger cas-Gene (cas1) verhindert trotz vorhandener Spacer die Abwehr von Phagen. Die Aktivität anderer cas-Gene (cas7) ist notwendig zur Integration neuer Spacer in die CRISPR-Sequenz.
2. crRNA-Transkription und -Prozessierung: Der CRISPR-Genlocus wird zur prä-crRNA transkribiert und anschließend zur reifen crRNA prozessiert.
3. Interferenz: Es kommt zur Assoziation der reifen crRNA mit einem Cas-Protein oder einem Cas-Proteinkomplex und dadurch zur Bildung eines Interferenz-Komplexes. Bei den CRISPR/Cas-Systemtypen I und II kommt es bei Interaktion des Interferenz-Komplexes mit dem Sequenzmotiv PAM der Phagen-DNA zur Degradierung der DNA mithilfe von Cas3 bei Typ I und Cas9 bei Typ II, wohingegen bei Typ III kein PAM benötigt wird und neben DNA auch RNA zersetzt werden kann.
Im Zuge der Koevolution wurden von Bakteriophagen Anti-CRISPR-Proteine zur Hemmung der Abwehr entwickelt.
Mechanismus[Bearbeiten | Quelltext bearbeiten]

Trotz großer Fortschritte in den letzten Jahren wird der Mechanismus, durch den das CRISPR/Cas-System Prokaryoten Immunität verschafft, noch nicht genau verstanden. Man geht davon aus,[10] dass im Immunisierungsprozess die exogene DNA durch einen Cas-Proteinkomplex erkannt und als neuer Spacer in die CRISPR-Bereiche integriert wird. Wie diese Vorgänge im Detail ablaufen, ist derzeit noch nicht vollständig aufgeklärt.
Adaptation[Bearbeiten | Quelltext bearbeiten]
CRISPR/Cas-Systeme sind in der Lage, das Genom von Bakterien und Archaeen zu modifizieren, indem fremde DNA-Sequenzen, sogenannte Spacer, zwischen den Repeats des CRISPR-Arrays integriert werden. Dieser Prozess wird als Adaptation oder Spacer-Akquirierung bezeichnet. Die Adaptation kann in zwei Phasen unterteilt werden:
- Einfangen von Spacer-Sequenzen der fremden DNA (sogenannte Protospacer),
- Spacer-Integration.
Der Mechanismus der Adaptation wurde, mit einigen Ausnahmen, im CRISPR/Cas-System Typ I von E. coli (auch als CRISPR/Cas-System Typ I-E bekannt) im Detail untersucht. Die Hauptakteure der Adaptation werden durch die Gene cas1 und cas2 codiert, die in verschiedenen CRISPR/Cas-Systemtypen konserviert sind.[22]
Die erste Phase der Adaptation, das Einfangen von Spacer-Sequencen der fremden DNA, kann beim CRISPR/Cas-System Typ I in zwei Modi ablaufen: naiv oder primed.[23][24] Bei der naiven Adaptation werden unvoreingenommen zum Einfangen von Spacer nur die Proteine Cas1 und Cas2 benötigt,[25] wohingegen die primed adaptation von bereits existierenden Spacern (priming spacer) abhängt und somit eine Vorauswahl getroffen wird, welche Spacer in das Genom integriert werden. Neben den Proteinen Cas1 und Cas2 wird hierfür weiterhin ein Proteinkomplex, der sich aus Cas-Proteinen zusammensetzt (Interferenzkomplex Typ I, Cascade) und die Cas3-Nuklease benötigt.[23][26] Andere CRISPR/Cas-Systemtypen codieren zusätzliche Proteine zur Adaptation.
Der Mechanismus der primed adaption beginnt mit der Bindung des crRNA-gebundenen Proteinkomplexes Cascade (CRISPR-associated complex for antiviral defense) an den Protospacer Adjacent Motif (PAM) der eindringenden DNA mittels einer Kombination aus erleichterter 1D-Diffusion (Gleiten entlang der DNA) und 3D-Diffusion (hopping).[27] Nach der Beugung und Entwindung der DNA durch Cascade kommt es durch komplementäre Basenpaarung der crRNA und dem Cascade-gebundenen DNA-Strang zur Bildung eines R-Loops.[28] Durch vollständige Entwindung des Protospacers durch Cascade bildet sich der R-Loop vollständig aus. Durch die vollständige Bildung des R-Loops kommt es zur Konformationsänderung von Cascade und bewirkt somit eine Bindung von Cas3 an Cascade. Außerdem wird durch die vollständige Bildung des R-Loops eine Aufwölbung am nicht-gebundenen Strang ausgelöst und dadurch der Schnitt durch Cas3 an dieser Aufwölbung ermöglicht.[29] Die durch Cas3 erzeugten, einzelsträngigen Fragmente werden anschließend durch den Cas1-Cas2-Komplex zu einzelsträngige Protospacer verarbeitet. Nach Verarbeitung zum einzelsträngigen Protospacer erfolgt die Umwandlung zum vollständigen oder partiell-doppelsträngigen Protospacer, sodass eine Integration in das CRISPR-Array möglich wird.[30] Auch nach dem letzten Schritt der Immunität durch CRISPR, der Interferenz, ist das Einfangen von Spacer-Sequenzen möglich. Dabei werden die Fragmente der degradierten DNA durch das Enzym RecBCD oder andere Nukleasen zu Protospacer umgewandelt und mithilfe des Cas1-Cas2-Komplexes in das CRISPR-Array integriert (naive Adaptation).[27]
Die Spacer-Integration erfolgt nicht willkürlich im CRISPR-Array, sondern verläuft polarisiert, d. h., dass Spacer an gezielter Stelle im CRISPR-Array integriert werden, genauer in der Nähe der leader-Sequenz. Dieser Mechanismus stellt sicher, dass neue Spacer immer in der Nähe der leader-Sequenz integriert werden und durch die chronologische Integration der Spacer die adaptive Immunantwort gegenüber den jüngsten viralen Infektionen optimiert wird.[31] Beim CRISPR/Cas-System Typ I wird dafür das Protein Integration Host Factor (IHF) benötigt, das sich an der leader-Sequenz binden kann. Dadurch wird die leader-Sequenz um ca. 120° gebeugt und erzeugt eine Bindungsstelle für den Cas1-Cas2-Komplex, sodass sich der Komplex in der Nähe desjenigen Repeats befindet, der zur leader-Sequenz am nächsten lokalisiert ist. Dadurch wird die leader-Repeat-Grenze zum Ort der Spacer-Integration.[32] Beim CRISPR/Cas-System Typ II verläuft die Spacer-Integration ebenfalls polarisiert ab,[20] jedoch ohne Einsatz von zusätzlichen Proteinen. Dabei bindet sich die α-Helix von Cas1 des Cas1-Cas2-Komplexes Typ II an der kleinen Furche der leader-Sequenz, der auch als leader-anchoring sequence (LAS) bezeichnet wird. Aufgrund der Flexibilität der LAS-interagierenden Domäne von Cas1 muss die Spacer-Integration nicht unbedingt an der leader-Repeat-Grenze, sondern kann auch an einer Spacer-Repeat-Grenze stattfinden.[33] Bei einer mutierten LAS kann dies zu einer ektopischen Spacer-Integration führen, wobei Spacer in der Mitte des CRISPR-Arrays integriert werden.[34]
Bei E. coli erfolgt die Spacer-Integration durch zwei Umesterungen, wobei die erste Umesterung durch den nukleophilen Angriff der Hydroxygruppe am 3′-Ende des einen Stranges des Protospacers an der leader-Repeat-Grenze erfolgt und dadurch zur Bildung eines half-site-Integrationsintermediats führt.[35] Die erste Umesterung erzeugt eine Beugung des Repeats, das eine zweite Umesterung ermöglicht. Der Übergang zum vollständig integriertem Spacer, dem full-site-Produkt, geht durch eine zweite Umesterung vonstatten, wobei der nukleophile Angriff der Hydroxygruppe am 3′-Ende des gegenüberliegenden Stranges des Protospacers in der Nähe der Repeat-Spacer-Grenze erfolgt.[36] Die zweite Umesterung wird durch einen sogenannten Ruler-Mechanismus reguliert.[36] Bei E. coli beinhaltet der Repeat zwei inverse Repeats (IR), die für Strukturmotive codieren und als Anker für sogenannte „molekulare Lineale“ dienen. Diese molekularen Lineale sorgen dafür, dass der zweite nukleophile Angriff nur in der Nähe der Repeat-Spacer-Grenze stattfindet und die Länge des Repeats nach erfolgter Spacer-Integration und Repeat-Duplikation aufrechterhalten wird.[37] Die nach den Umesterungen erzeugten DNA-Lücken werden durch verschiedene DNA-Reparaturmechanismen geschlossen, dazu gehören homology-directed repair (HDR), non-homologous end joining (NHEJ) und microhomology-mediated end joining (MMEJ). Nach erfolgter Spacer-Integration wurde der an der leader-Sequenz angrenzende Repeat mit gleicher Länge dupliziert.[38]
crRNA-Transkription und -Prozessierung[Bearbeiten | Quelltext bearbeiten]
Die Biogenese einer reifen CRISPR-RNA (crRNA) kann in drei Schritten erfolgen und führt mithilfe seiner partiell einzigartigen Spacer-Sequenz ein oder mehrere Cas-Proteine zur eindringenden Nukleinsäure, das zur eventuellen Degradierung des genetischen Materials nach sequenzspezifischer RNA-Erkennung dient.
- Transkription eines langen primären Transkripts, der Präkursor-crRNA (prä-crRNA), durch einen Promotor, der sich innerhalb der leader-Sequenz befindet.
- Primäre Spaltung der prä-crRNA an spezifischen Stellen zur Erzeugung von crRNA mit einer gesamten Spacer-Sequenz mit partiellen Repeat-Sequenzen.
- In einigen Fällen wird eine zusätzliche sekundäre Spaltung benötigt, um eine aktive reife crRNA zu generieren.
In den CRISPR/Cas-Systemen I und III wird eine spezifische Endoribonuklease der Cas6-Familie oder alternativ Cas5d bei Typ I-C benötigt, die allein oder im Komplex mit anderen Cas-Proteinen die prä-crRNA innerhalb der Repeat-Regionen spaltet. Bei Typ II transaktiviert eine tracrRNA die Spaltung der prä-crRNA innerhalb der Repeat-Regionen durch die Endoribonuklease III (RNase III) in Anwesenheit von Cas9.[12]
Beim CRISPR/Cas-System Typ I wird die Prozessierung der prä-crRNA durch Endoribonukleasen der metallunabhängigen Cas6-Familie (oder alternativ beim Typ I-C durch Cas5d) katalysiert, welche die Repeat-Sequenz an konservierten Positionen, typischerweise 8 nt upstream („strangaufwärts“, in Richtung des 5′-Endes) von der Repeat-Spacer-Grenze, spaltet.[28][39] Während für die Typen I-C, I-E und I-F nur ein Reifungsschritt benötigt wird,[40] sind für die Typen I-A, I-B und I-D ein zweiter Reifungsschritt nötig, dessen Komponenten und Mechanismus derzeit noch unbekannt sind. Die palindromischen Repeats der prä-crRNA der Typen I-C, I-D, I-E und I-F besitzen Haarnadelstrukturen, die die Spaltungsstellen für die katalytische Domäne der jeweiligen Endoribonuklease freilegen. Nach der Spaltung bleiben die Haarnadelstrukturen an der jeweiligen Endoribonuklease assoziiert und die Untereinheiten von Cascade binden sich an der Sequenz am 5′-Ende und am Spacer, die zur Erkennung von genetischem Material verwendet werden.[12]
Beim CRISPR/Cas-System Typ II erfolgt nach Transkription des CRISPR-Arrays und von tracrRNA eine Basenpaarung des Anti-Repeats von tracrRNA mit dem Repeat der prä-crRNA und zur Bildung des tracrRNA:prä-crRNA-Duplex, der durch Cas9 stabilisiert wird. Außerdem führt die Duplex-Bildung zur Rekrutierung der RNase III und somit zur Co-Prozessierung des Duplex.[41] Darauf folgt der zweite Reifungsschritt, wobei es zum trimming durch eine Exonuklease und/oder zur Spaltung durch eine Endoribonuklease kommt. Typ II-C stellt einen alternativen Syntheseweg einer reifen crRNA dar. Dabei befinden sich die Promotoren innerhalb der Repeats des CRISPR-Arrays und es kann zur Bildung eines kurzen prä-crRNA-Transkripts kommen, sodass die Spaltung durch die RNase III nicht mehr nötig ist.[42] Der reife Duplex ist mit Cas9 komplexiert und bildet einen Interferenzkomplex Typ II, der doppelsträngige DNA (dsDNA) erkennen und spalten kann.[43]
Beim CRISPR/Cas-System Typ III erfolgt die Spaltung der prä-crRNA innerhalb der Repeats durch Cas6 und erzeugt somit crRNA-Intermediate, die an ihren 5′- und 3′-Enden jeweils eine partielle Sequenz der Repeats der prä-crRNA besitzen (1X-Intermediate).[39][44] Danach kommt es bei III-A zur Komplexierung des 1X-Intermediats mit dem Csm-Komplex und bei III-B mit dem Cmr-Komplex. Anschließend erfolgt der zweite Reifungsschritt mittels trimming am 3′-Ende durch Nukleasen, die noch nicht identifiziert werden konnten, zur reifen crRNA.[12]
Interferenz[Bearbeiten | Quelltext bearbeiten]
Die nach der Prozessierung der prä-crRNA zur reifen crRNA, welche die integrierten viralen Spacer-Sequenzen enthalten, assoziieren sich mit einem CRISPR-Ribonukleoprotein-Komplex (crRNP) und bilden einen Interferenzkomplex (auch als CRISPR-Surveillance-Komplex bekannt), mit dem nach einer weiteren Infektion die virale DNA oder RNA sequenzspezifisch degradiert werden kann.[28] Der Interferenz-Mechanismus ist bei allen CRISPR/Cas-Systemtypen durch bestimmte Schlüsselproteine gekennzeichnet: Cas 3 (Typ I), Cas 9 (Typ II) und Cas10 (Typ III) und unterscheiden sich hauptsächlich im Zusammenbau des crRNP-Komplexes (crRNP-Assemblierung) und im Degradierungsmechanismus des genetischen Materials. Sämtliche crRNP-Komplexe in Typ I werden als Cascade bezeichnet, wohingegen bei Typ II das Protein Cas9 als einzelnes Protein für die Spaltung der Nukleinsäure verantwortlich ist. Bei Typ III sind die crRNP-Komplexe Csm (Typ III-A) und Cmr (Typ III-B) für die Interferenz zuständig.[45]
Die Interferenz erfolgt beim CRISPR/Cas-System Typ I in fünf Schritten:

- Cascade-Assemblierung
- PAM-Erkennung und -Bindung
- R-Loop-Bildung
- Cas3-Rekrutierung
- DNA-Degradierung
Nach der Prozessierung der prä-crRNA besteht die reife crRNA von E. coli aus einem 5′-handle (8 nt) mit einer Hydroxygruppe, einer Spacer-Sequenz (32 nt) und einer Haarnadelstruktur am 3′-Ende (21 nt) mit einem 2′-3′-cyclischem Phosphatende,[40] wobei Cas6e nach der Prozessierung an der Haarnadelstruktur assoziiert bleibt. Nach Spaltung der reifen crRNA erfolgt die Cascade-Assemblierung, wobei der erste Schritt das sogenannte termini capping ist. Dabei bindet sich Cas5 am 5′-handle und erzeugt somit zunächst eine hakenähnliche Struktur der crRNA. Des Weiteren binden sich sechs Kopien des Proteins Cas7 an die Spacer-Sequenz und daraus ergibt sich das sogenannte Cas7-Backbone.[28] Das Besondere ist, dass die Strukturen von Cas5 und Cas7 eine sogenannte konservierte „Handfläche-Daumen-Domäne“ aufweisen, die zur Verflechtung des Cas7-Backbones beitragen.[46] Der „Daumen“ (eine β-Haarnadelstruktur) von entweder Cas5e oder von jedem der sechs Cas7-Untereinheiten (Cas7.1–Cas7.6) knickt die crRNA am 5′-handle an einer bestimmten Position und in 6-nt-Abständen innerhalb der Spacer-Sequenz und sorgt dafür, dass die geknickten Nukleotide eine deformierte Konfiguration annehmen und nicht mehr zur Basenpaarung mit der Ziel-DNA geeignet sind. Dahingegen ragen die angrenzenden 5-nt-Sequenzen bei jedem Knick heraus und behalten ihre diskontinuierliche A-DNA-ähnliche-Form, sodass diese Sequenzen zur Basenpaarung mit der Ziel-DNA geeignet sind.[47] Anschließend binden sich zwei weitere Proteine, Cse1 (große Untereinheit) und das Cse2-Dimer (kleine Untereinheiten), mittels Protein-Protein-Interaktion an die Cas7-Untereinheiten. Beide Proteine sind an der DNA-Bindung beteiligt, wobei die große Untereinheit außerdem zur Ziel-Auswahl beiträgt.[40] Damit wird sichergestellt, dass der Interferenzkomplex die Zelle jederzeit nach potentieller Ziel-DNA absucht.[46] Nach abschließender Assemblierung wird Cascade oftmals als Seepferdchen-ähnliche Struktur beschrieben.[48]
Nun erfolgt mithilfe von Cascade die Suche nach der Ziel-DNA, wobei die L1-Schleife von Cse1 zur PAM-Identifikation zuständig ist. Bei Typ I-E tritt nach PAM-Identifikation die doppelsträngige virale DNA in die Lücke zwischen Cas7.5 und Cas7.6 ein und wird anschließend zur großen Untereinheit (Cse1) weitergeleitet, die jedoch hauptsächlich nicht-spezifische Interaktionen mit der Ziel-DNA aufweist.[49] Die PAM-Erkennung durch die L1-Schleife von Cse1 bewirkt eine Destabilisierung der doppelsträngigen DNA, sodass zunächst die Basenpaarung zwischen der 7 nt langen seed-Region der PAM-angrenzenden DNA-Protospacersequenz mit der crRNA erfolgen kann. Die anschließende Bildung eines R-Loops bei vollständiger Basenpaarung des crRNA-Spacers mit dem viralen Protospacer erfolgt nach demselben Mechanismus wie beim Einfangen von Spacer-Sequenzen. Nach vollständiger R-Loop-Bildung kommt es zur Konformationsänderung der großen und kleinen Untereinheit, sodass Interaktionsstellen an der großen Untereinheit für die C-terminale Domäne (CTD) von Cas3 geschaffen werden.[50] Durch die Rekrutierung von Cas3 an der Gabelung öffnet sich der Kanal für die doppelsträngige DNA durch Dissoziation der CTD. Nach der Anlagerung der dsDNA im Kanal wird der Kanal durch Repositionierung der CTD geschlossen und der nicht-gebundene Strang der doppelsträngigen DNA in die HD-Nuklease-Domäne von Cas3 eingelagert, wo der Schnitt erfolgt.[51][52] Der Schnitt erfolgt ungefähr 11–15 nt downstream („strangabwärts“, in Richtung des 3′-Endes) vom PAM mithilfe von zwei katalytischen Übergangsmetall-Ionen.[53] Die durch den Schnitt ausgelöste Konformationsänderung von Cas3 im Helikaseteil (bestehend aus der RecA-ähnlichen Domäne (RecA) und der RecA-ähnlichen Domäne 2 (RecA2)) bewirkt eine ATP-Bindung und -Hydrolyse,[54] deren freigesetzte Energie zur Entwindung der dsDNA in 3′→5′-Richtung genutzt wird. Die Entwindung erfolgt an einer Haarnadelstruktur von RecA2. Durch die Bewegung des Helikaseteils löst dies eine Verlagerung der HD-Domäne an neue Substrate zur weiteren exonukleolytischen Degradierung aus.[51][52] Die nach der Degradierung gebildeten einzelsträngigen DNA (ssDNA) werden ebenfalls durch Cas3 exonukleolytisch degradiert.[53][54] Somit kann die Ziel-DNA effektiv von der Zelle entfernt werden und Cascade zur weiteren PAM-Erkennung recycelt werden.
Beim CRISPR/Cas-System Typ II erfolgt die Interferenz in vier Schritten:

- Bildung des aktiven Typ-II-CRISPR-Surveillance-Komplexes
- PAM-Erkennung und -Bindung
- R-Loop-Bildung
- DNA-Degradierung
Drei unabhängige Studien zur Struktur von Cas9 von S. pyogenes weisen auf, dass Cas9 aus zwei Lappen besteht, die zusammen eine Mondsichel-Konformation einnehmen.[55][56][57] Der REC-Lappen (engl. recognition lobe) besteht aus einer langen α-Helix (Brückenhelix), einer Rec2-Domäne und einer Rec1-Domäne zur Erkennung des tracrRNA:crRNA-Duplex. Der NUC-Lappen (engl. nuclease lobe) besteht aus zwei Nuklease-Domänen zur DNA-Spaltung, die als HNH (benannt nach charakteristischen Histidin- und Asparaginresten) und RuvC (benannt nach einem E. coli-Protein, das an der DNA-Reparatur beteiligt ist) bekannt sind,[58] und einer zusätzlichen C-terminalen Topoisomerase-Homologie-Domäne (CTD), die zur Erleichterung der PAM-Erkennung notwendig ist.
Die vor der Interferenz stattgefundene Aktivierung von Cas9 durch Bindung des Duplex an Rec1 löste eine Konformationsänderung von HNH aus, die zur Positionsänderung vom REC-Lappen und zur Bildung eines zentralen positiv geladenen Kanals für die eindringende DNA führte.[56][57] Der nach der Co-Prozessierung des Duplex gebildete aktive Typ-II-CRISPR-Surveillance-Komplex ist nun bereit zur Suche nach einer viralen DNA mit einer PAM-Sequenz. Nach der PAM-Bindung kommt es zum lokalen Schmelzen (engl. local melting) der DNA.[59] Dabei werden ungepaarte Nukleinbasen, sogenannte geschmolzene Blasen (engl. melted bubbles)[60] gebildet, die zur R-Loop-Bildung an einer PAM-proximalen 8–12 nt langen seed-Sequenz der DNA beitragen. Anschließend spaltete jede Nuklease-Domäne einen DNA-Strang in Anwesenheit von Mg2+-Ionen, wobei die HNH-Domäne den an die crRNA hybridisierten Ziel-DNA-Strang und die RuvC-Domäne den nicht-hybridisierten DNA-Strang spaltet.[58] Der daraus resultierende Schnitt, der etwa 3 nt strangaufwärts vom PAM erfolgt, führt zur Bildung von Doppelstrangbrüchen mit Blunt Ends (engl. für „glattes Ende“).[61] Danach bleibt Cas9 fest an den Blunt Ends der viralen DNA assoziiert.[59]
Bei CRISPR/Cas-Systemen Typ III erkennt der Interferenz-Komplex das entstehende RNA-Transkript, welches komplementär zur Sequenz des crRNA-Spacers ist, und degradiert sowohl das Transkript als auch die DNA, aus der das Transkript hervorgegangen ist. Dieser Prozess wird als transkriptionsabhängige DNA-Interferenz bezeichnet.[62] Der Interferenz-Komplex besitzt drei enzymatische Aktivitäten:[63]
- crRNA-gesteuerte Endoribonuklease-Aktivität gegen die Ziel-RNA durch Csm3 (Typ III-A) oder Cmr4 (Typ III-B)
- Ziel-RNA-stimulierte DNase-Aktivität durch die HD-Domäne von Cas10 (Csm1 bei Typ III-A und III-D oder Cmr2 bei Typ III-B und III-C)
- Ziel-RNA-stimulierte cOA (cyclisches Oligoadenylat)-Synthetase-Aktivität durch die „Handflächen-Domäne“ von Cas10 (Csm1 bei Typ III-A und III-D oder Cmr2 bei Typ III-B und III-C)
In Bakterien werden die crRNA-gesteuerten Komplexe Csm (Typ III-A) oder Cmr (Typ III-B) zum RNA-Transkript gebracht, welches die Spaltung des Transkripts durch die Untereinheiten Csm3 oder Cmr4 auslöst und gleichzeitig die DNase-Aktivität von Csm1 oder Cmr2 zur gekoppelten Degradierung von ssDNA in der Transkriptionsblase aktiviert. Die „Handflächen-Domäne“, genauer die Cyclase-Domäne, von Csm1 oder Cmr2 kann cOA aus ATP bei Bindung des RNA-Transkripts herstellen. cOA wiederum bindet und aktiviert die Ribonuklease Csm6 (Typ III-A) oder Csx1 (Typ III-B, III-C und III-D)[64] zur Verstärkung ihrer Ribonuklease-Aktivität, um RNA-Transkripte zu degradieren und bildet somit einen zusätzlichen Interferenz-Mechanismus.[63]
Auswirkungen[Bearbeiten | Quelltext bearbeiten]
Durch den CRISPR/Cas-Mechanismus können Bakterien Immunität gegen bestimmte Phagen erwerben und die so erworbene Immunität weitervererben, da sie einen virusspezifischen Spacer in ihr Genom integrieren und somit bei der Replikation weitergeben. Aus diesem Grund wurde auch die provokante These geäußert, dass es sich beim CRISPR-Cas-System um den ersten wirklich lamarckistischen Vererbungsmechanismus handele.[65]
Anwendungen[Bearbeiten | Quelltext bearbeiten]
Es gibt mehrere Vorschläge, CRISPR biotechnologisch zu nutzen:[66]
- Künstliche Immunisierung gegen Phagen durch Hinzufügen passender Spacer bei industriell wichtigen Bakterien, z. B. in der Milch- oder Weinindustrie,
- Knockdown endogener Gene durch Transformation mit einem Plasmid, das einen CRISPR-Bereich beinhaltet, mit crRNA, die zu dem stillzulegenden Gen passt,
- Multiplex Genome Editing erlaubt das gleichzeitige Mutieren verschiedener Zielsequenzen, was die Herstellungszeit transgener Tiere wie Mäuse von bis zu zwei Jahren auf wenige Wochen verkürzt,[67]
- Unterscheidung verschiedener Bakterienstämme durch Vergleich der Spacer-Regionen (spoligotyping),
- Gentherapie,
- Fluoreszenzmarkierung.
Literatur[Bearbeiten | Quelltext bearbeiten]
- Martin Jinek, Krzysztof Chylinski, Ines Fonfara, Michael Hauer, Jennifer Doudna, Emmanuelle Charpentier: A Programmable Dual-RNA–Guided DNA Endonuclease in Adaptive Bacterial Immunity. (PDF; 2,4 MB). In: Science. Vol. 337, Nr. 6096, 17. August 2012, S. 816 ff. ISSN 0036-8075. (englisch)
Einzelnachweise[Bearbeiten | Quelltext bearbeiten]
- ↑ Y. Ishino, H. Shinagawa K. Makino M. Amemura A. Nakata A: Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. In: Journal of Bacteriology. Band 169, 1987, S. 5429–5433.
- ↑ P. M. Groenen, A. E. Bunschoten, D. van Soolingen, J. D. van Embden: Nature of DNA polymorphism in the direct repeat cluster of Mycobacterium tuberculosis; application for strain differentiation by a novel typing method. In: Molecular Microbiolology. Band 10, 1993, S. 1057–1065.
- ↑ F. J. Mojica, C. Ferrer, G. Juez, F. Rodriguez-Valera: Long stretches of short tandem repeats are present in the largest replicons of the Archaea Haloferax mediterranei and Haloferax volcanii and could be involved in replicon partitioning. In: Molecular Microbiology. Band 17, 1995, S. 85–93.
- ↑ F. J. Mojica, C. Diez-Villasenor, E. Soria, G. Juez: Biological significance of a family of regularly spaced repeats in the genomes of Archaea, Bacteria and mitochondria. In: Molecular Microbiology. Band 36, 2000, S. 244–246.
- ↑ a b Sinan Al-Attar, Edze R. Westra, John van der Oos, Stan J.J. Brouns: Clustered regularly interspaced short palindromic repeats (CRISPRs): the hallmark of an ingenious antiviral defense mechanism in prokaryotes. In: Biological Chemistry. Band 392, 2011, S. 277–289. doi:10.1515/BC.2011.042, (Volltext)
- ↑ R. Jansen, J. D. Embden, W. Gaastra, L. M. Schouls: Identification of genes that are associated with DNA repeats in prokaryotes. In: Mol Microbiol. Band 43, 2002, S. 1565–1575.
- ↑ D. H. Haft, J. Selengut, E. F. Mongodin, K. E. Nelson: A guild of 45 CRISPR-associated (Cas) protein families and multiple CRISPR/Cas subtypes exist in prokaryotic genomes. In: PLoS Comput. Biol. 1, 2005; e60.
- ↑ 125/150 Archaeen, 1126/2480 Bakterien CRISPRdb (Memento vom 16. Mai 2015 im Internet Archive), Stand: 19. Januar 2013.
- ↑ a b T. R. Sampson, D. S. Weiss: Alternative roles for CRISPR/Cas systems in bacterial pathogenesis. In: PLoS pathogens. Band 9, Nummer 10, 2013, S. e1003621, ISSN 1553-7374. doi:10.1371/journal.ppat.1003621. PMID 24146613. PMC 3798603 (freier Volltext).
- ↑ a b c A. Horvath, R. Barrangou: CRISPR/Cas, the Immune System of Bacteria and Archaea. In: Science. Band 327, 2010, S. 167.
- ↑ L. A. Marraffini, E. J. Sontheimer: CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea. In: Nature Reviews Genetics. Band 11, Nummer 3, März 2010, S. 181–190, doi:10.1038/nrg2749. PMID 20125085, PMC 2928866 (freier Volltext) (Review).
- ↑ a b c d E. Charpentier, H. Richter, J. van der Oost, M. F. White: Biogenesis pathways of RNA guides in archaeal and bacterial CRISPR-Cas adaptive immunity. In: FEMS microbiology reviews. Band 39, Nummer 3, Mai 2015, S. 428–441, doi:10.1093/femsre/fuv023. PMID 25994611, PMC 5965381 (freier Volltext) (Review).
- ↑ a b c d e CRISPR Locus. In: Sino Biological. Abgerufen am 11. Januar 2020.
- ↑ F. J. Mojica, C. Diez-Villasenor, J. Garcia-Martinez, E. Soria: Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. In: Journal of Molecular Evolution. Band 60, 2005, S. 174–182.
- ↑ C. Pourcel, G. Salvignol, G. Vergnaud: CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. In: Microbiology. Band 151, 2005, S. 653–663.
- ↑ A. Bolotin, B. Quinquis, A. Sorokin, S. D. Ehrlich: Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin. In: Microbiology. Band 151, 2005, S. 2551–2561.
- ↑ Donghyun Ka, Dong Man Jang, Byung Woo Han, Euiyoung Bae: Molecular organization of the type II-A CRISPR adaptation module and its interaction with Cas9 via Csn2. In: Nucleic Acids Research. 46, 2018, S. 9805, doi:10.1093/nar/gky702.
- ↑ Eugene V. Koonin: CRISPR: a new principle of genome engineering linked to conceptual shifts in evolutionary biology. In: Biology & Philosophy. Band 34, 2019, doi:10.1007/s10539-018-9658-7.
- ↑ Omer S. Alkhnbashi, Shiraz A. Shah, Roger A. Garrett, Sita J. Saunders, Fabrizio Costa, Rolf Backofen: Characterizing leader sequences of CRISPR loci. In: Bioinformatics. Band 32, Nummer 17, 1. September 2016, S. i576–i585, doi:10.1093/bioinformatics/btw454.
- ↑ a b R. Barrangou, C. Fremaux, H. Deveau, M. Richards, P. Boyaval, S. Moineau, D. A. Romero, A. Horvath: CRISPR Provides Acquired Resistance Against Viruses in Prokaryotes. In: Science. Band 315, 2007, S. 1709–1712.
- ↑ Jennifer Doudna, Emmanuelle Charpentier.: The new frontier of genome engineering with CRISPR-Cas9. In: Science. 346(6213), 2014, S. 1077. PMID 25430774, doi:10.1126/science.1258096.
- ↑ CRISPR Spacer Acquisition. In: Sino Biological. Abgerufen am 16. Januar 2020.
- ↑ a b Kirill A. Datsenko, Ksenia Pougach, Anton Tikhonov, Barry L. Wanner, Konstantin Severinov, Ekaterina Semenova: Molecular memory of prior infections activates the CRISPR/Cas adaptive bacterial immunity system. In: Nature Communications. Band 3, 2012, doi:10.1038/ncomms1937.
- ↑ Daan C. Swarts, Cas Mosterd, Mark W. J. van Passel, Stan J. J. Brouns, Igor Mokrousov: CRISPR Interference Directs Strand Specific Spacer Acquisition. In: PLoS ONE. Band 7, 2012, S. e35888, doi:10.1371/journal.pone.0035888.
- ↑ Ido Yosef, Moran G. Goren, Udi Qimron: Proteins and DNA elements essential for the CRISPR adaptation process in Escherichia coli. In: Nucleic Acids Research. Band 40, 2012, S. 5569, doi:10.1093/nar/gks216.
- ↑ P. C. Fineran, M. J. H. Gerritzen, M. Suarez-Diez, T. Kunne, J. Boekhorst, Sacha A. F. T. van Hijum, Raymond H. J. Staals, Stan J. J. Brouns: Degenerate target sites mediate rapid primed CRISPR adaptation. In: Proceedings of the National Academy of Sciences. Band 111, Nummer 16, 2014, S. E1629–E1638, doi:10.1073/pnas.1400071111.
- ↑ a b Kaylee E. Dillard, Maxwell W. Brown, Nicole V. Johnson, Yibei Xiao, Adam Dolan, Erik Hernandez, Samuel D. Dahlhauser, Yoori Kim, Logan R. Myler, Eric V. Anslyn, Ailong Ke, Ilya J. Finkelstein: Assembly and Translocation of a CRISPR-Cas Primed Acquisition Complex. In: Cell. Band 175, 2018, S. 934, doi:10.1016/j.cell.2018.09.039.
- ↑ a b c d S. J. J. Brouns, M. M. Jore, M. Lundgren, E. R. Westra, R. J. H. Slijkhuis, A. P. L. Snijders, M. J. Dickman, K. S. Makarova, E. V. Koonin, J. van der Oost: Small CRISPR RNAs Guide Antiviral Defense in Prokaryotes. In: Science. Band 321, 2008, S. 960.
- ↑ Yibei Xiao, Min Luo, Robert P. Hayes, Jonathan Kim, Sherwin Ng, Fang Ding, Maofu Liao, Ailong Ke: Structure Basis for Directional R-loop Formation and Substrate Handover Mechanisms in Type I CRISPR-Cas System. In: Cell. Band 170, 2017, S. 48, doi:10.1016/j.cell.2017.06.012.
- ↑ Olga Musharova, Evgeny Klimuk, Kirill A. Datsenko, Anastasia Metlitskaya, Maria Logacheva, Ekaterina Semenova, Konstantin Severinov, Ekaterina Savitskaya: Spacer-length DNA intermediates are associated with Cas1 in cells undergoing primed CRISPR adaptation. In: Nucleic Acids Research. Band 45, 2017, S. 3297, doi:10.1093/nar/gkx097.
- ↑ R. Sorek, C. M. Lawrence, B. Wiedenheft: CRISPR-mediated adaptive immune systems in bacteria and archaea. In: Annual review of biochemistry. Band 82, 2013, S. 237–266, doi:10.1146/annurev-biochem-072911-172315. PMID 23495939 (Review).
- ↑ K. N. Yoganand, R. Sivathanu, S. Nimkar, B. Anand: Asymmetric positioning of Cas1-2 complex and Integration Host Factor induced DNA bending guide the unidirectional homing of protospacer in CRISPR-Cas type I-E system. In: Nucleic acids research. Band 45, Nummer 1, Januar 2017, S. 367–381, doi:10.1093/nar/gkw1151. PMID 27899566, PMC 5224486 (freier Volltext).
- ↑ Y. Xiao, S. Ng, K. H. Nam, A. Ke: How type II CRISPR-Cas establish immunity through Cas1-Cas2-mediated spacer integration. In: Nature. Band 550, Nummer 7674, 10 2017, S. 137–141, doi:10.1038/nature24020. PMID 28869593, PMC 5832332 (freier Volltext).
- ↑ J. McGinn, L. A. Marraffini: CRISPR-Cas Systems Optimize Their Immune Response by Specifying the Site of Spacer Integration. In: Molecular cell. Band 64, Nummer 3, 11 2016, S. 616–623, doi:10.1016/j.molcel.2016.08.038. PMID 27618488, PMC 5096952 (freier Volltext).
- ↑ J. K. Nuñez, A. S. Lee, A. Engelman, J. A. Doudna: Integrase-mediated spacer acquisition during CRISPR-Cas adaptive immunity. In: Nature. Band 519, Nummer 7542, März 2015, S. 193–198, doi:10.1038/nature14237. PMID 25707795, PMC 4359072 (freier Volltext).
- ↑ a b Jenny G Kim, Sandra Garrett, Yunzhou Wei, Brenton R. Graveley, Michael P. Terns: CRISPR DNA elements controlling site-specific spacer integration and proper repeat length by a Type II CRISPR–Cas system. In: Nucleic Acids Research. Band 47, 2019, S. 8632, doi:10.1093/nar/gkz677.
- ↑ M. G. Goren, S. Doron, R. Globus, G. Amitai, R. Sorek, U. Qimron: Repeat Size Determination by Two Molecular Rulers in the Type I-E CRISPR Array. In: Cell Reports. Band 16, Nummer 11, September 2016, S. 2811–2818, doi:10.1016/j.celrep.2016.08.043. PMID 27626652, PMC 5039180 (freier Volltext).
- ↑ S. H. Sternberg, H. Richter, E. Charpentier, U. Qimron: Adaptation in CRISPR-Cas Systems. In: Molecular cell. Band 61, Nummer 6, März 2016, S. 797–808, doi:10.1016/j.molcel.2016.01.030. PMID 26949040 (Review).
- ↑ a b J. Carte, R. Wang, H. Li, R. M. Terns, M. P. Terns: Cas6 is an endoribonuclease that generates guide RNAs for invader defense in prokaryotes. In: Genes & development. Band 22, Nummer 24, Dezember 2008, S. 3489–3496, doi:10.1101/gad.1742908. PMID 19141480, PMC 2607076 (freier Volltext).
- ↑ a b c M. M. Jore, M. Lundgren, E. van Duijn, J. B. Bultema, E. R. Westra, S. P. Waghmare, B. Wiedenheft, U. Pul, R. Wurm, R. Wagner, M. R. Beijer, A. Barendregt, K. Zhou, A. P. Snijders, M. J. Dickman, J. A. Doudna, E. J. Boekema, A. J. Heck, J. van der Oost, S. J. Brouns: Structural basis for CRISPR RNA-guided DNA recognition by Cascade. In: Nature structural & molecular biology. Band 18, Nummer 5, Mai 2011, S. 529–536, doi:10.1038/nsmb.2019. PMID 21460843.
- ↑ E. Deltcheva, K. Chylinski, C. M. Sharma, K. Gonzales, Y. Chao, Z. A. Pirzada, M. R. Eckert, J. Vogel, E. Charpentier: CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. In: Nature. Band 471, Nummer 7340, März 2011, S. 602–607, doi:10.1038/nature09886. PMID 21455174, PMC 3070239 (freier Volltext).
- ↑ Y. Zhang, N. Heidrich, B. J. Ampattu, C. W. Gunderson, H. S. Seifert, C. Schoen, J. Vogel, E. J. Sontheimer: Processing-independent CRISPR RNAs limit natural transformation in Neisseria meningitidis. In: Molecular cell. Band 50, Nummer 4, Mai 2013, S. 488–503, doi:10.1016/j.molcel.2013.05.001. PMID 23706818, PMC 3694421 (freier Volltext).
- ↑ M. Jinek, K. Chylinski, I. Fonfara, M. Hauer, J. A. Doudna, E. Charpentier: A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. In: Science. Band 337, Nummer 6096, August 2012, S. 816–821, doi:10.1126/science.1225829. PMID 22745249, PMC 6286148 (freier Volltext).
- ↑ J. Carte, N. T. Pfister, M. M. Compton, R. M. Terns, M. P. Terns: Binding and cleavage of CRISPR RNA by Cas6. In: RNA. Band 16, Nummer 11, November 2010, S. 2181–2188, doi:10.1261/rna.2230110. PMID 20884784, PMC 2957057 (freier Volltext).
- ↑ K. S. Makarova, D. H. Haft, R. Barrangou, S. J. Brouns, E. Charpentier, P. Horvath, S. Moineau, F. J. Mojica, Y. I. Wolf, A. F. Yakunin, J. van der Oost, E. V. Koonin: Evolution and classification of the CRISPR-Cas systems. In: Nature reviews. Microbiology. Band 9, Nummer 6, 06 2011, S. 467–477, doi:10.1038/nrmicro2577. PMID 21552286, PMC 3380444 (freier Volltext) (Review).
- ↑ a b A. Plagens, H. Richter, E. Charpentier, L. Randau: DNA and RNA interference mechanisms by CRISPR-Cas surveillance complexes. In: FEMS microbiology reviews. Band 39, Nummer 3, Mai 2015, S. 442–463, doi:10.1093/femsre/fuv019. PMID 25934119, PMC 5965380 (freier Volltext) (Review).
- ↑ R. N. Jackson, S. M. Golden, P. B. van Erp, J. Carter, E. R. Westra, S. J. Brouns, J. van der Oost, T. C. Terwilliger, R. J. Read, B. Wiedenheft: Structural biology. Crystal structure of the CRISPR RNA-guided surveillance complex from Escherichia coli. In: Science. Band 345, Nummer 6203, September 2014, S. 1473–1479, doi:10.1126/science.1256328. PMID 25103409, PMC 4188430 (freier Volltext).
- ↑ B. Wiedenheft, G. C. Lander, K. Zhou, M. M. Jore, S. J. Brouns, J. van der Oost, J. A. Doudna, E. Nogales: Structures of the RNA-guided surveillance complex from a bacterial immune system. In: Nature. Band 477, Nummer 7365, September 2011, S. 486–489, doi:10.1038/nature10402. PMID 21938068, PMC 4165517 (freier Volltext).
- ↑ D. G. Sashital, B. Wiedenheft, J. A. Doudna: Mechanism of foreign DNA selection in a bacterial adaptive immune system. In: Molecular cell. Band 46, Nummer 5, Juni 2012, S. 606–615, doi:10.1016/j.molcel.2012.03.020. PMID 22521690, PMC 3397241 (freier Volltext).
- ↑ E. R. Westra, P. B. van Erp, T. Künne, S. P. Wong, R. H. Staals, C. L. Seegers, S. Bollen, M. M. Jore, E. Semenova, K. Severinov, W. M. de Vos, R. T. Dame, R. de Vries, S. J. Brouns, J. van der Oost: CRISPR immunity relies on the consecutive binding and degradation of negatively supercoiled invader DNA by Cascade and Cas3. In: Molecular cell. Band 46, Nummer 5, Juni 2012, S. 595–605, doi:10.1016/j.molcel.2012.03.018. PMID 22521689, PMC 3372689 (freier Volltext).
- ↑ a b B. Gong, M. Shin, J. Sun, C. H. Jung, E. L. Bolt, J. van der Oost, J. S. Kim: Molecular insights into DNA interference by CRISPR-associated nuclease-helicase Cas3. In: Proceedings of the National Academy of Sciences. Band 111, Nummer 46, November 2014, S. 16359–16364, doi:10.1073/pnas.1410806111. PMID 25368186, PMC 4246338 (freier Volltext).
- ↑ a b Y. Huo, K. H. Nam, F. Ding, H. Lee, L. Wu, Y. Xiao, M. D. Farchione, S. Zhou, K. Rajashankar, I. Kurinov, R. Zhang, A. Ke: Structures of CRISPR Cas3 offer mechanistic insights into Cascade-activated DNA unwinding and degradation. In: Nature structural & molecular biology. Band 21, Nummer 9, September 2014, S. 771–777, doi:10.1038/nsmb.2875. PMID 25132177, PMC 4156918 (freier Volltext).
- ↑ a b S. Mulepati, S. Bailey: In vitro reconstitution of an Escherichia coli RNA-guided immune system reveals unidirectional, ATP-dependent degradation of DNA target. In: Journal of Biological Chemistry. Band 288, Nummer 31, August 2013, S. 22184–22192, doi:10.1074/jbc.M113.472233. PMID 23760266, PMC 3829311 (freier Volltext).
- ↑ a b T. Sinkunas, G. Gasiunas, C. Fremaux, R. Barrangou, P. Horvath, V. Siksnys: Cas3 is a single-stranded DNA nuclease and ATP-dependent helicase in the CRISPR/Cas immune system. In: The EMBO Journal. Band 30, Nummer 7, April 2011, S. 1335–1342, doi:10.1038/emboj.2011.41. PMID 21343909, PMC 3094125 (freier Volltext).
- ↑ M. Jinek, F. Jiang, D. W. Taylor, S. H. Sternberg, E. Kaya, E. Ma, C. Anders, M. Hauer, K. Zhou, S. Lin, M. Kaplan, A. T. Iavarone, E. Charpentier, E. Nogales, J. A. Doudna: Structures of Cas9 endonucleases reveal RNA-mediated conformational activation. In: Science. Band 343, Nummer 6176, März 2014, S. 1247997, doi:10.1126/science.1247997. PMID 24505130, PMC 4184034 (freier Volltext).
- ↑ a b C. Anders, O. Niewoehner, A. Duerst, M. Jinek: Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease. In: Nature. Band 513, Nummer 7519, September 2014, S. 569–573, doi:10.1038/nature13579. PMID 25079318, PMC 4176945 (freier Volltext).
- ↑ a b H. Nishimasu, F. A. Ran, P. D. Hsu, S. Konermann, S. I. Shehata, N. Dohmae, R. Ishitani, F. Zhang, O. Nureki: Crystal structure of Cas9 in complex with guide RNA and target DNA. In: Cell. Band 156, Nummer 5, Februar 2014, S. 935–949, doi:10.1016/j.cell.2014.02.001. PMID 24529477, PMC 4139937 (freier Volltext).
- ↑ a b Elitsa Y. Dimova, Thomas Kietzmann: Genomveränderungen–CRISPR/Cas9 als Methode der Wahl oder Qual? In: BIOspektrum. 24, 2018, S. 702, doi:10.1007/s12268-018-0977-7.
- ↑ a b S. H. Sternberg, S. Redding, M. Jinek, E. C. Greene, J. A. Doudna: DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. In: Nature. Band 507, Nummer 7490, März 2014, S. 62–67, doi:10.1038/nature13011. PMID 24476820, PMC 4106473 (freier Volltext).
- ↑ R. A. Forties, R. Bundschuh, M. G. Poirier: The flexibility of locally melted DNA. In: Nucleic acids research. Band 37, Nummer 14, August 2009, S. 4580–4586, doi:10.1093/nar/gkp442. PMID 19487242, PMC 2724272 (freier Volltext).
- ↑ R. Sapranauskas, G. Gasiunas, C. Fremaux, R. Barrangou, P. Horvath, V. Siksnys: The Streptococcus thermophilus CRISPR/Cas system provides immunity in Escherichia coli. In: Nucleic acids research. Band 39, Nummer 21, November 2011, S. 9275–9282, doi:10.1093/nar/gkr606. PMID 21813460, PMC 3241640 (freier Volltext).
- ↑ S. Silas, P. Lucas-Elio, S. A. Jackson, A. Aroca-Crevillén, L. L. Hansen, P. C. Fineran, A. Z. Fire, A. Sánchez-Amat: Type III CRISPR-Cas systems can provide redundancy to counteract viral escape from type I systems. In: eLife. Band 6, 08 2017, S. , doi:10.7554/eLife.27601. PMID 28826484, PMC 5576922 (freier Volltext).
- ↑ a b Type Ⅲ CRISPR-Cas Systems. In: Sino Biological. Abgerufen am 14. Februar 2019.
- ↑ Rafael Molina, Stefano Stella, Mingxia Feng, Nicholas Sofos, Vykintas Jauniskis, Irina Pozdnyakova, Blanca López-Méndez, Qunxin She, Guillermo Montoya: Structure of Csx1-cOA4 complex reveals the basis of RNA decay in Type III-B CRISPR-Cas. In: Nature Communications. 10, 2019, doi:10.1038/s41467-019-12244-z.
- ↑ E. V. Kooni, Y. I. Wolf: Is evolution Darwinian or/and Lamarckian? In: Biology Direct. Band 4, 2009, S. 42.
- ↑ R. Sorek, V. Kunin, P. Hugenholtz: CRISPR – a widespread system that provides acquired resistance against phages in bacteria and archaea. In: Nat Rev Microbiol. Band 6, 2007, S. 181.
- ↑ H. Wang, H. Yang, C. S. Shivalila, M. M. Dawlaty, A. W. Cheng, F. Zhang, R. Jaenisch One-Step Generation of Mice Carrying Mutations in Multiple Genes by CRISPR/Cas-Mediated Genome Engineering. In: Cell. Volume 153, Issue 4, 9 May 2013, S. 910–918.