Bakteriophagen

Als Bakteriophagen oder kurz Phagen (Singular Phage, der; von altgriechisch βακτήριον baktérion ‚Stäbchen‘ und φαγεῖν phageín ‚fressen‘) bezeichnet man herkömmlicherweise verschiedene Gruppen von Viren, die auf Bakterien als Wirtszellen spezialisiert sind, d. h. Bakterienviren.[3] Herkömmlicherweise werden die Bakterienviren (Bakteriophagen) entsprechend ihrer Wirtsspezifität in verschiedene Gruppen klassifiziert, zum Beispiel in Coli-, Staphylokokken-, Diphtherie- oder Salmonella-Bakteriophagen (oder -viren). Mit einer geschätzten Anzahl von 1030 Virionen im gesamten Meerwasser sind Bakterienviren häufiger als jede Art zellulärer Lebewesen und bilden zusammen mit Viren der Archaeen und Protisten (mikrobiellen Eukaryoten) das sogenannte Virioplankton; zu ihnen zählen insbesondere viele Cyanophagen (Viren der Cyanobakterien).
Traditionell wurden (und werden) auch die Viren der Archaeen (Archaeenviren, en. archaeal viruses, archaeoviruses)[4] gelegentlich noch als Phagen oder Bakteriophagen bezeichnet, was eine Reminiszenz an die frühen 1970er Jahre ist, als Archaeen noch nicht von Bakterien unterschieden wurden. Seinerzeit wurden besonders Viren mit charakteristischer Kopf-Schwanz-Struktur (Caudoviren, heute Klasse Caudoviricetes) erforscht. Diese infizieren zum Teil Bakterien und zum Teil Archaeen, und man bezeichnete sie kurz als „Phagen“. Zudem wurden die Archaeen anfänglich noch als „Archaebakterien“ bezeichnet, weshalb die Archaeenviren sich zunächst weiterhin als „Bakteriophagen“ bezeichnen ließen.
Viren (d. h. die Viruspartikel) besitzen keinen eigenen Stoffwechsel, sondern „leihen“ diesen und Teile des Replikationsmechanimus von ihren Wirten „aus“, um sich genetisch mittels ihrer DNS (oder RNS) zu vermehren („replizieren“). Sie werden daher (von den meisten Autoren) nicht als Lebewesen (im eigentlichen Sinne) angesehen, aber von einigen Wissenschaftlern als „dem Leben nahe“ bezeichnet.[5] Die meisten Bakterienviren (insbesondere die Caudoviren) besitzen, wie zelluläre Organismen, ein dsDNA-Genom (linear oder zirkulär), es gibt aber auch Beispiele für andere Genomorganisationen (ssDNA, und RNA).
Geschichte
[Bearbeiten | Quelltext bearbeiten]Die Wirkung von Bakterienviren („Phagen“) wurde im Jahr 1917 von dem Frankokanadier Félix Hubert d’Hérelle erstmals beschrieben.[6] Zwar hatte der Engländer Frederick Twort bereits zwei Jahre zuvor an Staphylokokken-Kulturen Zersetzungsprozesse beobachtet, die auf die Einwirkung von Bakteriophagen zurückzuführen sind, jedoch wurde seine Veröffentlichung praktisch nicht beachtet. D’Hérelle gilt somit neben Frederick Twort als einer der Entdecker der Bakteriophagen, den sogenannten „Bakterienfressern“. Ihren Namen verdanken sie d’Hérelle. Parallel zu d’Hérelle postulierte der deutsche Mikrobiologe Philalethes Kuhn aufgrund von Beobachtungen der Veränderungen von Bakterienkulturen unter bestimmten Bedingungen die Existenz von Bakterienparasiten. Er bezeichnete diese als Pettenkoferien und sah die von d’Hérelle beschriebene „unsichtbare, dem Ruhrbazillus entgegenwirkende Mikrobe“ als Sonderfall dieser Parasiten an. Wie sich später herausstellte, beruhten seine Beobachtungen jedoch nicht auf der Existenz eines Bakterienparasiten, sondern lediglich auf Formveränderungen der von ihm untersuchten Bakterien.
D’Hérelle stellte sich einen Bakteriophagen als ein „ultravisibles, korpuskulares Lebewesen“ vor, das in einer Grundform existiere und sich an verschiedene Wirte – also Bakterien – anpasse. Tatsächlich sind Bakteriophagen nach heutigem Wissensstand hochspezialisierte Viren, die an einen spezifischen Wirt gebunden sind. Auch wenn in diesem Kontext von Wirten die Rede ist, sind nach heutiger Definition Bakteriophagen, da sie als Viren keine Lebewesen sind, keine Parasiten.[7] Die ersten Phagen, die untersucht wurden, waren sieben Phagen des Bakteriums Escherichia coli. Sie wurden von Max Delbrück in der Reihenfolge ihrer Entdeckung als Typ (englisch Type) 1 (T1), Typ 2 (T2) und so weiter benannt. Die aktuelle taxonomische Einordnung dieser Phagenstämme nach ICTV mit Spezies und Familie ist wie folgt:
- T1: Escherichia-Phage T1: Spezies Tunavirus T1 (Escherichia-Virus T1), Drexlerviridae, Siphoviren
- T2: Escherichia-Phage T2: Spezies Tequatrovirus T2 (Escherichia-Virus T2), Straboviridae, Myoviren
- T3: Enterobacteria phage T3: Spezies Teetrevirus T3 (Enterobacteria-Virus T3), Autographiviridae, Podoviren
- T4: Escherichia-Phage T4: Spezies Tequatrovirus T4 (Escherichia-Virus T4), Straboviridae, Myoviren
- T5: Escherichia-Phage T5: Spezies Tequintavirus T5 (Escherichia-Virus T5), Demerecviridae, Siphoviren
- T6: Enterobacteria-Phage T6: Spezies Tequatrovirus T6 (Enterobacteria-Virus T6), Straboviridae, Myoviren
- T7: Escherichia-Phage T7: Spezies Teseptimavirus T7 (Escherichia-Virus T7), Autographiviridae, Podoviren
Diese sieben Escherichia-Phagen (echte Bakterienviren) werden manchmal unter der Sammelbezeichnung „T-Phagen“ (englisch T phages) zusammengefasst,[8][9] was aber keine Verwandtschaftsgruppe (Taxon) darstellt. Stattdessen werden diese Viren vom ICTV (mit Stand Januar 2021) nach einigen Verschiebungen den oben angegebenen Familien zugeordnet. Lediglich die Vertreter mit gerader Typ-Nummer (T-even phages mit T2, T4 – Referenz – und T6) erwiesen sich zufällig als näher miteinander verwandt, so dass für diese vom ICTV zunächst eine gemeinsame Spezies als Taxon eingerichtet wurde (inzwischen aber aufgetrennt). Die Typen mit ungerader Nummer (T-odd/T-uneven phages) bilden sowieso kein Taxon. Allerdings ist allen diesen Phagentypen ein Kopf-Schwanz-Aufbau gemeinsam, weshalb sie früher in einer morphologisch begründeten Ordnung Caudovirales zusammengefasst wurden, welche die drei Morphotypen Myo-, Sipho- und Podoviren zunächst jeweils als taxonomische Familien umfasste. Diese ursprüngliche Ordnung wurde inzwischen aufgrund der genomischen Diversität zur Klasse Caudoviricetes hochgestuft. Von anderen Autoren wurde die Gepflogenheit bei der Benennung anderer Caudoviren teilweise weitergeführt (z. B. „T12“, Vorschlag, ohne Familienzuordnung)[10].
Aufbau
[Bearbeiten | Quelltext bearbeiten]
Die Gestalt der Bakterienviren mit Kopf-Schwanz-Struktur (Caudoviren, Klasse Caudoviricetes) wurde vorwiegend an den Bakteriophagen der T-Reihe (T-Serie) von Escherichia coli aufgeklärt. Der Bakteriophage T2 besteht aus einem polyedrischen Kopf von 100 nm Länge, an dem ein etwa gleich langer Schwanz sitzt. Viren werden taxonomisch in erster Linie nach ihrem Genom-Aufbau, und nachrangig nach ihrer Morphologie und ihrem Wirt eingeteilt. So unterscheidet man DNA-Viren mit einzelsträngiger DNA, sogenannte ssDNA-Viren (von engl. single-stranded), und solche mit doppelsträngiger DNA, sogenannte dsDNA-Viren (von engl. double-stranded). Die hier exemplarisch behandelten Escherichia coli-Viren der T-Reihe werden zu letzterer Gruppe gezählt.
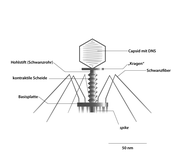
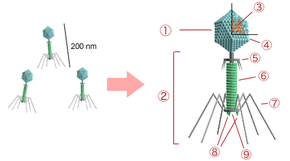
Die oben bereits erwähnten T-Phagen (wie z. B. die Gattung Tequattrovirus mit der Spezies Escherichia-Virus T4) zeichnen sich zusammen mit anderen Mitgliedern der Klasse Caudoviricetes gegenüber sonstigen Bakteriophagen durch einen relativ komplexen Aufbau mit „Kopf-Schwanz-Struktur“ aus: Grundlegend setzen sie sich aus einer Grundplatte (9), einem Einspritzapparat (Injektionsapparat oder Schwanz, 2) und einem Kopf (1), bestehend aus dem so genannten Kapsid (4) und der darin enthaltenen Nukleinsäure (Genom, 3), zusammen. Die Module Kopf und Einspritzapparat/Schwanz sind durch einen Hals (Collar, 5) verbunden. Die Grundplatte (die wie Kapsid und Injektionsapparat aus Proteinen aufgebaut ist) ist mit Schwanzfibern (7) und Spikes (8) besetzt, die der Adsorption auf der Wirtszellwand dienen. Der Injektionsapparat besteht aus einem dünnen Rohr (Schwanzrohr, 6), durch das die Phagen-Nukleinsäure (3) in die Wirtszelle injiziert wird. Das Rohr wird von einer kontraktilen Schwanzscheide umhüllt, die sich während der Injektion zusammenzieht. Das Kapsid ist mit ikosaedrischer Symmetrie aus 152 Kapsomeren aufgebaut und enthält die DNA des Phagen. Aufgrund dieses Aufbaus zählen die Phagen der Gattung Tequattrovirus (Morphotyp Myoviren) zu den strukturell komplexesten Viren.

Phagen mit einzelsträngiger DNA sind dagegen meist klein, sphärisch und schwanzlos (Microviridae) oder filamentös (Tubulavirales). Die ebenfalls auftretenden RNA-Phagen bestehen meist (soweit bis zu diesem Zeitpunkt beschrieben) aus einer Proteinhülle, die ein einsträngiges RNA-Molekül umschließt. Der Durchmesser dieser Phagen beträgt etwa 25 nm, sie gehören also zu den kleinsten Phagen.
- Verschiedene Phagen
-
Phage T2 (Straboviridae, Myoviren); Kapsid axial geschnitten
-
Synechococcus-Phage S-PM2 (Kyanoviridae, Myoviren) aus Meerwasser
Vermehrung
[Bearbeiten | Quelltext bearbeiten]
Viren benötigen mangels eines eigenen Stoffwechsels zur Reproduktion einen Wirt, im Falle der Bakteriophagen eine geeignete, lebende Bakterienzelle. Die Reproduktion lässt sich in fünf Phasen gliedern:
- Adsorption an spezifische Zellwandrezeptoren: Bei der Adsorption koppeln die Enden der Schwanzfasern an passende Moleküle (Rezeptoren) der Oberfläche des Bakteriums.
- Injektion der Phagen-Nukleinsäure in die Wirtszelle: Die phageneigene Nukleinsäure, DNA bzw. RNA, gelangt in das Bakterium. Die nun funktionslosen Proteine der leeren Phagenhülle bleiben außen auf der Oberfläche des Bakteriums zurück.
- Latenzphase: Während dieser Phase lassen sich im Bakterium keine Phagen nachweisen. Nun beginnt die Transkription des Virusgenoms, die Translation der viralen mRNA und die Replikation der Virusnukleinsäure. Dieser Vorgang dauert maximal einige Stunden.
- Produktionsphase: Nachdem die Phagengene in einer festgelegten Reihenfolge aktiv geworden sind, werden alle Virusbestandteile, Hüllproteine und Schwanzfasern, gebildet.
- Reifephase: In dieser Phase der Morphogenese erfolgt der Zusammenbau (assembly) zu reifen Phagenpartikeln. Zunächst wird ein Kopfteil, das Kapsid, gebildet. Die Proteine im Innern dienen als Platzhalter und werden später durch die Phagen-Nukleinsäure, die in das Kapsid eindringt, ersetzt. Dabei nehmen die Nukleinsäure-Fäden, gleich einem Wollknäuel, eine platzsparende Form an.
- Freisetzung: Die fertigen Viruspartikel werden durch enzymatische Auflösung der Wirtszelle befreit. Das Lysozym, welches von dem umprogrammierten Bakterium gebildet wurde, löst die bakterielle Mureinzellwand auf. Die Zelle platzt, und etwa 200 infektiöse Phagen werden frei.
Die Vermehrung verläuft bei einigen Phagenarten nicht immer nach dem oben beschriebenen, lytischen Schema ab. Bei temperenten Phagen unterscheidet man zwischen lysogenen und lytischen Vermehrungszyklen beziehungsweise Infektionszyklen. Bei einem lysogenen Zyklus wird die DNA des Phagen in das Chromosom des Bakteriums eingebaut, wodurch ein Prophage entsteht. Bei jeder folgenden Zellteilung werden die Gene des Phagen und die des Bakteriums gemeinsam verdoppelt und weitergegeben. Dieser Zyklus kann später in den lytischen Zyklus münden.
Riesenphagen
[Bearbeiten | Quelltext bearbeiten]
Doppelstrang-DNA-Phagen mit einer Genomgröße von mehr als 540 kbp werden als Megaphagen bezeichnet, kleinere mit mehr als 200 kbp als Jumbo-Phagen.[12][13] Die Autoren hatten 2018/2019 Fäkalien von Menschen in Bangladesch und Tansania sowie von Pavianen in Afrika und Schweinen in Dänemark untersucht. Die Proben enthielten Bakterien der Gattung Prevotella (Prevotellaceae), die von einer Reihe von dsDNA-Megaphagen infiziert waren, die von den Autoren „Lak-Phagen“[14] (nach dem Subdistrikt – bengalisch Upazila – Laksam, Bangladesch) genannt wurden. Die gefundenen Phagen wurden (vorläufig) als Lak-A1, Lak-A2, Lak-B1 bis Lak-B9 und Lak-C1 bezeichnet. Es könnte eine lose phylogenetische Beziehung zum „Sphingomonas-Phagen PAU“[15][16] (dieser Riesenphage infiziert Bakterien der Spezies Sphingomonas paucimobilis, Sphingomonadaceae) vom Morphotyp der Myoviren bestehen. Die Autoren kommen zu dem Schluss, dass „Lak-Phagen“ „weit verbreitete, aber bisher übersehene Mitglieder des Darm-Mikrobioms“ sind.[12][17][18][19]
Im Februar 2020 veröffentlichten Basem Al-Shayeb und Kollegen eine Analyse, die diese Untersuchungen fortführt.[20] Darin ziehen sie die Grenze für Megaphagen bei 500 kbp (was offenbar Basenpaare im doppelsträngigen Fall und Basen oder Nukleotide in einzelsträngigen Fall bedeutet). Die Autoren ziehen es aber vor, alle Phagen mit mehr als 200 kbp (also Jumbo-Phagen und Megaphagen) als „englisch huge phages“ (hier mit Riesenphagen übersetzt) zusammengefasst zu betrachten. Die Autoren identifizierten unter dieser Gruppe eine Reihe von zehn Kladen, für die sie folgende Namen vorschlugen: „Kabirphage“, „Mahaphage“ (darunter die Gruppe der „Lak-Phagen“), „Biggiephage“ (nicht zu verwechseln mit der vorgeschlagenen Spezies Biggie virus[21]), „Dakhmphage“, „Kyodaiphage“, „Kaempephage“, „Jabbarphage“, „Enormephage“, „Judaphage“ und „Whopperphage“ (alle Namen beziehen sich auf „riesig“ oder engl. „huge“ in den verschiedenen Sprachen der Autoren).[22] Durch ihre Metagenomanalysen verschiedener Proben konnten sie 351 dsDNA-Phagensequenzen identifizieren, davon nur drei unter 200 kbp. Das größte Genom hatte eine Länge von 735 kbp (ein Mahaphage, was offenbar neuer Rekord ist; der vorherige lag bei 596 kbp); gewöhnliche Nicht-Riesenphagen haben im Mittel lediglich 52 kbp. Einige Riesenphagen scheinen einen vom Standard abweichenden genetischen Code zu benutzen, in dem das Stop-Codon UAG für eine Aminosäure kodiert. Die Wirte sind (meist) Bakterien der Firmicutes oder der Proteobacteria, aber auch – so bei den Mitgliedern der Mahaphage-Gruppe mit den „Lak-Phagen“ – der Bacteroidetes. Das Genom kodiert neben den phageüblichen Proteinen für tRNAs. Die Phagen interagieren darüber hinaus im CRISPR/Cas-System (siehe CRISPR, CRISPR/Cas-Methode, Genom-Editierung): Alle bedeutenden Typen des Systems waren vertreten, die meisten Phagen schienen aber Cas-Proteine des Wirts zu benutzen, um sich selbst zu schützen. Darüber hinaus schienen die Phagen das CRISPR-Immunsystem der Wirte darin zu unterstützen, konkurrierende Phagen abzuwehren. Manche Pseudomonas-infizierende Phagen kodieren auch für Anti-CRISPRs (Acrs) und Proteine, die ein Zellkern-ähnliches Kompartiment bilden, in dem der Phage sein Genom unabhängiger vom Wirt replizieren kann (siehe Viroplasma). Die Autoren sehen ihre Arbeit als einen weiteren Beleg für die weltweite Verbreitung der Riesenphagen. Sie fanden Belege, dass die Phagen zwischen verschiedenen Wirten und Ökosystemen wanderten, was eine Bedeutung für die Verbreitung von Toxin- und Antibiotikaresistenz-Genen hat. Ihre CRISPR-Werkzeuge könnten sich in Zukunft nutzen lassen, um die „Genschere“ CRISPR/Cas zu verbessern und ihre Funktionalität zu erweitern.[20][23][24][25][26][27][28]
Ein weiterer Riesenphage ist der Megasphaera-Phage A9 (alias Huge Phage A9),[29] nicht zu verwechseln mit dem Brochothrix-Phagen A9 (Spezies Brochothrix-Virus A9, Herelleviridae).
Schwanzlose Phagen
[Bearbeiten | Quelltext bearbeiten]Lange Zeit hat die Forschung nur Mitglieder der Ordnung Caudovirales betrachtet, deren Vertreter Phagen (Bakterien- und Archaeenviren) mit Kopf-Schwanz-Struktur sind. Erst in letzter Zeit sind „schwanzlose“ Phagen Gegenstand von Forschungsarbeiten geworden. Einige Vertreter sind:
- Ordnung Tubulavirales (filamentöse Bakteriophagen) u. a. mit Familie Inoviridae
- Familie Finnlakeviridae (ssDNA, mit Spezies Flavobacterium virus FLiP alias Phage FLiP[30])
- Familie Autolykiviridae (dsDNA)[31][32][33][34][35][36]
- Spezies Planktothrix-Phage PaV-LD (Cyanophage PaV-LD)[37][38][39]
crAssphagen und Gubaphagen
[Bearbeiten | Quelltext bearbeiten]Camarillo-Guerrero, Almeida t al. beschrieben 2019/2020 die Ergebnisse ihrer Metagenomanalysen der menschlichen Darmflora hinsichtlich Bakteriophagen. Sie machen dabei eine neue Klade aus, genannt „Gubaphagen“ (englisch Gut Bacteroidales phage, Gubaphage clade) (mit zwei Gattungen: G1 – infiziert Bacteroides, und G2 – infiziert Parabacteroides), die nach den crAssphagen (offiziell Ordnung Crassvirales[40]) mit ca. zehn Gattungen[41] (en. crAsslike phages, aufgrund ihres Podoviren-Morphotyps ursprünglich vorgeschlagene Mitglieder der früheren Familie Podoviridae (ehemalige Ordnung Caudovirales), mit ca. zehn Gattungen)[42][43][44] die zweithäufigsten Viren (d. h. Bakteriophagen) in dieser Umgebung darstellen. Die Merkmale der Gubaphagen erinnern dabei an die von „p-crAssphage“.[45][46] Die Gubaphagen sind wegen ihrer Ähnlichkeit mit den crAssphagen wahrscheinlich ebenfalls Mitglieder der Crassvirales (oder jedenfalls der Caudoviricetes).
Anwendungsgebiete
[Bearbeiten | Quelltext bearbeiten]Phagen haben in Medizin, Biologie, Agrarwissenschaften, vor allem im Bereich der Gentechnik, ein breites Anwendungsspektrum gefunden. So verwendet man Phagen in der Medizin aufgrund ihrer Wirtsspezifität zur Bestimmung von bakteriellen Erregern. Dieses Verfahren nennt man Lysotypie. Aufgrund der immer häufiger auftretenden multiplen Antibiotikaresistenzen wird zurzeit intensiv an der Anwendung von Bakteriophagen als Antibiotika-Ersatz in der Humanmedizin (siehe: Phagentherapie) geforscht. Probleme ergeben sich hierbei durch die geringe Stabilität von Phagen im Körper, da sie in recht kurzer Zeit durch Fresszellen als Fremdkörper beseitigt werden. Diese Anwendung von Phagen zur Therapie bakterieller Infektionen entdeckte Felix d’Hérelle (s. o.) lange vor Entdeckung des Penicillins und der Antibiotika. Später wurde die Phagentherapie jedoch mit der Einführung der Chemotherapie per Antibiotika als unpraktisch erachtet und geriet in Vergessenheit. D’Hérelle gründete 1934 zusammen mit dem georgischen Mikrobiologen Georgi Eliava in der Georgischen Sozialistischen Sowjetrepublik das Eliava-Institut für Phagenforschung, welches heute noch besteht.[47] Heute wird dort sowie am Ludwik-Hirszfeld-Institut für Immunologie und Experimentelle Therapie in Breslau (Teil der Polnischen Akademie der Wissenschaften) die Phagentherapie bei ansonsten therapieresistenten bakteriellen Infektionen durchgeführt.[2] In Deutschland ist die Anwendung zu therapeutischen Zwecken bisher nicht zulässig.
Die Anwendungen in der Lebensmittelproduktion sind vielfältig; so kommt beispielsweise ein Sprühnebel aus Phagen beim Verpacken von Würstchen oder dem Aufschneiden von Käseaufschnitt zum Einsatz.[48]
In der Gentechnik werden temperente Phagen als Vektoren (z. B. der Phage λ) benutzt. Hierzu werden Phagen so präpariert, dass ihrem Genom die Gene, welche die Virulenz hervorrufen, entnommen und durch Gene ersetzt werden, die für gentechnische Belange interessant sind, so beispielsweise Gene, die zur Insulinproduktion benötigt werden. Diese veränderten Phagen werden nun mit geeigneten Bakterien, zum Beispiel E. coli, in Kontakt gebracht. Nach einer Überprüfung, ob das gewünschte Gen in die Erbsubstanz des Bakteriengenoms integriert wurde (man bedient sich hierzu genexprimierter Antibiotikaresistenzen, die an die zu klonierenden Wunschgene angeschlossen werden), können die modifizierten Bakterienzellen weiterkultiviert werden und das in diesem Falle produzierte Insulin isoliert werden. Ähnlich werden Phagen in der Agrartechnik zur Transduktion bestimmter Gene in Nutzpflanzen eingesetzt. Eine wichtige Anwendung in der Biochemie ist das Phagen-Display zur Identifikation von Bindungspartnern, z. B. bei der Isolierung neuer Wirkstoffe.
Einfacher als die Nutzung von Phagen ist jedoch die Transformation freier DNA, die heutzutage überwiegend zum Transfer in die Bakterienzellen verwendet wird.
Phagen und deren Bestandteile werden für die Entfernung von mikrobiellen Verunreinigungen in Lebensmitteln (z. B. per affinitätsmagnetische Separation) sowie mit Endotoxinen kontaminierten Laborproben verwendet.[49][50] Des Weiteren ergeben sich humandiagnostische Anwendungen, vor allem im klinischen Bereich zur Dekolonisierung von pathogenen Krankenhauskeimen wie MRSA.[51][52] Durch Proteindesign lassen sich die Phagenproteine zum jeweiligen Anwendungszweck optimieren.
Möglicher wirtschaftlicher Schaden
[Bearbeiten | Quelltext bearbeiten]Bakteriophagen können überall dort Schaden anrichten, wo bakterielle Prozesse dem Menschen dienen und erwünscht sind. Infektion von Milchsäurebakterien (LAB) durch Phagen aus Rohmilch ist die häufigste Ursache für verringerte oder fehlende Enzymaktivität in Starterkulturen für die Käse- oder Dickmilchproduktion.[53]
Klassifikation
[Bearbeiten | Quelltext bearbeiten]Prokaryoten infizierende Viren (Bakterien- und Archaeenviren; der Begriff „Bakteriophagen“ umfasst herkömmlich auch Viren der Archaeen, da diese ursprünglich nicht von den Bakterien unterschieden wurden) bilden keine geschlossene Verwandtschaftsgruppe (Taxon). Für viele Gruppen dieser Viren finden sich noch informelle Bezeichnungen nach ihren Wirten (s. o.), z. B. Cyanophagen (Cyanobakterien), Coliphagen (Colibakterium E. coli) und stellen meist ebenfalls keine Verwandtschaftsgruppen dar. Eine weitere Besonderheit sind Satellitenviren, deren Helferviren Bakteriophagen sind; diese werden gelegentlich Satellitenphagen genannt. Ein Beispiel ist „Escherichia-Phage P4“ (Caudoviricetes), der den Phagen der Gattung Peduovirus (früher P2likevirus, Familie Peduoviridae) als Helfervirus benötigt.[54][55]
Klassifikation nach Baltimore
[Bearbeiten | Quelltext bearbeiten]Die Phagen-Familien (Bakterien- und Archaeenviren, ohne Virophagen) werden nach der Baltimore-Klassifikation anhand des Aufbaus ihres Genoms wie folgt gruppiert:
- dsDNA-Phagen:
- Ackermannviridae
- Aggregaviridae
- Ahmunviridae *
- Ahpuchviridae *
- Ampullaviridae *
- Anaerodiviridae *
- Aliceevansviridae
- Aoguangviridae *
- Arenbergviridae
- Assiduviridae
- Autographiviridae
- Autolykiviridae
- Bicaudaviridae *
- Casjensviridae
- Chaacviridae *
- Chaseviridae
- Clavaviridae *
- Corticoviridae
- Crevaviridae
- Demerecviridae
- Drexlerviridae
- Druskaviridae *
- Duneviridae
- Ekchuahviridae *
- Fervensviridae *
- Forsetiviridae
- Fuselloviridae *
- Globuloviridae *
- Guelinviridae
- Graaviviridae *
- Guttaviridae *
- Hafunaviridae *
- Haloferuviridae *
- Halomagnusviridae *
- Halspiviridae *
- Helgolandviridae
- Herelleviridae
- Guttaviridae
- Intestiviridae
- Itzamnaviridae *
- Kyanoviridae
- Leisingerviridae *
- Lipothrixviridae
- Madisaviridae *
- Mesyanzhinovviridae
- Molycolviridae
- Naomviridae
- Ovaliviridae *
- Orlajensenviridae
- Pachyviridae
- Peduoviridae
- Pervagoviridae
- Plasmaviridae
- Portogloboviridae *
- Pungoviridae *
- Pyrstoviridae *
- Rountreeviridae
- Rudiviridae
- Salasmaviridae
- Saparoviridae *
- Schitoviridae
- Shortaselviridae *
- Simuloviridae *
- Skuldviridae *
- Soleiviridae *
- Sphaerolipoviridae *
- Speroviridae *
- Stanwilliamsviridae
- Straboviridae
- Suoliviridae *
- Steigviridae
- Straboviridae
- Tectiviridae
- Thaspiviridae *
- Tristromaviridae *
- Turriviridae *
- Verdandiviridae *
- Vertoviridae *
- Ungulaviridae *
- Vilmaviridae
- Winoviridae
- Yangangviridae *
- Yanlukaviridae *
- Zierdtviridae
- Zobellviridae
- ssDNA-Phagen:
- Sonderfall
- Pleolipoviridae (mit Gattung Gammapleolipovirus und Spezies Gammapleolipovirus His2 alias His 2 virus, Haloarcula virus His2[56])
- dsRNA-Phagen:
- ssRNA-Phagen:
- Fiersviridae (alias Leviviridae)
* kennzeichnet Archaeenviren
Taxonomische Klassifizierung nach ICTV
[Bearbeiten | Quelltext bearbeiten]In der Systematik der Virus-Taxonomie nach dem International Committee on Taxonomy of Viruses (ICTV) finden sich Phagen in folgenden taxonomischen Gruppen:
| Realm | Klasse/Ordnung | Familie/Unterfamilie | Morphologie | Genom | Beispiele |
|---|---|---|---|---|---|
| Riboviria | Levivirales | Leviviridae | unbehüllt, isometrisch[58] | ssRNA, linear | MS2, Qβ |
| Mindivirales | Cystoviridae | behüllt, sphärisch | dsRNA, segmentiert | Phi6 | |
| Varidnaviria | Belfryvirales | Turriviridae | behüllt, isometrisch | dsDNA, linear | STIV1 |
| Halopanivirales | Sphaerolipoviridae | behüllt, isometrisch | dsDNA, linear | Phage SH1 | |
| Kalamavirales | Tectiviridae | unbehüllt, isometrisch | dsDNA, linear | PRD1 | |
| Vinavirales | Corticoviridae | unbehüllt, isometrisch | dsDNA, zirkulär | PM2 | |
| Duplodnaviria | Caudoviricetes | Ackermannviridae | unbehüllt, kontraktiler Schwanz (Myoviren) | dsDNA, linear | ϕMAM1 |
| Autographiviridae | unbehüllt, kontraktiler Schwanz (Podoviren) | dsDNA, linear | Acinetobacter-Phage P2 | ||
| Straboviridae | unbehüllt, kontraktiler Schwanz (Myoviren) | dsDNA, linear | T4 | ||
| Peduoviridae | unbehüllt, kontraktiler Schwanz (Myoviren) | dsDNA, linear | Coliphage P2 | ||
| nicht zugeordnet | unbehüllt, kontraktiler Schwanz (Myoviren) | dsDNA, linear | Mu, P1 | ||
| Drexlerviridae | unbehüllt, nichtkontraktiler Schwanz (lang) (Siphoviren) | dsDNA, linear | λ | ||
| Hendrixvirinae | unbehüllt, nichtkontraktiler Schwanz (lang) (Siphoviren) | dsDNA, linear | HK97 | ||
| Demerecviridae | unbehüllt, nichtkontraktiler Schwanz (lang) (Siphoviren) | dsDNA, linear | T5 | ||
| nicht zugeordnet | unbehüllt, nichtkontraktiler Schwanz (lang) (Siphoviren) | dsDNA, linear | N15 | ||
| Autographiviridae | unbehüllt, nichtkontraktiler Schwanz (kurz) (Podoviren) | dsDNA, linear | T7, T3 | ||
| Salasmaviridae | unbehüllt, nichtkontraktiler Schwanz (kurz) (Podoviren) | dsDNA, linear | Φ29 | ||
| nicht zugeordnet | unbehüllt, nichtkontraktiler Schwanz (lang) (Podoviren) | dsDNA, linear | P22 | ||
| Monodnaviria | Haloruvirales | Pleolipoviridae | behüllt, pleomorph | ssDNA, zirkulär / dsDNA, zirkulär / dsDNA linear | HHPV1, HRPV1 |
| Petitvirales | Microviridae | unbehüllt, isometrisch | ssDNA, zirkulär | ΦX174 | |
| Tubulavirales | Inoviridae | unbehüllt, filamentös | ssDNA, (meist) zirkulär | M13, CTXφ | |
| Adnaviria | Ligamenvirales | Lipothrixviridae | behüllt, stabförmig | dsDNA, linear | AFV1 |
| Rudiviridae | unbehüllt, stabförmig | dsDNA, linear | SIRV1 | ||
| Primavirales | Tristromaviridae | behüllt, stabförmig | dsDNA, linear | TTSV1 | |
| nicht zugeordnet | nicht zugeordnet | Ampullaviridae[59] | behüllt, flaschenförmig | dsDNA, linear | ABV |
| Bicaudaviridae[60] | unbehüllt, zitronenförmig | dsDNA, zirkulär | ATV | ||
| Clavaviridae | unbehüllt, stabförmig | dsDNA, zirkulär | APBV1 | ||
| Finnlakeviridae | dsDNA | FLiP[61] | |||
| Fuselloviridae[62] | unbehüllt, zitronenförmig | dsDNA, zirkulär | SSV1 | ||
| Globuloviridae[63] | behüllt, isometrisch | dsDNA, linear | PSV | ||
| Guttaviridae | unbehüllt, ovoid | dsDNA, zirkulär | SNDV, APOV1 | ||
| Plasmaviridae | behüllt, pleomorph | dsDNA, zirkulär | L2-Phage | ||
| Portogloboviridae | behüllt, isometrisch | dsDNA, zirkulär | SPV1 | ||
| Spiraviridae | unbehüllt, stabförmig | ssDNA, zirkulãr | ACV |
Die Mitglieder der Familie Picobirnaviridae (Ordnung Durnavirales) scheinen ebenfalls Bakterien zu infizieren, keine Säugetiere.[64]
Eine weitere vorgeschlagene Phagenfamilie sind die „Autolykiviridae“ (dsDNA).[31]
Literatur
[Bearbeiten | Quelltext bearbeiten]- Nicholas H. Mann: The third age of phage. In: PLOS Biology. Band 3, Nr. 5, 17. Mai 2005, Artikel e182, doi:10.1371/journal.pbio.0030182, (Volltext online).
- Nancy Trun, Janine Trempy: Bacteriophage. In: Nancy Jo Trun, J. E. Trempy, Janine Trempy: Fundamental Bacterial Genetics. Blackwell, Oxford 2003, ISBN 0-632-04448-9; blackwellpublishing.com (PDF; 263 kB).
- A Górski, B Weber-Dabrowska: The potential role of endogenous bacteriophages in controlling invading pathogens. In: Cellular and Molecular Life Sciences. Band 62, Nr. 5, März 2005, S. 511–519, doi:10.1007/s00018-004-4403-6, PMID 15747058 (englisch).
- Forest Rohwer, Merry Youle, Heather Maughan, Nao Hisakawa, Leah L Pantéa: Life in our phage world: a centennial field guide to the Earth’s most diverse inhabitants. Wholon, San Diego CA 2014, ISBN 978-0-9904943-0-0.
- Hans Günther Schlegel, Georg Fuchs (Hrsg.): Allgemeine Mikrobiologie. 8. Auflage. Thieme, Stuttgart 2006, ISBN 3-13-444608-1.
- Jong-Geol Kim, So-Jeong Kim, Virginija Cvirkaite-Krupovic, Mart Krupovic et al.: Spindle-shaped viruses infect marine ammoniaoxidizing thaumarchaea. In: Proceedings of the National Academy of Sciences (PNAS), Juli 2019, Band 116, Nr. 31, Artikel 201905682, doi:10.1073/pnas.1905682116; Volltext (PDF) researchgate.net
Weblinks
[Bearbeiten | Quelltext bearbeiten]- Aufbau und Vermehrung mit Animation
- Bakteriophagen und Phagentherapie: Fragen und Antworten im Überblick, Informationen des Leibniz-Instituts DSMZ (Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH) in Braunschweig
- Bundesinstitut für Risikobewertung (BfR): Fragen und Antworten zu Bakteriophagen.
- Website des Eliava-Instituts
- Phagentherapie am Ludwik-Hirszfeld-Institut für Immunologie und Experimentelle Therapie, Breslau
- Phagentherapie gegen Lungenentzündung. Forschungsprojekt in Paris; idw-online.de
- Phagoburn. – EU-Forschungsprojekt zur Phagentherapie bei Brandverletzten; phagoburn.eu
- „Bakterienfresser“ statt Antibiotika. In: VDI-Nachrichten, 25. September 2015
- Heilende Viren – Infektionen bekämpfen mit Bakteriophagen. Radiosendung, Bayern 2, 29. September 2012, abgerufen am 25. September 2015.
- Bacterial Predator Could Help Reduce COVID-19 Deaths – “Potential Game Changer”. scitechdaily.com, 26. Juni 2020, Quelle: University of Birmingham.
- Jean Crépu: Mit Viren aus der Antibiotika-Krise. Doku auf ARTE (F 2019, 54 Min).
- Robert C. Edgar et al.: Petabase-scale sequence alignment catalyses viral discovery. In: Nature, 26. Januar 2022, Band 602, S. 142–147; doi:10.1038/s41586-021-04332-2 – Über die Identifizierung zahlreicher neuer Verwandter der Coronaviren, Hepatitis-Deltavirus und Riesenphagen (en. huge phages) aus Datensätzen öffentlich zugänglicher Gendatenbanken.
- Thomas G. Laughlin, Amar Deep, Amy M. Prichard, Christian Seitz, Yajie Gu, Eray Enustun, Sergey Suslov, Kanika Khanna, Erica A. Birkholz, Emily Armbruster, J. Andrew McCammon, Rommie E. Amaro, Joe Pogliano, Kevin D. Corbett, Elizabeth Villa: Architecture and self-assembly of the jumbo bacteriophage nuclear shell. In: Nature, 3. August 2022; doi:10.1038/s41586-022-05013-4. Dazu:
- Tessa Koumoundouros: Giant Viruses Called 'Jumbo Phages' Could Help Us Fight Antibiotic Resistance. sciencealert, 5. August 2022.
- Bakteriophagen. Universität Hohenheim (Memento im Webarchiv vom 5. Juni 2019). Suche Uni-Hohenheim-Artikeln zu „Bakteriophagen“
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ Bacillus phage Gamma (species). NCBI.
- ↑ a b Daniel Bojar: Nützliche Bakterienkiller. spektrum.de – Spektrum der Wissenschaft, Juni 2020, S. 40–45.
- ↑ SIB: Viruses infecting bacteria. ViralZone.
- ↑ Mart Krupovic, Anja Spang, Simonetta Gribaldo, Patrick Forterre, Christa Schleper: A thaumarchaeal provirus testifies for an ancient association of tailed viruses with archaea. In: Biochemical Society Transactions. Januar 2011, Band 39, Nr. 1, S. 82-88, doi:10.1042/BST0390082, PMID 21265751.
- ↑ Karin Mölling: Supermacht des Lebens. Reisen in die erstaunliche Welt der Viren. 1. Auflage. Beck, München 2015, ISBN 978-3-406-66969-9.
- ↑ F. d’Hérelle: Sur un microbe invisible antagoniste des bacilles dysentériques. In: l’Académie des Sciences. Nr. 165, Gauthier-Villars, Paris 1917, S. 373–375.
- ↑ Richard Lucius, Brigitte Loos-Frank, Richard P. Lane: Biologie von Parasiten. 3., aktualisierte und überarbeitete Auflage. Springer-Verlag, Berlin 2018, ISBN 978-3-662-54862-2, S. 4 (google.de [abgerufen am 17. März 2019]).
- ↑ T-Phages. NCBI.
- ↑ Rolf Sauermost, Doris Freudig et al.: T-Phagen. In: Lexikon der Biologie, Spektrum.de; abgerufen am 31. Januar 2021. Die Familienzuordnungen entsprechen nicht mehr den aktuellen Stand nach ICTV.
- ↑ L. McKane, J. J. Ferretti: Phage-host interactions and the production of type A streptococcal exotoxin in group A streptococci. In: Infection and Immunity. Band 34. Jahrgang, Nr. 3, Dezember 1981, S. 915–919, PMID 7037644, PMC 350956 (freier Volltext) – (englisch).
- ↑ Victor Krylov, Maria Bourkaltseva, Elena Pleteneva, Olga Shaburova, Sergey Krylov, Alexander Karaulov, Sergey Zhavoronok, Oxana Svitich, Vitaly Zverev; Julie Thomas, Lindsay Black (Hrsg.): Phage phiKZ—The First of Giants. In: Viruses. Band 13, Nr. 2, 20. Januar 2021, Sonderausgabe: Giant or Jumbo Phages. S. 149, doi:10.3390/v13020149.
- ↑ a b Audra E. Devoto, Joanne M. Santini et al.: Megaphages infect Prevotella and variants are widespread in gut microbiomes. In: Nature Microbiology, 28. Januar 2019, Band 4, S. 693–700, doi:10.1038/s41564-018-0338-9, insbes. Tbl. 1 und Supplementary Fig. 11 (PDF; 9,2 MB).
- ↑ Lakshminarayan M. Iyer, Vivek Anantharaman, Arunkumar Krishnan, A. Maxwell Burroughs, L. Aravind: Jumbo Phages: A Comparative Genomic Overview of Core Functions and Adaptions for Biological Conflicts. In: MDPI Viruses. Band 13, Nr. 1: Special Issue Giant or Jumbo Phages. 5. Januar 2021, 63; doi:10.3390/v13010063, insbesondere Supplement (Zip mit PDF und xlsx).
- ↑ NCBI: Lak megaphage sp. (species).
- ↑ NCBI: Sphingomonas phage PAU (species)
- ↑ Richard Allen White III, Curtis A. Suttle: The Draft Genome Sequence of Sphingomonas paucimobilis Strain HER1398 (Proteobacteria), Host to the Giant PAU Phage, Indicates That It Is a Member of the Genus Sphingobacterium (Bacteroidetes). In: Genome Announcements. Band 1, Nr. 4, Juli-August 2013, Artikel e00598-13, doi:10.1128/genomeA.00598-13, PMID 23929486, PMC 3738902 (freier Volltext).
- ↑ University of California – Berkeley, ScienceDaily: Whopping big viruses prey on human gut bacteria. Largest phages ever found in humans target bacteria associated with hunter-gatherer diets. sciencedaily.com, 28. Januar 2019.
- ↑ UCL: New, giant bacterial virus found in human gut. University College London, 29. Januar 2019.
- ↑ Colm Gorey: Gargantuan viruses discovered in humans raise questions about life itself. siliconrepublic.com, 29. Januar 2019.
- ↑ a b Basem Al-Shayeb, Rohan Sachdeva, L. Chen, Jillian F. Banfield et al.: Clades of huge phages from across Earth’s ecosystems. In: Nature. vom 12. Februar 2020, doi:10.1038/s41586-020-2007-4, bioRxiv: 10.1101/572362v1 (Preprint-Volltext).
- ↑ NCBI: Biggie virus. (species)
- ↑ Ed Yong: A Huge Discovery in the World of Viruse. In: The Atlantic, 20. Februar 2020.
- ↑ Michael Le Page: Giant viruses have weaponised CRISPR against their bacterial hosts. In: NewScientist, 30. März 2019.
- ↑ Giant Bacteriophages Bridge Gap between Living Microbes and Viral Machines. SCI-NEWS, 13. Februar 2020.
- ↑ Tessa Koumoundouros: Scientists Discover Giant Viruses With Features Only Seen Before in Living Cells. ScienceAlert, 14. Februar 2020.
- ↑ Daniela Albat: Phage mit rekordgroßem Genom entdeckt. scinexx.de, 18. Februar 2020.
- ↑ Jan Osterkamp: Anti-CRISPR soll CRISPR besser machen. Spektrum.de, 16. Januar 2020.
- ↑ Annika Röcker: Gegen manche Viren ist die Genschere machtlos. Spektrum.de, 10. Dezember 2019.
- ↑ Basem Al-Shayeb, Rohan Sachdeva, Lin-Xing Chen, Cindy J. Castelle, Alexander L. Jaffe, Jennifer A. Doudna, Jillian F. Banfield et al.: Clades of huge phage from across Earth’s ecosystems. In: Nature. Nr. 578, 12. Februar 2020, S. 425–431, doi:10.1038/s41586-020-2007-4. Dazu:
- Volltext. (PDF) PrePrint, 11. März 2019; doi:10.1101/572362
- Huge bacteria-eating viruses close gap between life and non-life – Large bacteriophages carry bacterial genes, including CRISPR and ribosomal proteins. eurekalert.org, 12. Februar 2020.
- ↑ Elina Laanto, Sari Mäntynen, Luigi De Colibus, Jenni Marjakangas et al.: Virus found in a boreal lake links ssDNA and dsDNA viruses. In: Proceedings of the National Academy of Sciences. Band 114, Nr. 31, Juli 2017, doi:10.1073/pnas.1703834114.
- ↑ a b Kathryn M. Kauffman, Fatima A. Hussain, Joy Yang et al.: A major lineage of non-tailed dsDNA viruses as unrecognized killers of marine bacteria. In: Nature. Band 554, S. 118–122, 24. Januar 2018, doi:10.1038/nature25474.
- ↑ Scientists Find New Type of Virus in World’s Oceans: Autolykiviridae. sci-news, 25. Januar 2018.
- ↑ David L. Chandler: Researchers Discover a Missing Link in Virus Evolution. SciTechDaily, 25. Januar 2018.
- ↑ Forscher entdecken ein mysteriöses Virus, das die Ozeane dominiert. business insider, 29. Januar 2018.
- ↑ Never-Before-Seen Viruses With Weird DNA Were Just Discovered in The Ocean. sciencealert.com, 25. Januar 2018.
- ↑ Autolykiviridae. (family) – unclassified dsDNA viruses. NCBI.
- ↑ E-Bin Gao, Xiu-Ping Yuan, Ren-hui Li, Qi-Ya Zhang: Isolation of a novel cyanophage infectious to the filamentous cyanobacterium “Planktothrix agardhii” (Cyanophyceae) from Lake Donghu, China. In: Aquatic microbial ecology. (AME), Band 54, Nr. 1, Februar 2009, S. 163–170, doi:10.3354/ame01266; Volltext (PDF; 650 kB).
- ↑ Mélanie Gerphagnon, Deborah J. Macarthur, Delphine Latour, Claire M. M. Gachon, Floris Van Ogtrop, Frank H. Gleason, Télesphore Sime-Ngando: Microbial players involved in the decline of filamentous and colonial cyanobacterial blooms with a focus on fungal parasitism. ( des vom 12. Mai 2021 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis. In: sfam Environmental Microbiology. Band 17, Nr. 8, S. 2573–2587, doi:10.1111/1462-2920.12860; Volltext als PDF ( des vom 12. Mai 2021 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis. – insbesondere Supplement; Volltext als PDF ( des vom 12. Mai 2021 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis. Table 2.
- ↑ Planktothrix phage PaV-LD. (species). NCBI.
- ↑ Dann Turner, Andrew M. Kropinski, Evelien M. Adriaenssens: A Roadmap for Genome-Based Phage Taxonomy. In: MDPI Viruses. Band 13, Nr. 3, Section Bacterial Viruses, 18. März 2021, S. 506, doi:10.3390/v13030506.
- ↑ CrAssphage: Previously Unknown Ancient Gut Virus Lives in Half World’s Population. sci-news, 11. August 2014 (englisch).
- ↑ Natalya Yutin, Kira S. Makarova, Ayal B. Gussow, Mart Krupovic, Anca Segall, Robert A. Edwards, Eugene V. Koonin: Discovery of an expansive bacteriophage family that includes the most abundant viruses from the human gut. In: Nature Microbiology. Band 3. Jahrgang, Nr. 1, 2017, S. 38–46, doi:10.1038/s41564-017-0053-y, PMID 29133882, PMC 5736458 (freier Volltext) – (englisch).
- ↑ Eugene V. Koonin: Behind the paper: The most abundant human-associated virus no longer an orphan ( des vom 30. Juni 2022 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis., November 13th, 2017
- ↑ SIB: crAsslike phages. viralzone.expasy.org – Order: Caudovirales, Estimated about 10 genera.
- ↑ Luis Fernando Camarillo-Guerrero, Alexandre Almeida, Guillermo Rangel-Pineros, Robert D. Finn, Trevor D. Lawley: Massive expansion of human gut bacteriophage diversity. In: Cell Resource, 18. Februar 2021, Band 184, Nr. 4, S. 1098–1109.e9, doi:10.1016/j.cell.2021.01.029. Preprint vom 3. September 2020: bioRxiv, europepmc.org – doi:10.1101/2020.09.03.280214. Dazu:
- Martin Vieweg: Tausende Virenarten der Darmflora entdeckt. wissenschaft.de, 18. Februar 2021 (deutsch)
- Daniel Lingenhöhl: Virobiom: Darm beherbergt zehntausende unbekannte Virenarten. spektrum.de, 19. Februar 2021 (deutsch)
- Biologists Find Almost 143,000 Bacteriophage Species in Human Gut. sci-news, 19. Februar 2021 (englisch)
- Peter Dockrill: Scientists Find 140,000 Virus Species in The Human Gut, And Most Are Unknown. sciencealert.com, 28. Februar 2021 (englisch)
- Scientists identify more than 140,000 virus species in the human gut. sciencedaily.com, 18. Februar 2021 (englisch)
- ↑ Luis Fernando Camarillo Guerrero: Integrative Analysis of the Human Gut Phageome Using a Metagenomics Approach. Doktorarbeit, Gonville & Caius College, University of Cambridge, August 2020; doi:10.17863/CAM.63973.
- ↑ Daria Vaisman: Eat Me. slate.com, Mai 2006.
- ↑ Bettina Hofer: Konservieren mit Viren. Heise Technology Review, 28. Februar 2013, abgerufen am 7. August 2014.
- ↑ J. W. Kretzer, R. Lehmann et al.: Use of high affinity cell wall-binding domains of bacteriophage endolysins for immobilization and separation of bacterial cells. In: Applied and Environmental Microbiology. Band 73, 2007, S. 1992–2000.
- ↑ C. Rozand, P. C. H. Feng: Specificity analysis of a novel phage-derived ligand in an Enzyme-linked fluorescent assay for detection of Escherichia coli O157:H7. J. In: food protection. Band 72, 2009, S. 1078–1081.
- ↑ Bacteriophages – New Applications in Food Microbiology ( vom 2. März 2013 im Internet Archive) bioFood n°3 Dezember 2006, S. 2.
- ↑ Anwendungen der Phageligand-Technologie (Endotoxinentfernung, Endotoxinnachweis, Lebensmittelqualitätstestung). hyglos.de
- ↑ D. M. Guglielmotti, D. J. Mercanti, J. A. Reinheimer, A. D. L. Quiberoni: Efficiency of physical and chemical treatments on the inactivation of dairy bacteriophages. In: Frontiers in Microbiology. Band 2, 2012, doi:10.3389/fmicb.2011.00282.
- ↑ Renata Filipa Cruz de Matos: „Enterococcus faecalis“ V583 prophages: Dynamic interactions and contribution to bacterial pathogenic traits. Dissertation, Universidade Nova de Lisboa (UNL), Juli 2013 (Volltext als PDF).
- ↑ NCBI: Phage P4 satellite (no rank)
- ↑ Gammapleolipovirus His2. ICTV Taxonomy history, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- ↑ S. McGrath: Bacteriophage: Genetics and Molecular Biology. Hrsg.: D. van Sinderen. 1st Auflage. Caister Academic Press, 2007, ISBN 978-1-904455-14-1 (Online).
- ↑ isometrisch bei Virusteilchen: in jeder Richtung etwa gleiche Raumausdehnung, also beispielsweise kugelförmig oder ikosaedrisch.
- ↑ SIB: Ampullaviridae. viralzone.expasy.org
- ↑ SIB: Bicaudaviridae. viralzone.expasy.org
- ↑ Elina Laanto, Sari Mäntynen, Luigi De Colibus, Jenni Marjakangas, Ashley Gillum, David I. Stuart, Janne J. Ravantti, Juha Huiskonen, Lotta-Riina Sundberg: Virus found in a boreal lake links ssDNA and dsDNA viruses. In: Proceedings of the National Academy of Sciences. Band 114, Nr. 31, July.2017, doi:10.1073/pnas.1703834114.
- ↑ SIB: Fuselloviridae. viralzone.expasy.org
- ↑ SIB: Globuloviridae. viralzone.expasy.org
- ↑ S. R. Krishnamurthy, D. Wang: Extensive conservation of prokaryotic ribosomal binding sites in known and novel picobirnaviruses. In: Virology. Band 516. Jahrgang, 2018, S. 108–114, doi:10.1016/j.virol.2018.01.006, PMID 29346073 (englisch).