Vorlage Diskussion:Infobox Virus
neu
[Quelltext bearbeiten]Hallo zusammen, ich habe die Vorlage fuer Viren gebaut und wuerde mich gerne ueber Rueckmeldungen freuen.
Die Vorlagen mit dem direkten Klick auf NCBI, Ref-Sequenz und ICTV sind natürlich klasse. Vielleicht sollte man statt Links das untere Kästchen anders nennen, mir fällt aber gerade nix ein (mein Hirn ist noch leer). Und bei ICTV vielleicht "ICTV Nummer" oder so; dort heißt es "decimal code" [1]. Um das ganze nicht zu lang auszudehnen kann man meiner Meinung nach auch die Baltimore-Klassifikation rausschmeißen, hatte ich ja schon erzählt, ist ohne Bedeutung. Aber das ist ja jetzt oberschick, einfach die Ref.-Nr eingeben, und schon hat man die Sequenz; einfach den Taxoncode rein, und schon ist der aktuelle Link zur Datenbank da. Ach ja, das fiel mir auch schon bei meinen Vorlagen auf, den Link auf Systematik (Biologie) kann man am besten gegen Virusklassifikation austauschen. Jetzt müssen wir das noch mit der renovierten Taxobox der iologen abgleichen, da meine jetzige Version auf deren Vorläufer beruht. Aber Viren sind ja fast Chemie ..... Märci vielmals! --Gleiberg 20:34, 5. Mai 2009 (CEST)
- Ja mir fiel auch nichts besseres ein für das untere Kästchen, vielleicht kommt da noch ein guter Name. Die ICTV habe ich mal auf "ICTV Nummer" geändert, ich habe auch "ICTVdB Virus Code" als offizielle Bezeichung gefunden, ev. wäre auch "ICTV Code" ein Vorschlag? Wegen der Baltimore Klassifikation, das können wir jetzt schon machen aber es ist auch denkbar, das später rauszunehmen. Der Vorteil ist, dass ich zentral an dieser Vorlage etwas ändern kann und die Baltimore-Klassifikation mit einem Klick in allen Artikeln mit dieser Vorlage zum Verschwinden bringen kann - aber wenn sie total obsolet ist, kann sie auch gleich raus. Anstatt Virusklassifikation wäre da nicht sogar Virus-Taxonomie besser? Gruss -- hroest Disk 21:04, 5. Mai 2009 (CEST)
- Das mit der zentralen Änderung an der Vorlage ist natürlich ein gutes Argument, stimmt. ICTVdB Virus Code ist auch besser, und mit Virus-Taxonomie hast du auch recht, das hatte ich gemeint, kenne meine eigenen Artikel nicht mehr (*Auf-Kur-nach-Bad-Alzheim-geh*) --Gleiberg 21:35, 5. Mai 2009 (CEST)
- ok, ich habe das mal soweit umgesetzt, der Link geht jetzt nach Virus-Taxonomie und der Link von ICTV heisst jetzt ICTVdB Virus Code; siehe http://de.wikipedia.org/wiki/Benutzer:Hannes_R%C3%B6st/Werkstatt/Slot5 hier]. Dann wollen wir Baltimore vorläufig drin lassen? Weitere Fragen:
- Das mit der zentralen Änderung an der Vorlage ist natürlich ein gutes Argument, stimmt. ICTVdB Virus Code ist auch besser, und mit Virus-Taxonomie hast du auch recht, das hatte ich gemeint, kenne meine eigenen Artikel nicht mehr (*Auf-Kur-nach-Bad-Alzheim-geh*) --Gleiberg 21:35, 5. Mai 2009 (CEST)
- Momentan sind alle Felder optional, sollten einige obligatorisch sein? Wenn ja welche? Ich dachte an Name und Genom, alles andere kann ja stark variieren (zB keine Ordnung für gewisse Viren, da kann man das nicht obligatorisch machen - ausser man zwingt sie "nicht klassifiziert" reinzuschreiben in dem Falle, wie wäre das?).
- Spezifisch noch, wie stark wollen wir den Nutzer bei dem Punkt "Genom" einschränken? Darf man da angeben was man will oder soll das von der Vorlage kontrolliert werden? Wir könnten einzelne Felder haben wie Polarität, ss/ds, RNA/DNA, linear JA/NEIN etc - aber das wäre etwas einschränkend für den Benutzer oder?
- sollten wir Kategorien haben für Artikel bei denen zB das Bild fehlt? Bei denen einer der Weblinks fehlt?
- Gibt es noch weitere Felder die 'nice to have' sind und jetzt fehlen wie zB Websiten etc? die könnte man auch optional machen...
- Nur um sicher zu gehen, die Referenz-Sequenz kann man zB von hier sich besorgen, richtig? Ich möchte da nichts falsch machen.
- Eine weitere Frage, statt auf diese Seite mit allen Untergruppen zu verweisen koennte man auch direkt auf die Seite des Organismus verweisen, was denkst du ist besser? Ich denke, beides hat Vor und Nachteile, beim einen sieht man die Verwandtschaft besser, beim anderen kann man direkt auf die weitern Links klicken.
- Ebenfalls sinnvoll könnte eine Auflistung aller Untergenera in der Box sein, so wie bei Humane_Papillomviren
- Gruss -- hroest Disk 13:55, 6. Mai 2009 (CEST)
- Ich fange mal von hinten an. Zu 7: Humane Papillomviren ist eigentlich kein Taxon, sondern ein Kompromiss, deswegen sind hier wahrscheinlich die Gattungen aufgezählt. Eigentlich gehört jedes untergeordnete Taxon dann in den Abschnitt "Systematik" innerhalb des Artikels. Würde man sie aufnehmen, dann könnte das bei manchen Familien eine riesige Liste werden, entspr. bei Ordnungen lange Familienlisten. Ok dann lassen wir es so; das Problem ist nur, dass mit der neuen Box eine solche Auflistung dann nicht mehr möglich sein wird
- zu 6: Den Klick zur Listenform auf NCBI fände ich besser, da man bei einigen Taxa gar nicht alle untergeordneten Spezies oder Subtypen aufzählen kann, da hätte man gleich eine aktuelle Liste und mit einem Klick auf eines davon, wäre man bei der Info-Seite. Die Listenform hätte für Nicht-Virologen wahrscheinlich auch mehr Nährwert. Ok dann lassen wir es so
- zu 5: Prinzipiell würde die Ref-Sequenz nur bei Boxen für Spezies/Subtypen/Isolaten Sinn machen. Meistens gibt es nur ein Referenz-Genom, das ist bei deinem Beispiel VZV anders. Ganz gut wären hier die Links auf z.B. GenBank: X04370.1 (von hier gibt´s nämlich weitere Links zu PubMed, Blast etc. wie gesagt, die ist optional, ich war nur verwirrt, denn hier gibt es NC_001348 und X04370.1 aber die beziehen sich auf dasselbe Paper, sind also identisch?
- Wenn es eine Methode gibt, das herauszufinden, sollte es in der Doku erwähnt werden. --Ernsts (Diskussion) 18:12, 9. Dez. 2018 (CET)
- zu 4: Für manche Gruppen (z.B. Pflanzenviren) gäbe es besondere Verzeichnisse, aber die könnte man dann besser bei Weblinks reinsetzen. Ein Parameter wäre noch zu bedenken, das Wirtsspektrum. Im ICTV-Report gibt es eine entsprechende mehrseitige Tabelle aller Familien und Gattungen, in der unter "Host" die folgende Einteilung vorgenommen wird: Bakterien (B), Archaeen (Ar), Vertebraten (V), Invertebraten (I), Algae (Al), Fungi (F), Pflanzen (P), Protozoen (Pr). Bei vielen Taxa tauchen hier mehrere Wirtskürzel auf, beui den meisten nur eines. Erst hatte ich mit überlegt, dies durchgehend per Kategorien zu lösen, da es die Kategorien Bakteriophage und Pflanzenvirus bereits gibt. Vielleicht ist das dann auch zu viel für eine Taxobox und die Kats wären besser.
- ja wir sollten die taxobox schlank halten, andererseits wäre es schon interessant das gleich auf einen Blick zu haben :-). Die Pflanzen können gut in die Weblinks, da können wir immer noch eine extra Vorlage basteln wenn wir wollen. Bei den Wirten sollten wir vielleicht noch Drittmeinungen einholen, es ist schwierig zu sagen was jetzt besser ist. Irgendann wird es zuviel -- hroest Disk 12:54, 7. Mai 2009 (CEST)
- Habe die neuen Ränge eingebaut nach ICTV Stand Nov. 2018, aber eben nur soweit sie tatsächlich auch benutzt werden (bis jetzt). Außerdem den Parameter Klassifikation (wie bei der Taxobox für Biota) mit Default Viren und weiteren mögl. Werten für Viroide und Satelliten. Sollten mal wirklich noch weitere gebraucht werden, dann kann man immer noch erweitern nach Schema F. Details s.u.--Ernsts (Diskussion) 21:46, 10. Dez. 2018 (CET)
- Seitdem seitens ICTV fast alle für Viren definierten Ränge auch im Gebrauch sind, hatte ich auch die restlichen implementiert.--23:44, 1. Feb. 2022 (CET)~
- Habe die neuen Ränge eingebaut nach ICTV Stand Nov. 2018, aber eben nur soweit sie tatsächlich auch benutzt werden (bis jetzt). Außerdem den Parameter Klassifikation (wie bei der Taxobox für Biota) mit Default Viren und weiteren mögl. Werten für Viroide und Satelliten. Sollten mal wirklich noch weitere gebraucht werden, dann kann man immer noch erweitern nach Schema F. Details s.u.--Ernsts (Diskussion) 21:46, 10. Dez. 2018 (CET)
- ja wir sollten die taxobox schlank halten, andererseits wäre es schon interessant das gleich auf einen Blick zu haben :-). Die Pflanzen können gut in die Weblinks, da können wir immer noch eine extra Vorlage basteln wenn wir wollen. Bei den Wirten sollten wir vielleicht noch Drittmeinungen einholen, es ist schwierig zu sagen was jetzt besser ist. Irgendann wird es zuviel -- hroest Disk 12:54, 7. Mai 2009 (CEST)
- zu 3: Die Biologen führen per Tool Listen über Artikel mit fehlenden Bildern, da werden unsere Viren auch hübsch aufgelistet. Ein Suchtool für fehlende Ref- oder taxolinks könnte man basteln (lassen). Ok dann braucht es das ja nicht
- zu 2: Die bisherigen Alternativen sind ja schon hier genannt, ich würde das nicht einschränken, da man auf Überraschungen bei noch zu klassifizierenden oder zu entdeckenden Viren gefasst sein sollte. Ok dann lassen wir das mal, wir können immer noch jemand anderen Fragen, das Problem ist eben nur, dass man irgendwas reinschreiben kann - müssen wir dann halt von Hand kontrollieren
- zu 1: Hmm, schwierig. Ich bin mir nicht ganz sicher, welchen Vorteil die Optionalität hätte außer, dass man ohne gewisse daten keine Box reinkleben kann. Kann ich nicht einschätzen.
- Andere Boxen machen das auch so und eine leere Box ist auch nicht so lustig :-) dann schreibt man lieber einen halben Satz statt eine Box hinzupflastern in der nicht mal der Name steht...deshalb dachte ich, Name und Genom sollte schon ausgefüllt sein. Vor allem sieht die Box dann auch nicht so hübsch aus :-) -- hroest Disk 12:54, 7. Mai 2009 (CEST)
- Noch ein Gedanke von mir: Bei der Chemobox sind einige Links auf Standardseiten etwas kleiner geschrieben, überhaupt fände ich es schicker, wenn unsere Schrift insgesamt eine Nuance kleiner ist, da man bei manchen Taxa extrem breite Taxoboxen produziert. Soweit meine Gedanken dazu, viellciht fällt uns noch as ein, es eilt ja nicht. Wenn wir es für akzeptabel halten, sollten wir bei den Biologen anklopfen, dass die von der Renovierung auch was mitbekommen bevor wir dann umstellen. Grüße, --Gleiberg 10:24, 7. Mai 2009 (CEST)
- zu deiner letzten Frage, da denke ich, dass es durchaus möglich ist aber Momentan verwende ich den CSS style mit dem Namen taxobox, das garantiert dass alle Taxoboxen gleich aussehen. Wenn wir davon abweichen sollten wir das vorher vielleicht mit den anderen besprechen. Bis dahin mache ich mir weiter Gedanken, was man noch verbessern könnte... Gruss -- hroest Disk 12:54, 7. Mai 2009 (CEST)
- Name sollte obligatorisch sein, Genom wohl auch (zumindest ob RNA oder DNA sollte wohl immer klar sein. Außer es gäber Viren mit beidem, aber halt: Retroviren/Pararetroviren. Weiß man es da immer? --Ernsts (Diskussion) 18:12, 9. Dez. 2018 (CET)
´n paar kleine Bugs im Feldversuch
[Quelltext bearbeiten]| Arenaviridae | ||||||||
|---|---|---|---|---|---|---|---|---|
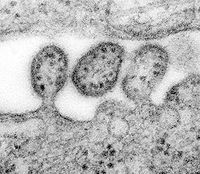
Knospung des Lassa-Virus | ||||||||
| Systematik | ||||||||
| ||||||||
| Taxonomische Merkmale | ||||||||
| ||||||||
| Wissenschaftlicher Name | ||||||||
| Arenavirus | ||||||||
| Links | ||||||||
|
Bei Arenaviridae wollte ich mal die neue Vorlage ausprobieren. Dazu habe ich dann Nebenstehendes reingetippt und die Vorschau angeschaut. Problem 1: Da der Artikel Arenaviridae heißt, erscheint der Name bei "Familie" wegen des selbstreferentiellen Links fett. Problem 2: Im Bild ist ja nicht "Arenaviridae" zu sehen, sondern als Beispiel das Lassa-Virus. Was muß ich da machen? Wird die erste Bildunterschrift automatisch von der Box-Beschriftung übernommen? Problem 3: "Arenavirus" ist ein Redirect auf diesen Artikel (da monogenerische Familie). Sollte man einen Link besser frei wählen können? Sind nur Kleinigkeiten, aber ich wollte deine Vorlage einfach mal von der Leine lassen :-) Grüße Christian --Gleiberg 23:29, 9. Jun. 2009 (CEST)
- Hallo, ich habe mal unter Infobox Virus/Testvorlage die ersten beiden Punkte behoben, wenn du also diese Vorlage in den Artikel kopierst dann wird kein fetter Link mehr erscheinen. Allerdings hast du Recht, es wäre eventuell sinnvoll, die Link selber zu wählen weil dann allerlei mühsame Dinge verhindern werden können. Die Idee wäre dann, dass man einfach statt Familie = Arenaviridae entweder schreibt Familie = [[Arenaviridae]] um den Link zu haben oder Familie = Arenaviridae um eben keinen Link zu haben; hast du dir das so gedacht? Gruss -- hroest Disk 10:42, 23. Jun. 2009 (CEST)
- Ja, genau, das wäre etwas flexibler. Danke und Gruß --Gleiberg 10:48, 23. Jun. 2009 (CEST)
- eine weitere Frage: sollen die '' auch von User gesteuert werden können oder nicht? also man kann es auch so machen, dass man für das volle Programm dann Familie = ''[[Arenaviridae]]'' schreiben kann, das würde helfen, Dinge wie "nicht klassifiziert" nicht auch kursiv zu haben - wobei sich die Frage stellt wie oft das vorkommt und wie nützlich das dann ist. Gruss -- hroest Disk 11:42, 23. Jun. 2009 (CEST)
- Bei Ordnungen wäre es egal, da diese ja stets nicht-kursiv geschrieben werden. Es gibt aber eine ganze Reihe von Gattungen ohne Familienklassifizierung, so dass man das am besten auch frei entscheiden lassen sollte, sonst steht bei Familie das "nicht klassifiziert" in kursiv (wenn ich das richtig verstanden habe). --Gleiberg 12:32, 23. Jun. 2009 (CEST)
- Ja genau, dann werde ich das dahin ändern - aber dann mache ich alle gleich, nicht dass man bei Familien die '' einfügen muss und bei Gattung nicht, sonst wird das etwas schwierig, denkst du nicht? Gruss -- hroest Disk 13:36, 23. Jun. 2009 (CEST)
- Bei Ordnungen wäre es egal, da diese ja stets nicht-kursiv geschrieben werden. Es gibt aber eine ganze Reihe von Gattungen ohne Familienklassifizierung, so dass man das am besten auch frei entscheiden lassen sollte, sonst steht bei Familie das "nicht klassifiziert" in kursiv (wenn ich das richtig verstanden habe). --Gleiberg 12:32, 23. Jun. 2009 (CEST)
- eine weitere Frage: sollen die '' auch von User gesteuert werden können oder nicht? also man kann es auch so machen, dass man für das volle Programm dann Familie = ''[[Arenaviridae]]'' schreiben kann, das würde helfen, Dinge wie "nicht klassifiziert" nicht auch kursiv zu haben - wobei sich die Frage stellt wie oft das vorkommt und wie nützlich das dann ist. Gruss -- hroest Disk 11:42, 23. Jun. 2009 (CEST)
- Ja, genau, das wäre etwas flexibler. Danke und Gruß --Gleiberg 10:48, 23. Jun. 2009 (CEST)
- OK, ich habe es so gelöst, dass man immer die [[ und ]] angeben muss aber niemals die '', ich denke das sollte möglich sein. Gruss -- hroest Disk 23:41, 27. Jun. 2009 (CEST)
- Habe das in diesem Sinn auf die neuEn Taxon-Stufen des ICTV erweitert.--Ernsts (Diskussion) 00:04, 30. Nov. 2018 (CET)
Variable Breite der Box?
[Quelltext bearbeiten]| Arenaviridae | ||||||||
|---|---|---|---|---|---|---|---|---|

Knospung des Lassa-Virus | ||||||||
| Systematik | ||||||||
| ||||||||
| Taxonomische Merkmale | ||||||||
| ||||||||
| Wissenschaftlicher Name | ||||||||
| Arenavirus Dies ist ein Test ... Dies ist ein Test ... Dies ist ein Test ... Dies ist ein Test ... Dies ist ein Test ... | ||||||||
| Links | ||||||||
|
Ist es eigentlich Absicht, daß die Box mit der Länge der Textzeilen, wie in diesem Beispiel, immer breiter wird?
Das Phänomen tritt übrigens auch bei der Parameterzeile „Name =” auf. Gruß, --Jochen (Diskussion) 09:50, 4. Sep. 2013 (CEST)
- Diese Box hatte (im Gegensatz zur verwandten Vorlage:Taxobox) keine festgelegte Breitenangabe. Ich habe jetzt deren Breite als Obergrenze für diese Vorlage eingebaut. Gruß --Entlinkt (Diskussion) 17:56, 19. Apr. 2014 (CEST)
- Hm, scheint bei iDevices nicht zu funktionieren (iPad, klassische = Desktop-Ansicht)? Die Box ist total breit. Macht man da aber explizite br's rein, wird am PC alles zerpflückt, Beispiel Negarnaviricota. Gruß --Ernsts (Diskussion) 17:59, 9. Dez. 2018 (CET)
Der Dienst ICTVdB ist eingestellt
[Quelltext bearbeiten]Der Dienst ICTVdB wurde eingestellt und der dazugehörige ICTVdB Virus Code ist somit veraltet. Könnte der auch aus der Vorlage raus genommen werden? --Kim Holger Kelting • Diskussion 22:24, 6. Dez. 2015 (CET)
- Nein, auf keinen Fall. Die Kennziffer behält ihre Gültigkeit und ist keine Ziffer für die Webpage-Navigation. --Gleiberg (Diskussion) 10:52, 10. Feb. 2016 (CET)
- Dieser Revert meiner Parameterwartung, die nichts mit obiger Kennziffer zu tun hat, ist unsinnig. Du müsstest die beiden vorhergehenden Änderungen revertieren. --Leyo 14:51, 10. Feb. 2016 (CET)
- Folglich wird trotz deines Reverts in Retroviren die genannte Kennziffer in der Infobox nicht aufgeführt. Was ist am sinnvollsten, unverlinkt anzeigen? --Leyo 01:36, 12. Feb. 2016 (CET)
- Man kann z. B. im Web-Archiv weiter nach der ICTVdb Nummer recherchieren. Beispiel Hirse-Mosaik-Satelliten-Virus mit Treffer ICTVdB — The Universal Virus Database: Panicum mosaic satellite virus --Ernsts (Diskussion) 15:33, 8. Dez. 2018 (CET)
- @Leyo:Der Link könnte wenn gewünscht so reaktiviert werde, dass automatisch eine Suche im Webarchiv gestartet wird.
{{#if: {{{ICTV<includeonly>|</includeonly>}}} |
{{!}} [[International Committee on Taxonomy of Viruses|ICTVdB Virus Code]]:
{{!}} [https://web.archive.org/web/*/https://www.ncbi.nlm.nih.gov/ICTVdb/ICTVdB/{{{ICTV}}}.htm {{{ICTV}}}]
}}
- Über einen Zusatzparameter (Archivdatum) könnte dann auch gleich ein Verweis auf ein Memento erzeugt werden, indem das * durch dieses Datum ersetzt wird.--Ernsts (Diskussion) 21:23, 22. Jan. 2021 (CET)
- Habe einen Update auf die Vorlage vor, ursprünglich wegen Verbesserungen zur Kursivsetzung. Siehe Wikipedia Diskussion:Redaktion Biologie# Frage zur Kursiv- oder Aufrechtschreibung von Taxa. Habe beim Testen auf der Spielwiese auch einen Link aufs Webarchiv implementiert, dass eine Archivierung der jeweiligen ICTVdb-Seite aus den 200x-er Jahren sucht, der Dienst wurde ja Augst 2013 engestellt. Vielleicht mal probieren, wenn's nicht guttut, kann der Link ja wieder weg. --Ernsts (Diskussion) 19:16, 17. Jan. 2022 (CET)
- Über einen Zusatzparameter (Archivdatum) könnte dann auch gleich ein Verweis auf ein Memento erzeugt werden, indem das * durch dieses Datum ersetzt wird.--Ernsts (Diskussion) 21:23, 22. Jan. 2021 (CET)
- Irgendwie habe ich im Januar 2021 den Ping nicht erhalten. Mittels
http://timetravel.mementoweb.org/list/2013/https://www.ncbi.nlm.nih.gov/ICTVdb/ICTVdB/{{{ICTV}}}.htmkönnte man einen Link auf eine Archivversionsuche setzen. Aber ob dies sinnvoll ist, ist eine andere Frage … --Leyo 22:12, 17. Jan. 2022 (CET)- Habe auf Vorschlag von Succu die Anzeige des Parameters mit dem neuen Update (s. u.) herausgenommen. Grund: die Infos sind inzwischen völlig veraltet. Grüße--Ernsts (Diskussion) 23:55, 1. Feb. 2022 (CET)
- Irgendwie habe ich im Januar 2021 den Ping nicht erhalten. Mittels
ICTV Stand Nov 2018
[Quelltext bearbeiten]Das ICTV hat neue Gruppen eingeführt wie z. B. Untergattung, Unterfamilie, Unterordnung, Klasse, Subphylum, Phylum. Bitte einbauen zumindest soweit diese tatsächlich auch vom ICTV bereits benutzt sind (das sind die hier genannten).
Weiter sollten auch die Namen der höheren Ränge (z. B. Ordnung, Unterordnung,...) automatisch kursiv dargestellt werden, wenn ich das ICTV richtig verstehe.
Die Benenung 'Reich' bei Reich: Viren ist obsolet/inkorrekt.
- Die Benennung als Reich (engl. kingdom) stellt die Viren als Gruppe auf gleiche Ebene wie Pflanzen, Tiere und Pilze. Darüber steht bei den Lebewesen (Biota) aber noch die Domäne (domain oder superkingdom). Die Viren (bzw. der Begriff Virosphäre) sollte auf derselben Stufe stehen wie Lebewesen/Biota (bzw. der Begriff Biosphäre). (Es wird sogar diskutiert, eine Gruppe der Viren (NCLDV) als Domäne aufzufassen)
- Das ICTV hat 2018 (Herbst) neue Ränge eingeführt, realm (Zone?), subrealm (Unterzone?), kingdom (Reich), subkingdom (Unterreich), phylum (Stamm), subphylum (Unterstamm), class (Klasse), subclass (Unterklasse), ... mit entsprechenden Namenskonventionen, insbesondere ...viriae für kingdom (Reich). Auch wenn aktuell für die hohen Ränge (oberhalb von Phylum) noch keine konkreten Namen vergeben sind, zeigt dies, in welche Richtung das ICTV gehen will: Innerhalb der Viren könnte es verchiedene kingdoms geben. Wenn überhaupt, dann käme real = Viuses (Viren) in Frage. Vielleicht will das ICTV aber auch in der Taxomomie berücksichtigen, dass die Viren als Gruppe (sehr wahrscheinlich) nicht momophyletisch sind. Die Zusammenfassung als 'Viren' wäre dann nur eine informelle Klassifizierung.
Bitte daher die Bezeichnung der Viren als 'Reich' entfernen oder gleich die ganze Zeile aus den Boxen entfernen.
Vielen Dank! --Ernsts (Diskussion) 09:57, 29. Nov. 2018 (CET)
Habe die für den aktuellen Stand notwendigen Ergänzungen/Änderungen selbst in die 'Infobox Virus' eingebracht:
- Zusätzliche Rangstufen (ab Phylum abwärts)
- generell Schrägschrift. Nebeneffekt: Vorgeschlagene Taxa in einfachen Hochkommata erscheinen fett. Man muss da wohl oder übel ‚‘ benutzen. Beispiel: Viren der Gruppe NCLDV = ‚Megavirales‘
- Die Angabe ‚nicht klassifiziert‘ ist jetzt bis auf (Sub)Species überall möglich. Warum nicht?
- Wg. Reich: Viren und Einführung der noch fehlenden hohen Rangstufen bitte Diskussion :-) .
--Ernsts (Diskussion) 19:05, 29. Nov. 2018 (CET)
@Gleiberg, Gerbil: Meinungen hierzu? Grüße, --Ghilt (Diskussion) 00:19, 30. Nov. 2018 (CET)
- Noch ein Zusatz: Das ICTV hat ebenso eine Taxonomie von Viroiden und jetzt aktuell erstmals auch eine von Satelliten(viren) veröffentlicht. Die benutzten Stufen sind jeweils ein Subset der der Viren, die Endungen enthalten im Wesentlichen den String viroid bzw. satellit anstelle von vir. Prinzipiell könnte die Infobox Virus also auch für Viroide und Satelliten verwendet werden, wäre da nicht die fragliche feste Überschrift Reich:Viren. Eine einfache Lösung wäre wohl, das Ganze flexibel zu machen mit einem weiteren Parameter, Wert entweder Viren (Default wenn nichts angegeben), Viroide oder Satelliten. Aber vielleicht gibt es bessere Vorschläge?
- Die Verwendung des Begriffs Reich ist klar nicht ICTV konform, Reich ist in der Bio englisch kingdom und das ist nach den neuen Regel bereits vergeben, wenn auch nicht benutzt. Es ist jedenfalls nicht die höchste Rangstufe (da sich diese Gegenüber LHT von 1962 verschoben haben). Vielleicht hat das ICTV ja realm (etwa: Bereich , Zone, Sphäre im Sinn von Viro-/Biosphäre) im Sinn.
- Gibt es schon eine etablierte Übersetzung von realm? Wäre diese nicht Bereich, dann wäre die Infobox-Überschrift mit Bereich eh nur informell/klassifizierend. Wäre sie gleich Bereich, dann wäre der Name ein Taxon und Werte wie Viren oder Satelliten müssten nach der reinen Lehre in Anführungszeichen - weil das zum einen nur von der Qualität Vorschlag, vor allem aber weil Viren sehr wahrscheinlich ployphyletische Ursprung sind und damit kein Taxon, sondern eine Klassifizierung (das dürfte auch der Grund sein warum sich das ICTV mit Namen vom Rang realm zurückhält).
- Mein Vorschlag: Reich ersetzen durch Bereich und das als neuen optionalen Parameter einführen mit Voreinstellung Viren und 3 möglichen Werten:Viren, Viroide und Satelliten, in der Anzeige automatisch hinterlegt mit einem QV auf Viren bzw. Viroid oderSatellit (Biologie) und ggf. in Hochkommata).
--Ernsts (Diskussion) 19:46, 2. Dez. 2018 (CET)
Habe diese Änderung (Reich zu Klassifikation - mit QV wie in Vorlage:Taxobox und die Erweiterung (optionaler Parameter Klassifikation mit erlaubten Werten Viren (Standard), Viroide, Satelliten realisiert. Die erlaubten Werte werden automatisch ebenfalls mit einem QV versehen, aber nicht in irgendwelche Anführungszeichen gesetzt. Fehlerbehandlung für unzulässige Werte (Bitte korrigieren wenn es dazu einen abweichenden Wikipedia-Standard geben sollte. Ist meine erste editierte Vorlage hier). Keine Anführungszeichen, auch weil nicht klar ist welche (siehe Diskussion zu NCLDV). Die vorübergehende Idee, Bereich statt Reich zu verwenden habe ich zugunsten einer besseren Übereinstimmung mit der Taxobox (siehe etwa Lokiarchaeota) wieder fallengelassen. Auch wird dadurch ein möglicher künftiger Konflikt vermieden, sollte sich für den ICTV-Taxonomie.Rang realm die deutsche Übersetzung Bereich (statt Zone, Sphäre,...) etablieren. Da man von einer monophyletischen Abstammung der Gruppen Viren, Viroide und Satelliten kaum ausgehen kann, stellen diese Gruppen tatsächlich eine Klassifikation dar (vermutlich im Gegensatz zur Klassifikation Lebewesen in der Taxobox unter der Annahme eines LUGA, ggf. in der RNA-Welt, aber ‚lebend‘).
Da es bisher keinen Widerspruch gab, hoffe ich dem allgemeinen Verständnis einigermaßen entgegengekommen zu sein. :-) --Ernsts (Diskussion) 18:35, 4. Dez. 2018 (CET)
- Das ICTV hat in der Master Species List 2018b.v1 (MSL #34) noch höhere Ränge vergeben, darunter Realm (z. B. Riboviria). Die macht es nötig, diese Ränge in der Infobox virus zur Verfügung zu stellen. --Ernsts (Diskussion) 17:15, 6. Mai 2019 (CEST)
Symmetrie Verweis auf Kapsid
[Quelltext bearbeiten]Der Default (wenn keiner der gelisteten Begriffe zutrifft) verweist auf Kapsid, der Titel dieser Zeile auch. Das ist zum einen überflüssig. Zum anderen beschränkt es die Freiheit des Bearbeiters, da z. B. die Angabe einer Referenz suboptimal rendert. Werde das daher entfernen. Beispiel: Mimiviridae kommt dann gleich. Hoffe, das ist ok so. MfG --Ernsts (Diskussion) 21:49, 15. Jan. 2019 (CET)
Subspezies nicht kursiv
[Quelltext bearbeiten]- 'Subspezies' ist kein vom ICTV vergebener Rang
- das ICTV listet in seiner Master Species List keine Ränge unterhalb von Species
- Gemäß ICTV werden 'Virusnamen' im Gegensatz zu Speziesnamen, Gattungsnamen und höher niemals kursiv geschrieben – selbst wenn sie einen solchen (Speziesnamen) beinhalten.
Verstehe ich richtig, dass 'Virusname' hier eine Linie, Isolat, en. 'strain' meint? Und wird in der Praxis der Parameter Subspezies nicht genau in diesem Sinn verwendet?
==> Wenn es so ist, sollte dann der Wert dieses Parameters nicht automatisch kursiv gesetzt werden, oder?
Was sind eure Meinungen dazu?
Beispiel: Humanes Enterovirus 71
Referenz: [2] Zitat: "A virus name should never be italicized" (Im gegensatz zu Spezies-, Gattungs-, Familien-... -namen) Beispiel: Sida ciliaris golden mosaic virus (SCGMV) - nicht kursiv (keine Spezies, sondern darunter) --Ernsts (Diskussion) 10:09, 29. Jan. 2019 (CET)
- Im Allgemeinen bezeichnen wir mit Virusspezies das Virus, nicht den Stamm. Es gibt Artikel zu prominenten Stämmen, wie das Beispiel. Grüße, --Ghilt (Diskussion) 11:29, 29. Jan. 2019 (CET)
- Von mir aus lassen wir es bis auf weiteres so wie es ist. --Ernsts (Diskussion) 20:03, 12. Apr. 2019 (CEST)
- neue Idee: einen Schalter für Nicht-kursiv: Titel, Name und Subspezies, siehe nächsten Abschnitt.--Ernsts (Diskussion) 06:01, 21. Sep. 2019 (CEST)
- Von mir aus lassen wir es bis auf weiteres so wie es ist. --Ernsts (Diskussion) 20:03, 12. Apr. 2019 (CEST)
Name und Titel kursiv
[Quelltext bearbeiten]Nach Durchsicht scheint nirgends der Name in der Virusbox kursiv gesetzt zu sein, so wie hier im Beispiel Rubellavirus. Genauso der Seitentitel. Das entspricht nicht den ICTV-Regeln für Virustaxa. Statt alle Virusartikel entsprechend zu ändern könnte man hier auch den Parameter Name in der Boxüberschrift kursiv setzen, ebenso den Seitentitel. Nur im Namensraum:
- {{SEITENTITEL:''{{PAGENAME}}''}}
Auf Benutzerunterseiten alles kursiv:
- {{SEITENTITEL:''{{#rel2abs: .}}''}}
Am besten:
- {{SEITENTITEL:{{#ifeq:{{#rel2abs: ..}}|||{{#rel2abs: ..}}/}}''{{SUBPAGENAME}}''}
oder gleichwertig
- {{SEITENTITEL:{{#ifeq:{{#titleparts: {{FULLPAGENAME}}|-1}}|||{{#titleparts: {{FULLPAGENAME}}|-1}}/}}''{{SUBPAGENAME}}''}}
(das funktioniert auch in Benutzerunterseiten).
Für die wenigen nicht-taxonomische Namen könnte das über einen weiteren Parameter abschaltbar gemacht werden, wobei die Subspezies dann zweckmäßigerweise ebenfalls nicht kursiv zu setzen wäre.
Würde mir das zutrauen zu implementieren. Eure Meinungen? --Ernsts (Diskussion) 02:10, 21. Sep. 2019 (CEST)
- Hi Ernsts, von mir aus gerne, Grüße, --Ghilt (Diskussion) 16:50, 21. Sep. 2019 (CEST)
- Hi Ghilt, danke für den Zuspruch! habe auf rel2abs verzichtet zugunsten der Benutzung fertiger Variablennamen wie FULLPAGENAME. Habe zudem den Default für Name geändert von PAGENAME auf SUBPAGENAME für Benutzung der Virusbox in Baustellen unter der eigenen Kennung, damit nur der eigentliche Name (SUBPAGENAME) angezeigt wird (insbesondere wenn kursiv). Hoffe, das liefert so immer das Gewünschte. Doku kommt. --Ernsts (Diskussion) 12:31, 23. Sep. 2019 (CEST)
- Musste noch ein Änderung machen für alle Viruskrankheiten mit einer Virusbox zum Erreger, wie etwa Pest. Habe nicht herausgefunden, wie ich in der Vorlage den Namen des Abschnitts herausfinden kann, und lasse jetzt überprüfen, ob der Name (Box-Überschrift) mit dem Lemma übereinstimmt. Kursiv nur wenn ja. (dabei ist der Default-Name = Lemma). Hoffe das passt so. --Ernsts (Diskussion) 15:56, 23. Sep. 2019 (CEST)
- Pest ist keine Viruskrankheit. Krankheiten brauchen zudem keine Virobox. --Ghilt (Diskussion) 15:59, 23. Sep. 2019 (CEST)
- Sorry, da hab ich mich hier vertan, die Pesterreger sind nätürlich die Yersinien Yersina pestis. Gemeint waren die Pocken mit dem Erreger Variola virus (Sorry für die Verwirrung). Dieser hat kein eigenes Lemma (schön wäre es!), wie bei einigen anderen Viruskrankheiten findet sich die einzige Beschreibung des Virus bei der verursachten Krankheit. Eine saubere Lösung wäre ein eigenes Lemma, aber wer macht's? Daher habe ich die Taxobox der (bedauerlichen) Realität angepasst, so dass in diesen Fällen das Lemma (Name der verursachten Krankheit) nicht kursiv geschrieben wird, auch wenn beim Erreger eine (kursiv zu schreibende) Spezies oder Gattung angegeben ist. --Ernsts (Diskussion) 11:52, 24. Sep. 2019 (CEST)
- Pest ist keine Viruskrankheit. Krankheiten brauchen zudem keine Virobox. --Ghilt (Diskussion) 15:59, 23. Sep. 2019 (CEST)
- @Ghilt:Hier steht ein Update an, siehe Wikipedia Diskussion:Redaktion Biologie# Frage zur Kursiv- oder Aufrechtschreibung von Taxa. Grüße --Ernsts (Diskussion) 19:04, 17. Jan. 2022 (CET)
Mehrere NCBI- und ICTV_Tax-IDs
[Quelltext bearbeiten]@Ghilt:@Lómelinde: Habe aktuell in der Vorlage:Spielwiese einen Vorschlag für eine neue Erweiterung der Virusbox. Bei Durchsicht der Artikel mit Referenz auf die Virusbox (um fehlede IDs nachzutragen) bin ich auf etliche Beispiele gestoßen, wo mehrere Spezies/Gattungen in einem Artikel behandelt werden. Prominente Beispiele sind HIV und Influenzavirus, aber es gibt noch einige mehr.
- Meist sind es Spezies, die vom ICTV irgendwann aufgeteilt wurden (was öfters vorkommt), etwa: Herpes-simplex-Viren 1 und 2, Adeno-assoziierte Viren A und B, Virus der Bornaschen Krankheit 1, 2(?), Coxsackie-Virus, Bovines Virusdiarrhoe-Virus, GB-Viren, Rhinovirus A, B, C ---Ernsts (Diskussion) 14:12, 24. Sep. 2019 (CEST)
Eigentlich sollte das nicht sein, aber es macht in der Praxis nicht immer Sinn, mal abgesehen vom Arbeitsaufwand. Das gilt insbesondere eben für HIV, ähnlich beim Influenzavirus. Bei HIV gäbe es einfach zu viel doppelt, beim Influenzavirus wäre es zumindest viel Arbeit (im Vergleich zu einer erhofften Verbesserung).
Habe mich daher schon seit einiger Zeit gefragt, ob es technisch möglich wäre, die Virusbox so aufzubohren, dass die Angabe mehrerer IDs korrekt in entsprechende URLs umgesetzt wird. Habe jetzt (endlich :-) ) eine Lösung gefunden, siehe Spielwiesenvorlage.
Die Erweiterung ist voll abwärtskompatibel: nur wenn in der ID ein Komma vorkommt, geht es in den neuen Zweig (programmtechnich also ein 'Balkon'). Hinter der ID kann optional in Klammern ein Hinweis (etwa Acronym) angegeben werden. Beispiel zu HIV:
| Humanes Immundefizienz-Virus | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
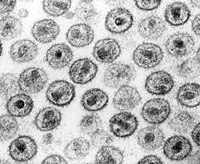
HI-Viren im TEM | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Taxonomische Merkmale | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Human immunodeficiency virus | ||||||||||||
| Kurzbezeichnung | ||||||||||||
| HIV-1, HIV-2 | ||||||||||||
| Links | ||||||||||||
|
Bisher sind nur die IDs der Typusspezies HIV-1 angegeben. Trotz der vollen Abwärtskompatibiität wolle ich Euch (und alle anderen) gerne erst um Eure Meinung fragen, bevor ich's einbaue. :-) Viele Grüße --Ernsts (Diskussion) 12:16, 24. Sep. 2019 (CEST)
- Sorry, ist nicht mein Arbeitsbereich, da kann ich wenig dazu sagen. Dazu müsste ich mich in die IB-Logik hineindenken. --Liebe Grüße, Lómelinde Diskussion 12:27, 24. Sep. 2019 (CEST)
- Danke trotzdem für die Stellungnahme. Habe das Coding jetzt veröffntlicht, da abwärtskompatibel und um die Spielwiesenvorlage wieder freizugeben. Strichpunkt statt Komma als alternativen Trenner (Rhinovirus). Für NCBI_Ref habe ich die Mimik ebenfalls implementiert, ViralZone fehlt noch.--Ernsts (Diskussion) 17:36, 24. Sep. 2019 (CEST)
- Habe in den oben genannten Beispielen die (multiplen) IDs in die Virusbox eingefügt. --Ernsts (Diskussion) 17:43, 24. Sep. 2019 (CEST)
- Da das betreffende Coding 4x benötigt wird, dieses in die neue Vorlage:MakeURLsFromIDs ausgelagert und dort ausgiebig kommentiert. Schließlich noch für ViralZone implementiert. ViralZone IDs für Infuenzavirus und HIV nachgetragen. Bitte Rückmeldung bei Auffälligkeiten :-) Vielen Dank! --Ernsts (Diskussion) 16:51, 25. Sep. 2019 (CEST)
Belege innerhalb der Infobox Virus (z. B. NCBI_Ref)
[Quelltext bearbeiten]Hallo zusammen, im Artikel 2019-nCoV werden Belege mit ref-tags innerhalb der Infobox Virus verwendet, ich habe das auch schon in anderen Artikeln über Viren gesehen. Als ich im genannten Artikel letztens weitere Einträge im Feld NCBI_Ref vorgenommen habe, wollte ich das mit einem Beleg koppeln, was aber nur halbwegs funktioniert, wenn man ein Leerzeichen vor das ref-tag setzt. Als Aka das überflüssige Leerzeichen entfernte (siehe hier), war der letzte Eintrag im Feld NCBI_Ref nicht mehr als Weblink abrufbar. Den Sachverhalt habe ich auf der Seite Benutzer Diskussion:Aka#Leerzeichen vor Beleg entfernt beschrieben, und Aka und PerfektesChaos haben dort bereits geantwortet. Folglich würde ich den Beleg aus der Infobox Virus entfernen, möchte aber an dieser Stelle nachfragen, ob die Infobox nicht so geändert werden kann, so dass man Eintragungen bei dem Parameter NCBI_Ref auch mit einem Beleg versehen kann? Viele Grüße, --A doubt (Diskussion) 18:07, 6. Feb. 2020 (CET)
- @A doubt:Habe eine Lösung gefunden, die ohne das obskure Blank auskommt und in der Spielwiese mit SARS-CoV-2 getestet. Muss vorher noch mehr testen wegen anderer Änderungen. Siehe Wikipedia Diskussion:Redaktion Biologie#Frage zur Kursiv- oder Aufrechtschreibung von Taxa. Grüße --Ernsts (Diskussion) 19:08, 17. Jan. 2022 (CET)
ICTV Taxon ID
[Quelltext bearbeiten]Habe jetzt bei (hoffentlich) allen offiziellen Virus-Taxa die ICTV Taxon ID ergänzt. Geht zurück auf einen alten Tipp von User:Succu. Diese sollte bei allen offiziellen Taxa angegeben sein. Die Vorschläge könnten dagegen wie bereits gelegentlich diskutiert in (doppelten) AFZ eingeschlossen werden.
Aus der Diskussion mit User:Wurgl:
Vielleicht ein nice-to-have für die Vorlage: Genau dann, wenn der ICTV_Tax Parameter nicht versorgt ist, sollte der Name in AFZ stehen (Ausnahme Subspezies). Wenn nicht, dann könnte vielleicht eine Vorlagen-Warnung ausgegeben werden. Natürlich müsste es eine Anleitung geben, wie man beim ICTV Taxonomy Browser die IDs abfragt (nach dem Taxon suchen, View, Spalte ganz rechts anklicken->Taxon History).
Alternativ automatische AFZ, wenn keine ICTV_Tax wäre vielleicht nicht so gut.... Zumal da aktuell meist explizit AFZ angegeben sind bei unbestätigten Taxa.--Ernsts (Diskussion) 22:07, 22. Jan. 2021 (CET)
Kursiv- oder Aufrechtschreibung
[Quelltext bearbeiten]Klärung von Detailfragen in Fortführung von Wikipedia Diskussion:Redaktion Biologie#Frage zur Kursiv- oder Aufrechtschreibung von Taxa.
- Zusammenfassung des aktuellen Standes
- Taxa (Art bis Realm): Diese Felder sind den aktuellen von ICTV vorgegebenen wissenschaftlichen Bezeichnungen vorbehalten (jedenfalls für ICTV-bestätigte Taxa). Nach ICTV-Regeln sind diese kursiv zu schreiben, das geschieht automatisch.
- Virusnamen und Akronyme sind nach ICTV in Normalschrift zu schreiben.
- Feld Name ist reserviert für deutsche Bezeichnungen wie Nanoviren (Gattung), Nanoviriden (zu Unterscheidung der Familie, analog zu Hominiden), Xyz-Virus (statt en. Xyz virus) etc. werden wie in der Biologie zellulärer Organismen auch in Normalschrift gesetzt.
- Das Lemma sollte dann automatisch kursiv gesetzt werden, wenn es mit dem Wiss_Name (bzw. dem niedrigsten spezifizierten Rang, s. u.) übereinstimmt und nicht kursiv=nein angegeben ist.
- Grund: Es gibt einige Artikel über Viruskrankheiten mit Abschnitt Erreger (oder ähnlich) und einer eingebundenen Vorlage Infobox Virus. Das Lemma (die Krankheit) darf dann nicht kursiv gesetzt werden.
Ich glaube, soweit dürfte Klarheit bestehen.
- Thema Unterart/Stamm/Isolat
- Für Virusstämme und Isolate hat die Vorlage Infobox Virus keinen eigenen Parameter. Stattdessen bietet sie den Parameter Unterart/Subspezies. Das wäre en./lat. subspecies, was aber kein vom ICTV unterstützter taxonomischer Rang ist (es ist anders als in der sonstigen Biologie), sondern darunter. AFAIK war auch für diesen Parameter analog zur Biologie der Lebewesen automatisch kursiv eingestellt. Das ist sinnvoll, sollte sich das ICTV irgendwann entscheiden, nach der Empfehlung für Binomialnamen für Virusarten, auch Unterarten mit 3er-Namen zu unterstützen.
- Solange für Stämme und Isolate kein eigener Parameter geschaffen wird (das wollte ich meinerseits bisher nicht), muss für diese der Parameter Unterart herhalten; das war AFAIK damals schon gängige Praxis. Der Name darf dann aber nicht kursiv erscheinen. Das war der ursprüngliche Grund für mich, die Option kursiv=nein einzubauen.
Zur Diskussion stellen möchte ich daher:
- Wollen wir es dabei belassen, Unterart/Subspezies im allgemeinen Sinn zu verstehen und (auch) für Stämme/Isolate herzunehmen, Voreinstellung kursiv, aber abschaltbar?
- Plädiere selbst für ja, aber bitte... :-) – (Änderungen bedeuten aber weitere Arbeit)
- Thema Wiss_Name für Stamm/Isolat
- Es gibt keinen Parameter Rang für die mit dem Name bzw. Wiss_Name bezeichnete Gruppe. Die gängige Praxis ist, dass diese Gruppe übereinstimmt mit dem niedrigsten Rang angefangen mit Unterart/Subspezies(!), Art/Spezies bis hin zu Realm (der taxonomische Rang der beschriebenen Gruppe ist dann dadurch festgelegt).
- Genauer gesagt, stimmt für Art/Spezies bis Realm der Wiss_Name mit der unter dem niedrigsten Rang spezifizieren Bezeichnung überein (habe ein paar Ausnahmen gesehen, monotypische etwa oder Aufzählungen von Subtaxa). Unklar: Gilt das auch für Unterart/Subspezies.
Zur Diskussion stellen möchte ich daher:
- Falls das Lemma einen Stamm/Isolat bezeichnet, soll mit dem Wiss_Name dann (auch) die englische Bezeichnung verstanden werden?
- Plädiere selbst für ja (technisch einfacher da genauso wie bei den ICTV-unterstützen Rängen)
- Wenn der Wiss_Name üblicherweise mit dem niedrigsten spezifizierten Rang übereinstimmt, kann dieser als Default für Wiss_Name dienen (wenn das Feld Name nicht angegeben ist)?
- Finde selbst den Zwang zur doppelten Angabe unbefriedigend (ein Default wäre aber in jedem Fall gut auszutesten).
Hoffe, das alles war nicht zu kompliziert, es geht mir nur um eine genaue Beschreibung der Spezifikation, damit die Erwartungen dann auch erfüllt werden. Bitte Rückmeldung, wem meine Annahmen nicht plausibel erscheinen, und auch wenn zur Zusammenfassung noch Anmerkungen/Fragen etc. sind. :-)
Vielen Dank! --Ernsts (Diskussion) 15:55, 21. Jan. 2022 (CET)
- FYI, aus dem aktuellen Newsletter des ICTV, Zitat: „In March 2021, the ICTV ratified a proposal to adopt a standard two-part "binomial nomenclature" as the norm for naming virus species. Adoption of the binomial format is now mandatory for all new virus species names. Also, SC Chairs will liaise with the relevant Study Groups to retrospectively change the names of existing non-binomial speciesnames. The aim is to complete this process in time to be considered during the annual EC meeting in 2023. However, the new nomenclature is in its infancy and the EC has received many questions regarding the new format.“
- In einer/einigen Familien (Genomoviridae) wurden diese Änderungen bereits in MSL#36 ausgeführt, als Artepitheton ist bei der genannten Familie dann üblicherweise ein Kofferwort gewählzt worden, weder en noch lat. Wir müssen das so zur Kenntnis nehmen. Bin gespannt, wie es das ICTV löst, wenn die zehn- und hunderttausende an vorgeschlagenen Phagen offiziell werden ;-) Grüße --Ernsts (Diskussion) 18:03, 22. Jan. 2022 (CET)
- Falls keine Einwände kommen, werde ich die Änderungen wie in der Spielwiese dokumentiert ab Wochenmitte scharf machen. Brauche ein Zeitfenster, groß genug um alle Infoboxen danach zu prüfen. Grüße!--Ernsts (Diskussion) 12:22, 24. Jan. 2022 (CET)
- Werde die Änderungen jetzt implementieren. Danach alle Artikel im Namensraum mit der Infobox Virus überprüfen, ggf. nötige Korrekturen/Anpassungen vornehmen (kann bis einschl. morgen dauern). Zuletzt Aktualisierung der Doku (inklusive der eventuellen Anpassungen). Grüße! --Ernsts (Diskussion) 09:33, 26. Jan. 2022 (CET)