Salasmaviridae
| Salasmaviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
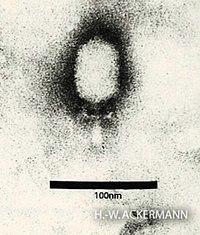
EM-Aufnahme eines Partikels von Bacillus-Phage Phi29-, Gattung Salasvirus (Picovirinae) | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Salasmaviridae | ||||||||||||||
| Links | ||||||||||||||
|


Salasmaviridae ist die Bezeichnung für eine 2021 vom International Committee on Taxonomy of Viruses (ICTV) neu eingerichtete Familie von Viren mit sog. Kopf-Schwanz-Aufbau (Klasse Caudoviricetes).[3] Die Verwandtschaft der Familienmitglieder und Zugehörigkeit zu einer gemeinsamen Klade wurde durch phylogenetische Analysen ermittelt.
Die Spezies der Gattung Salasvirus (Unterfamilie Picovirinae) besitzen ebenso wie die Spezies Bacillus-Virus Goe4, der Gattung Claudivirus (Unterfamilie Northropvirinae) im Gegensatz zu vielen anderen Caudoviricetes ein langgestrecktes, nicht-isometrisches Kapsid.
Historie
[Bearbeiten | Quelltext bearbeiten]Als Ergebnis einer umfangreichen Analyse der Genomsequenzen von Phagen, die (im weiteren Sinne) als Phi29-ähnlich beschrieben wurden und zunächst noch provisorisch der Unterfamilie Picovirinae (damals noch in der früheren Familie Podoviridae) zugeordnet waren, wurde diese Unterfamilie 2021 vom International Committee on Taxonomy of Viruses (ICTV) neu geordnet. Es wurde die neue Familie Salasmaviridae für Bacillus-Phagen mit podovirenartiger Morphologie eingerichtet mit der (modifizierten) Unterfamilie Picovirinae und zwei weiteren neuen Unterfamilien, Northropvirinae und Tatarstanvirinae.[3]
Systematik
[Bearbeiten | Quelltext bearbeiten]Innere Systematik
[Bearbeiten | Quelltext bearbeiten]Die Familie Salasmaviridae setzt sich nach ICTV MSL #36 mit Stand Ende Juni 2021 als Ergebnis dieser Analysen wie folgt zusammen:[1][3]
Familie Salasmaviridae
- Unterfamilie Northropvirinae
- Gattung Claudivirus
- Spezies Claudivirus aurora (Bacillus-Virus Aurora) mit Bacillus phage Aurora
- Spezies Claudivirus baseballfield mit Bacillus phage Baseball_field
- Spezies Claudivirus claudi (Bacillus-Virus Claudi) mit Bacillus phage Claudi
- Spezies Claudivirus Goe4 (Bacillus-Virus Goe4, früher auch englisch Bacillus virus vB_BthP-Goe4, Bacillus thuringiensis virus vB_BthP-Goe4) mit Bacillus phage vB_BthP-Goe4
- Spezies Claudivirus juan (Bacillus-Virus Juan) mit Bacillus phage Juan
- Spezies Claudivirus konjotrouble (Bacillus-Virus KonjoTrouble) mit Bacillus phage KonjoTrouble und Bacillus phage VioletteMad
- Spezies Claudivirus QCM11 (Bacillus-Virus QCM11) mit Bacillus phage QCM11
- Spezies Claudivirus serpounce (Bacillus-Virus SerPounce) mit Bacillus phage SerPounce
- Spezies Claudivirus stitch (Bacillus-Virus Stitch) mit Bacillus phage Stitch, Bacillus phage RadRaab und Bacillus phage StevenHerd11
- Spezies Claudivirus thornton mit Bacillus phage Thornton
- Gattung Hemphillvirus
- Spezies Hemphillvirus bece5 mit Bacillus phage BC-5
- Spezies Hemphillvirus DK1 (Bacillus-Virus DK1) mit Bacillus phage DK1
- Spezies Hemphillvirus DK2 (Bacillus-Virus DK2) mit Bacillus phage DK2
- Spezies Hemphillvirus DK3 (Bacillus-Virus DK3) mit Bacillus phage DK3
- Gattung Klosterneuburgvirus (nicht zu verwechseln mit „Klosneuvirus“, vorgeschlagenes Mitglied der Mimiviridae)
- Spezies Klosterneuburgvirus MGB1 (Bacillus-Virus MGB1) mit Bacillus phage MG-B1 – infiziert Bacillus weihenstephanensis MG1.[4]
- Gattung Layangcvirus
- Spezies Layangcvirus LY3 mit Bacillus phage vB_BceP_LY3
- Gattung Claudivirus
- Unterfamilie Picovirinae[5][6]
- Gattung Bahkauvirus
- Spezies Bahkauvirus chedec mit Bacillus phage Chedec 11
- Gattung Beecentumtrevirus
- Spezies Beecentumtrevirus B103 mit Bacillus phage B103
- Spezies Beecentumtrevirus Goe1 mit Bacillus phage vB_BsuP-Goe1
- Spezies Beecentumtrevirus Nf mit Bacillus phage Nf
- Gattung Salasvirus (veraltet Phi29virus, Phi29likevirus, Phi29-like viruses, Phi29-ähnliche Viren, Φ29-ähnliche Viren)[7]
- Spezies Salasvirus Goe6 mit Bacillus phage vB_BveP-Goe6
- Spezies Salasvirus Gxv1 mit Bacillus phage Gxv1
- Spezies Salasvirus phi29 mit Bacillus phage phi29 alias Bakteriophage Φ29
- Spezies Salasvirus PZA mit Bacillus phage PZA
- Spezies „Bacillus phage BS32“ („Bacillus-Phage BS32“, Vorschlag)[8][9][10]
- Spezies „Bacillus phage M2“ („Bacillus-Phage M2“, Vorschlag)[11][9][10]
- Spezies „Bacillus phage M2Y“ („Bacillus-Phage M2Y“, Vorschlag)[12][9][10]
- Gattung Bahkauvirus
- Unterfamilie Tatarstanvirinae
- Gattung Gaunavirus
- Spezies Gaunavirus GA1 mit Bacillus phage GA1
- Spezies Gaunavirus SRT01hs mit Bacillus phage SRT01hs
- Spezies Gaunavirus syybuna mit Bacillus phage vB_BaeroP_SYYB1
- Gattung Karezivirus
- Spezies Karezivirus karezi mit Bacillus phage Karezi
- Gattung Gaunavirus
- Unterfamilie nicht zugewiesen
- Gattung Bundooravirus
- Spezies Bundooravirus PumA1 (Bacillus-Virus PumA1) mit Bacillus phage vB_Bpu_PumA1
- Spezies Bundooravirus PumA2 (Bacillus-Virus PumA2) mit Bacillus phage vB_Bpu_PumA2
- Spezies Bundooravirus whyphy mit Bacillus phage WhyPhy
- Gattung Harambevirus
- Spezies Harambevirus beachbum (Bacillus-Virus BeachBum) mit Bacillus phage BeachBum
- Spezies Harambevirus harambe (Bacillus-Virus Harambe) mit Bacillus phage Harambe
- Gattung Huangshavirus
- Spezies Huangshavirus dlcuna mit Bacillus phage Dlc1
- Gattung Mingyongvirus
- Spezies Mingyongvirus VMY22 (Bacillus-Virus VMY22) mit Bacillus phage VMY22
- Gattung Bundooravirus
Anmerkung zu den Veränderungen in der Unterfamilie Picovirinae:
- Aus dieser Unterfamilie sind die Gattungen Delislevirus (mit Spezies Delislevirus P1, informell Mycoplasma-Virus P1) und Dybvigvirus (mit Spezies Dybvigvirus Av1, informell Actinomyces-Virus Av1[13]) ohne nähere Zuordnung in der Klasse Caudoviricetes verblieben,
- die Gattung Negarvirus (mit Spezies Negarvirus WP2, informell Lactococcus-Virus WP2) wurde in die Familie Rountreeviridae verschoben.
- Die Spezies Kurthia virus 6 (Kurthia-Virus 6) der Gattung Salasvirus wurde gestrichen, da ohne Beleg.
Die Gattung Cepunavirus wurde inzwischen in die neue Familie Madridviridae verschoben:
- Gattung Cepunavirus (veraltet Cp1virus)
- Spezies Cepunavirus Cp1 (Streptococcus-Virus Cp1, ehem. Typus) mit Streptococcus phage Cp1 und Streptococcus phage Cp9 (ursprünglich Complutense-Phage 1 und 9)[14][15][16]
- Spezies Cepunavirus Cp7 (Streptococcus-Virus Cp7) mit Streptococcus phage Cp-7 (ursprünglich Complutense-Phage 7)[17]
- Spezies „Streptococcus phage Cp-5“ („Streptococcus-Phage Cp-5“, Vorschlag), mit „Complutense-Phage 5“[18]
- Gattung Cepunavirus (veraltet Cp1virus)
Äußere Systematik
[Bearbeiten | Quelltext bearbeiten]Als im weiteren Sinn Phi29-ähnliche Viren stehen die Rountreeviridae vermutlich den Salasmaviridae innerhalb der gemeinsamen Klasse Caudoviricetes nahe.
Beschreibung
[Bearbeiten | Quelltext bearbeiten]

- Die Gattung Salasvirus besitzt ein analog den Adenoviridae und Tectiviridae an das Genom gebundenes terminales Protein, das dem Start der DNA-Replikation (Protein-priming) dient. Die Φ29-ähnlichen Viren kodieren für eine DNA-Polymerase Typ B, z. B. die Φ29-DNA-Polymerase, die der Polymerase II aus Escherichia coli homolog ist.
- Gattung Claudivirus:
- Der Bacillus-Phage Claudi (Spezies Claudivirus claudi) wurde 2014 aus dem Boden durch Anreicherung mit Bacillus thuringiensis subspecies kurstaki (Btk) als Wirtsbakterium von Demetrius Carter (Virginia Commonwealth University, Richmond, VA, USA) isoliert. Auch die meisten anderen Vertreter dieser Gattung wurden in den USA isoliert.[3]
- Der Phage vB_BthP-Goe4 (Spezies Claudivirus Goe4) wurde in Deutschland isoliert. Der Phage vB_BthP-Goe4 hat einen länglichen Kopf (Kapsid) mit einer Höhe von 70,7 nm und einer Breite (Durchmesser) von 50,4 nm. Er hat eine kurze, nicht kontraktile, Schwanzstruktur mit einer Länge von 45,4 nm und einer Breite (Durchmesser) von 6,6 nm (Tobias Schilling et al. 2018).[19][3]
- Alle diese Phagen besitzen invertierte terminale Repeats (en. inverted terminal repeats), die aber teilweise (etwa bei der Spezies Bacillus virus KonjoTrouble) nur unvollkommen invertiert sind.[3]
- Sowohl der Phage MG-B1 (Spezies Klosterneuburgvirus MGB1) als auch sein Wirt (der Bacillus weihenstephanensis-Stamm MG01) wurden aus Waldboden in Österreich isoliert. Seine invertierten kurzen terminalen Repeats mit 22 Nukleotiden (5′-AAATATAGTGGGGTACACTTTT) sind viel größer als bei den zuvor beschriebenen Phagen Phi29, B103 und GA-1, die nur 6, 8 bzw. 7 Nukleotide haben.[20][3]
- Die drei Hemphillvirus-Arten wurden vom Guangdong Institute of Microbiology (China) unter Verwendung von Bacillus cereus als Wirtsbakterium isoliert.[3]
- Die beiden Harambevirus-Arten wurden aus dem Erdboden isoliert, wobei Bacillus thuringiensis subsp. kurstaki (Btk), Stamm ATCC 33679 als Wirtsbakterium verwendet wurde.[3]
- Die einzige Karezivirus-Art (Karezivirus karezi) wurde in den USA aus dem Erdboden isoliert, wobei ebenfalls Bacillus thuringiensis subsp. kurstaki (Btk) als Wirtsbakterium verwendet wurde. An den Genomenden befinden sich 6 Nukleotide invertierte Repeats (AAATTA).[3]
- Der Wirt für die Spezies Bacillus-Phage SRT01hs (Spezies Gaunavirus SRT01hs) ist Bacillus safensis, der für Spezies Bacillus-Phage GA1 (Spezies Gaunavirus GA1) ist Bacillus sp. G1R.[3]
Etymologie
[Bearbeiten | Quelltext bearbeiten]Familie Salasmaviridae: Dieses Taxon ist zu Ehren von Margarita Salas Falgueras (1938–2019), Marquesa de Canero, benannt. Sie war eine spanische Wissenschaftlerin und Forscherin sowie Autorin auf den Gebieten der Biochemie und Molekulargenetik. Ihre Entdeckung der bakteriellen φ29-DNA-Polymerase wurde vom spanischen Nationalen Forschungsrat als das höchstdotierte Patent Spaniens anerkannt. Sie war die erste Wissenschaftlerin, die in die Real Academia Española (en. Royal Spanish Academy) gewählt wurde. Kurz vor ihrem Tod wurde sie mit dem Europäischen Erfinderpreis (European Inventor Award) 2019 ausgezeichnet.
- Unterfamilie Northropvirinae: Diese ist benannt zu Ehren von John H. Northrop (1891–1987), amerikanischer Biochemiker und Nobelpreisträger (Chemie) im Jahr 1946. Er war Professor für Bakteriologie und medizinische Physik und dann Emeritus an der University of California, Berkeley. Northrop arbeitete in den frühen 1950er Jahren an Bacillus-Phagen.
- Gattung Claudivirus: Dessen Name leitet sich direkt vom Namen des ersten Isolats dieser Gattung, dem Bacillus-Phagen Claudi, ab.
- Gattung Hemphillvirus: Diese ist benannt nach Dr. H. Ernest Hemphill (geb. 1940), Professor Emeritus der Syracuse University (NY, USA), der schon früh in seiner Laufbahn an Bacillus-Phagen geforscht hat.
- Klosterneuburgvirus: Diese Gattung ist benannt nach dem Standort des Institute of Science and Technology Austria (ISTA) in Klosterneuburg bei Wien, Österreich, wo der Bacillus-Phage MG-B1 isoliert wurde.
- Unterfamilie Picovirinae: So benannt wegen der geringen Größe des Genoms.
- Gattung Beecentumtrevirus: Der Name dieser Gattung leitet sich vom Namen des Referenzstammes, Bacillus phage B103, ab.
- Gattung Salasvirus: Gleich Namensherkunft wie bei der Familie Salasmaviridae.
- Unterfamilie Tatarstanvirinae: Dieses Taxon ist nach Tatarstan (Republik der Russischen Föderation) benannt, wo das Bacillus-Virus SRT01hs erstmals isoliert wurde.
- Gattung Gaunavirus: Der Name dieser Gattung leitet sich von dem des Referenzstammes, Bacillus-Phage GA-1, ab.
- Gattung Karezivirus: Der Name dieser Gattung leitet sich ebenfalls vom Namen des von dem des Referenzstammes, Bacillus-Phage Karezi, ab.
- Gattungen ohne zugewiesene Unterfamilie:
- Gattung Bundooravirus: Diese Gattung ist nach dem Vorort Bundoora von Melbourne, Queensland, Australien, benannt, wo in der Physiologie, Anatomie und Mikrobiologie der La Trobe University der Bacillus-Phage vB_Bpu_PumA1 isoliert wurde.
- Gattung Harambevirus: Diese Gattung ist wieder nach dem Referenzstamm, Bacillus-Phage Harambe, benannt. Der Name Harambe kommt von Harambee, einer ostafrikanische Tradition von gemeinschaftlichen Selbsthilfeveranstaltungen und ist auch Namensgeber eines Songs von Rita Marley (1988, Harambe — Working Together for Freedom), sowie des Gorillas Harambe.
- Gattung Mingyongvirus: Diese Gattung ist nach dem Mingyong-Gletscher (en. Mingyong Glacier) in Yunnan, China, benannt, aus dem der Bacillus-Phage VMY22 isoliert wurde.
- Benennungen der verschobenen Gattungen:
- * Gattung Delislevirus: Benannt nach Allan L. Delisle, Department of Biomedical Sciences, School of Dentistry: Forschungen an Actinobacterien und deren Phagen,[13][21] sowie Streptokokken und deren Phagen.[22][23]
- * Gattung Dybvigvirus: Benannt nach Kevin F. Dybvig, Department of Genetics, University of Alabama in Birmingham, Alabama, USA: Forschung u. a. an Mykoplasmen und deren Phagen.
Wirte
[Bearbeiten | Quelltext bearbeiten]Die Wirte der Salasmaviridae sind Bakterien der Gattung Bacillus.
Die inzwischen in die neue Familie Madridvirus verschobene Gattung Cepunavirus parasitiert dagegen die Bakteriengattung Streptococcus (siehe Namensgebung der Spezies).
Literatur
[Bearbeiten | Quelltext bearbeiten]- John H. Northrop: Embryo Project Encyclopedia, Marine Biological Laboratory Archives, 1934, ISSN 1940-5030
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ a b c ICTV: ICTV Master Species List 2020.v1, New MSL including all taxa updates since the 2019 release, March 2021 (MSL #36)
- ↑ ICTV: ICTV Master Species List 2021.v2, New MSL including some corrections.
- ↑ a b c d e f g h i j k Evelien M. Adriaenssens, I. Tolstoy, Liliana Cristina Moraru, A. M. Kropinski, Jakub Barylski: 2020.143B.R.Salasmaviridae.zip (docx, xlsx), Vorschlag an das ICTV: Create one new family (Salasmaviridae) including three subfamilies and ten genera (Caudovirales) and create two new genera (Caudovirales: Podoviridae), Juni 2020
- ↑ NCBI Nucleotide: Bacillus phage MG-B1, …, ACCESSION NC_021336.
- ↑ Luis Fernando Camarillo Guerrero: Integrative Analysis of the Human Gut Phageome Using a Metagenomics Approach. Doktorarbeit, Gonville & Caius College, University of Cambridge, August 2020, doi:10.17863/CAM.63973 (englisch).
- ↑ SIB: Picovirinae. Auf: ViralZone.
- ↑ SIB: Salasvirus. Auf: ViralZone.
- ↑ NCBI: Bacillus phage BS32 (species)
- ↑ a b c S. Mc Grath: Bacteriophage: Genetics and Molecular Biology. Hrsg.: D. van Sinderen. 1. Auflage. Caister Academic Press, 2007, ISBN 978-1-904455-14-1.
- ↑ a b c Ana Camacho, Fernando Jimenez, Javier Torre, Jose L. Carrascosa, Rafael P. Mellado, Eladio Vinuela, Margarita Salas, Cesar Vasquez: Assembly of Bacillus subtilis Phage Phi29. 1. Mutants in the Cistrons Coding for the Structural Proteins. In: European Journal of Biochemistry. Band 73, Nr. 1, Februar 1977, S. 39–55, doi:10.1111/j.1432-1033.1977.tb11290.x.
- ↑ NCBI: Bacillus phage M2 (species)
- ↑ NCBI: Bacillus phage M2Y (species)
- ↑ a b NCBI: Actinomyces virus Av1 (species).
- ↑ NCBI: Streptococcus virus Cp1 (species)
- ↑ C. Ronda, R. López, E. García: Isolation and characterization of a new bacteriophage, Cp-1, infecting Streptococcus pneumoniae. In: J. Virol. Band 40, Nr. 2, 1981, S. 551–559, doi:10.1128/JVI.40.2.551-559.1981, PMID 6275103, PMC 256658 (freier Volltext).
- ↑ Rubens López: Streptococcus pneumoniae and its bacteriophages: one long argument. In: International Microbiology, Band 7, Nr. 3, S. 163–171, Oktober 2004; doi:10.1093/clinids/3.2.212, ResearchGate:8223977 (englisch))
- ↑ NCBI: Streptococcus virus Cp7 (species)
- ↑ NCBI: Streptococcus phage Cp-5 (species)
- ↑ Tobias Schilling, Michael Hoppert, Robert Hertel: Genomic Analysis of the Recent Viral Isolate vB_BthP-Goe4 Reveals Increased Diversity of φ29-Like Phages. In: MDPI: Viruses, Band 10, Nr. 11, Special Issue Viruses of Microbes V: Biodiversity and Future Applications, 13. November 2018, pii: E624; doi:10.3390/v10110624, PMID 30428528 (englisch).
- ↑ R. A. Redondo, A. Kupczok, G. Stift, J. P. Bollback: Complete Genome Sequence of the Novel Phage MG-B1 Infecting Bacillus weihenstephanensis. In: Genome Announc. Band 1, Nr. 3, 2013, pii: e00216-13, doi:10.1128/genomeA.00216-13, PMID 23766400 (englisch).
- ↑ Allan L. Delisle, Gerard J. Barcak, Ming Guo: Isolation and Expression of the Lysis Genes of Actinomyces naeslundii Phage Av-1. In: ASM Applied and Environmental Microbiology., Band 72, Nr. 2, 17. Dezember 2020, doi:10.1128/AEM.72.2.1110-1117.2006.
- ↑ NCBI: Streptococcus phage M102AD (species, unclassified Siphoviridae).
- ↑ Allan L. Delisle, Ming Guo, Natalia I. Chalmers, Gerard J. Barcak, Geneviève M. Rousseau, Sylvain Moineau: Biology and Genome Sequence of Streptococcus mutans Phage M102AD. In: ASM Applied and Environmental Microbiology. Band 78, Nr. 7, 9. März 2012, doi:10.1128/AEM.07726-11.