Acinetobacter baumannii
| Acinetobacter baumannii | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
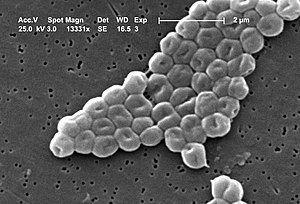
elektronenmikroskopische Aufnahme von Acinetobacter baumannii | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Acinetobacter baumannii | ||||||||||||
| Bouvet & Grimont 1986[1] |
Acinetobacter baumannii ist ein gramnegatives, aerobes Bakterium. Das Genom des Stammes Acinetobacter baumannii AF-401 wurde im Jahr 2017 vollständig sequenziert. Wie andere Arten der Gattung Acinetobacter gehört das häufig multiresistente (gramnegative) Bakterium zu den Verursachern von nosokomialen Infektionen. In vielen Ländern zählt A. baumannii zu den wichtigsten Krankenhauskeimen überhaupt und kommt vor allem auf Intensivstationen vor. Die amerikanische Gesellschaft für Infektionskrankheiten zählt Acinetobacter baumannii aufgrund seiner Fähigkeit, besonders ausgeprägte Antibiotikaresistenzen auszubilden, zu den sogenannten ESKAPE Pathogenen, gegen die therapeutische Möglichkeiten knapp werden.[2]
Merkmale[Bearbeiten | Quelltext bearbeiten]
Erscheinungsbild[Bearbeiten | Quelltext bearbeiten]
Bakterien der Art Acinetobacter baumannii sind stäbchenförmig und kommen in der stationären Phase auch kokkenförmig vor.[3] Traditionell wird A. bamannii als unbeweglich beschrieben, es gibt jedoch Nachweise für Oberflächenbeweglichkeit.[4] A. baumannii gehört zu den gramnegativen Bakterien, ist in der Gram-Färbung jedoch häufig uneindeutig (gramvariabel) und zeigt im lichtmikroskopischen Bild kokkoide Stäbchen.[2] Es werden keine Überdauerungsformen wie Endosporen gebildet.[3] Auf Casein-Soja-Pepton-Agar wachsen die Bakterienkolonien kreisrund, konvex, glatt und leicht opak. Sie haben einen Durchmesser von 1,5 bis 2,0 mm nach 24 Stunden Inkubation und 3,0 bis 4,0 mm nach 48 Stunden Bebrütung bei 30 °C.[5]
Wachstum und Stoffwechsel[Bearbeiten | Quelltext bearbeiten]
Acinetobacter-Arten sind im Oxidase-Test negativ, das unterscheidet sie von Pseudomonas aus der gleichen Ordnung Pseudomonadales.[3] Sie zählen zu den nicht-fermentierenden Bakterien (sogenannte Nonfermenter).[2]
Das Wachstum erfolgt zwischen 15 und 44 °C. Die meisten Stämme können aus Glucose Säure bilden. Auf Blutagar erfolgt keine Hämolyse, Gelatine kann nicht hydrolysiert werden. Auf Simmons Citrat-Agar können prototrophe Stämme Citrat verwerten, auxotrophe nicht, es sei denn, sie werden zusätzlich mit Wachstumsfaktoren im Nährmedium versorgt.[5] Weitere Stoffwechseleigenschaften sind im Abschnitt Nachweise aufgeführt.
Genetik[Bearbeiten | Quelltext bearbeiten]
Das Genom des Stammes Acinetobacter baumannii AF-401 wurde im Jahr 2017 vollständig sequenziert. Es handelt sich um ein Projekt zur Genom-Sequenzierung Carbapenem-resister A. baumannii Stämme. Mittlerweile ist das Genom von mehr als 20 Stämmen erforscht.[6] Das Genom weist eine Größe von 3982 Kilobasenpaaren (kb) auf, es sind 3953 Proteine annotiert. Die Ergebnisse der Sequenzierungen zeigen einen GC-Gehalt (den Anteil der Nukleinbasen Guanin und Cytosin) in der Bakterien-DNA von 39 Molprozent (Median).[7] Dies liegt etwas unterhalb des in der Erstbeschreibung angegebenen GC-Gehaltes von 40–43 Molprozent.[5] Zusätzlich zum Bakterienchromosom liegt ein Plasmid vor, auf dem sich mehrere Antibiotika-Resistenzgene befinden. Das Plasmid und damit auch die Antibiotikaresistenz kann mittels horizontalen Gentransfer zwischen Bakterienarten ausgetauscht werden.[2]
Nachweise[Bearbeiten | Quelltext bearbeiten]
Das Kriterium „Wachstum bei 42 °C“ kann zur Unterscheidung von Acinetobacter calcoaceticus verwendet werden, der bei dieser Temperatur nicht wächst.[5]
Biochemische Nachweise[Bearbeiten | Quelltext bearbeiten]
Biochemische Merkmale, wie beispielsweise die vorhandenen Enzyme und die daraus resultierenden Stoffwechseleigenschaften können in einer Bunten Reihe zur Identifizierung von Acinetobacter baumannii bzw. Unterscheidung zwischen den Acinetobacter-Arten oder Unterscheidung dieser von anderen gramnegativen Bakterien genutzt werden. Es erfolgt keine Bildung von Schwefelwasserstoff (H2S), auch die Indolbildung verläuft negativ. Das Enzym β-Galactosidase zum Abbau von Lactose ist ebenfalls nicht vorhanden. Zu den Substraten, die A. baumannii verwerten kann, gehören beispielsweise D- und L-Lactat, Glutarat, L-Aspartat, L-Tyrosin, 4-Aminobutyrat, Ethanol und 2,3-Butandiol.[5]
Für die biochemische Identifizierung können miniaturisierte Testsysteme verwendet werden, geeignet sind Systeme für gramnegative Stäbchen (Enterobacteriaceae) oder gramnegative Stäbchen (Nicht-Enterobacteriaceae). Die Unterscheidung der beiden Arten A. baumannii und A. calcoaceticus ist mit zusätzlichen Tests möglich.[8] Die Speziesdiagnostik mittels Biochemie ist jedoch schwierig.[2]
Weitere Nachweise[Bearbeiten | Quelltext bearbeiten]
Mittels MALDI-TOF MS ist eine sichere Abgrenzung zwischen verschiedenen Acinetobacter spp. möglich.[9] Durch partielle Gensequenzierung bei Verwendung des rpoB-Locus oder mittels PCR durch das blaOXA-51-like Gens kann ebenfalls die Spezies Acinetobacter baumannii sicher ermittelt werden.[2]
Vorkommen[Bearbeiten | Quelltext bearbeiten]
A. baumannii ist ein Umweltkeim und kommt im Boden und Wasser vor, zahlreiche Isolate stammen allerdings auch aus klinischen Proben von menschlichen Patienten.[5] Acinetobacter-Stämme mit der Carbapenemase NDM-1 wurden mittlerweile auch aus Klinikabwässern und Geflügelfleisch isoliert.[2]
Systematik und Taxonomie[Bearbeiten | Quelltext bearbeiten]
Das Epitheton wurde zu Ehren von Paul und Linda Baumann (US-amerikanische Bakteriologen) gewählt.[5] Der Typusstamm von Acinetobacter baumannii wurde in den Sammlungen von Mikroorganismen in den USA (als ATCC 19606), Deutschland (bei der DSMZ als DSM 30007) und weiteren Ländern hinterlegt.[1]
Die beiden Spezies A. baumannii und A. calcoaceticus sind nah miteinander verwandt, dies zeigen phänotypische Testergebnisse und genetische Untersuchungen mit Hilfe der DNA-DNA-Hybridisierung.[5] In der medizinischen Diagnostik werden sie daher als A.-calcoaceticus-A.baumannii-Komplex (ACB-Komplex) bezeichnet. Auch die Spezies Acinetobacter nosocomialis (früher Genom-Spezies 13TU) und Acinetobacter pittii (früher Genom-Spezies 3) werden dazu gezählt.[2]
Medizinische Bedeutung[Bearbeiten | Quelltext bearbeiten]
In vielen Länder zählt A. baumannii zu den wichtigsten Krankenhauskeimen überhaupt und kommt vor allem auf Intensivstationen vor. Die amerikanische Gesellschaft für Infektionskrankheiten zählt Acinetobacter baumannii aufgrund seiner Fähigkeit, besonders ausgeprägte Antibiotikaresistenzen auszubilden, zu den sogenannten ESKAPE Pathogenen, gegen die therapeutische Möglichkeiten knapp werden. Acinetobacter baumannii ist in der Lage, Biofilme zu bilden und auch Trockenheit zu überstehen. Oberflächen um besiedelte Patienten sind daher häufig kontaminiert. Das Übertragungspotenzial übersteigt das klassischer Krankenhauskeime wie MRSA deutlich.[2]
Klinische Erscheinungsformen[Bearbeiten | Quelltext bearbeiten]
Die durch Acinetobacter baumannii verursachten nosokomialen Infektionen zeigen sich in Form von Lungenentzündung (auch durch Beatmung bedingt), katheter-assoziierten Infektionen, Bakteriämie und Sepsis. Auch Wundinfektionen (etwa nach Verbrennungen) und nosokomiale Harnwegsinfektionen sowie Abszesse können auftreten. Selten kommt auch eine Hirnhautentzündung (z. B. nach neurochirurgischen Eingriffen) vor.[10]
Pathogenität[Bearbeiten | Quelltext bearbeiten]
A. baumannii wird durch die Biostoffverordnung in Verbindung mit der TRBA (Technische Regeln für Biologische Arbeitsstoffe) 466 der Risikogruppe 2 zugeordnet.[11]
Antibiotikaresistenz[Bearbeiten | Quelltext bearbeiten]
Acinetobacter baumannii muss nicht, aber kann zu den multiresistenten gramnegativen Stäbchen-Bakterien gehören und wird gemäß der KRINKO-Definition häufig als 3MRGN oder sogar 4MRGN klassifiziert.[2] Tatsächlich ist es im Verlauf der letzten 20 Jahre, analog zur allgemeinen Entwicklung der Resistenzlage, zu einer deutlichen Zunahme der hochresistenten Stämme gekommen. Hierzu hat u. a. die weltweit expandierende individuelle Mobilität beigetragen und möglicherweise existiert im Falle von Acinetobacter eine Assoziation zu bewaffneten Konflikten. Es lassen sich in Kriegsversehrten aus Nordafrika und dem Mittleren Osten zumindest eine Reihe von A. baumannii-Resistenzmechanismen nachweisen, die über das normale Maß hinausgehen[12]. Die meisten A. baumannii-Stämme sind gegenüber Ampicillin sowie Cephalosporinen der ersten und zweiten Generation unempfindlich und oft sind auch Cephalosporine der dritten Generation, Aztreonam und Piperacillin nicht mehr therapeutisch anwendbar. Insbesondere jedoch die Zunahme von Resistenzen gegen Chinolon-Antibiotika und Carbapeneme, die sonst als Reserveantibiotika fungieren, stellen Behandler in der Medizin vor große Probleme. Unter anderem da sich mit zunehmender Resistenz die Fähigkeit zur Biofilmbildung erhöht.[13] Als Behandlungsoptionen verbleiben häufig nur noch Colistin und Tigecyclin[14]. In der Literatur wird bereits vereinzelt das Vorkommen panresistenter Stämme beschrieben[15] und diskutiert, ob dies ein anekdotisches Phänomen bleiben wird. Vielversprechend für zukünftige Therapieversuche wirkt der in 2020 beschriebene β-Laktamase-Inhibitor QPX7728[16]. Der Inhibitor Clavulansäure scheint zumindest in Grenzen eine Eigenaktivität gegen A. baumannii aufzuweisen.[17] Heilversuche mit Cefiderocol wurden beschrieben.[18]
Ausbrüche[Bearbeiten | Quelltext bearbeiten]
Hohen Bekanntheitsgrad erlangte Acinetobacter baumannii (umgangssprachlich auch „Iraqibacter“) durch seinen häufigen Nachweis in infizierten Wunden von US-Soldaten, die aus Einsätzen im Irak und Afghanistan heimkehrten.[19][20]
Im Juli 2008 wurden in der Medizinischen Hochschule Hannover 23 Fälle in der Intensivstation für Schwerbrandverletzte gemeldet. Der A. baumanii-Stamm war nur noch gegenüber einem Antibiotikum sensibel. Durch konsequent verstärkte Hygienemaßnahmen konnte der resistente Erreger aus der Station eliminiert werden.[21]
Anfang 2015 kam es im Universitätsklinikum Schleswig-Holstein Campus Kiel zu einem Ausbruch, bei dem Acinetobacter baumannii bei 31 Patienten nachgewiesen wurde, 12 Patienten verstarben und bei dreien von diesen könnte die Infektion die direkte Todesursache gewesen sein. Grund zur Sorge war die Tatsache, dass es sich um einen multiresistenten 4MRGN-Erreger handelte.[20]
Therapie[Bearbeiten | Quelltext bearbeiten]
Behandelt werden Acinetobacter-baumannii-Infektionen mit Imipenem oder Meropenem und (je nach Antibiogramm mit) Chinolon bzw. Aminoglykosid. Alternativ kommen (bei nachgewiesener Sensibilität) auch Tigecyclin und Amikacin bzw. Ciprofloxacin in Frage. Resistente Stämme können mit Colistin und Meropenem oder Imipenem behandelt werden. Bei einer Carbapenem-Resistenz kommt eine Kombination von Colistin und Amikacin in Betracht.[22]
Im Jahr 2019 wurde mit dem Molekül Halicin ein potentiell wirksames Antibiotikum gegen Acinetobacter baumannii gefunden.[23] Des Weiteren wurde 2018 eine Methode zur Hemmung der Aufnahme von Eisen bei A. baumanni untersucht.[24]
Lebensmittelverderb[Bearbeiten | Quelltext bearbeiten]
Auf Frischfleisch kann Acinetobacter baumannii zu Geruchsabweichungen führen.[25]
Einzelnachweise[Bearbeiten | Quelltext bearbeiten]
- ↑ a b Jean Euzéby, Aidan C. Parte: Genus Acinetobacter. In: List of Prokaryotic names with Standing in Nomenclature (LPSN). Abgerufen am 4. April 2018.
- ↑ a b c d e f g h i j Acinetobacter baumannii – ein Krankenhauskeim mit beunruhigendem Entwicklungspotenzial. In: Robert Koch-Institut (Hrsg.): Epidemiologisches Bulletin 32/2013. 12. August 2013, S. 295–299 (rki.de [PDF; 135 kB; abgerufen am 4. April 2018]).
- ↑ a b c Michael T. Madigan, John M. Martinko, Jack Parker: Brock Mikrobiologie. Deutsche Übersetzung herausgegeben von Werner Goebel, 1. Auflage. Spektrum Akademischer Verlag GmbH, Heidelberg/Berlin 2000, ISBN 3-8274-0566-1, S. 530–531.
- ↑ Katy M. Clemmer, Robert A. Bonomo, Philip N. Rather: Genetic analysis of surface motility in Acinetobacter baumannii. In: Microbiology. Band 157, Nr. 9, 2011, ISSN 1465-2080, S. 2534–2544, doi:10.1099/mic.0.049791-0, PMID 21700662, PMC 3352170 (freier Volltext) – (microbiologyresearch.org [abgerufen am 5. Dezember 2023]).
- ↑ a b c d e f g h Philippe J. M. Bouvet, Patrick A. D. Grimont: Taxonomy of the Genus Acinetobacter with the Recognition of Acinetobacter baumannii sp. nov., Acinetobacter haemolyticus sp. nov., Acinetobacter johnsonii sp. nov., and Acinetobacter junii sp. nov. and Emended Descriptions of Acinetobacter calcoaceticus and Acinetobacter lwoffii. In: International Journal of Systematic and Evolutionary Microbiology. Band 36, Nr. 2, 1986, S. 228–240, doi:10.1099/00207713-36-2-228.
- ↑ Acinetobacter baumannii AF-401. In: Webseite JCI Genomes Online Database (GOLD). Abgerufen am 4. April 2018.
- ↑ Acinetobacter baumannii. In: Webseite Genome des National Center for Biotechnology Information (NCBI). Abgerufen am 4. April 2018.
- ↑ API® ID-Teststreifen. (PDF; 2,1 MB) In: Webseite der bioMérieux Deutschland GmbH. Abgerufen am 4. April 2018.
- ↑ Seri Jeong, Jun Sung Hong, Jung Ok Kim, Keon-Han Kim, Woonhyoung Lee, Il Kwon Bae, Kyungwon Lee, Seok Hoon Jeong: Identification of Acinetobacter Species Using Matrix-Assisted Laser Desorption Ionization-Time of Flight Mass Spectrometry. In: Annals of Laboratory Medicine. Band 36, Nr. 4, 1. Juli 2016, ISSN 2234-3806, S. 325–334, doi:10.3343/alm.2016.36.4.325, PMID 27139605, PMC 4855052 (freier Volltext) – (annlabmed.org [abgerufen am 5. Dezember 2023]).
- ↑ Marianne Abele-Horn (2009), S. 260.
- ↑ TRBA (Technische Regeln für Biologische Arbeitsstoffe) 466: Einstufung von Prokaryonten (Bacteria und Archaea) in Risikogruppen. In: Webseite der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA). 25. August 2015, abgerufen am 29. März 2018 (letzte Änderung vom 31. März 2017).
- ↑ Paul G. Higgins, Ralf Matthias Hagen, Bernd Kreikemeyer, Philipp Warnke, Andreas Podbielski: Molecular Epidemiology of Carbapenem-Resistant Acinetobacter baumannii Isolates from Northern Africa and the Middle East. In: Antibiotics. Band 10, Nr. 3, 11. März 2021, ISSN 2079-6382, S. 291, doi:10.3390/antibiotics10030291, PMID 33799540, PMC 8002098 (freier Volltext) – (mdpi.com [abgerufen am 17. September 2021]).
- ↑ Mona Mohamed Al-Shamiri, Sirui Zhang, Peng Mi, Yuqing Liu, Meng Xun: Phenotypic and genotypic characteristics of Acinetobacter baumannii enrolled in the relationship among antibiotic resistance, biofilm formation and motility. In: Microbial Pathogenesis. Band 155, Juni 2021, S. 104922, doi:10.1016/j.micpath.2021.104922 (elsevier.com [abgerufen am 17. September 2021]).
- ↑ Friedrich Burkhardt: Mikrobiologische Diagnostik; Bakteriologie - Mykologie - Virologie - Parasitologie. Hrsg.: Birgid Neumeister, Heinrich K. Geiss, Rüdiger Braun, Peter Kimmig. 2. Auflage. Georg Thieme Verlag, Stuttgart / New York 2009, ISBN 978-3-13-743602-7, S. 495–496.
- ↑ Stamatis Karakonstantis, Evangelos I. Kritsotakis, Achilleas Gikas: Is pandrug-resistance in A. baumannii a transient phenotype? Epidemiological clues from a 4-year cohort study at a tertiary referral hospital in Greece. In: Journal of Chemotherapy. Band 33, Nr. 5, 4. Juli 2021, ISSN 1120-009X, S. 354–357, doi:10.1080/1120009X.2020.1839689.
- ↑ Kirk Nelson, Debora Rubio-Aparicio, Ruslan Tsivkovski, Dongxu Sun, Maxim Totrov: In Vitro Activity of the Ultra-Broad-Spectrum Beta-Lactamase Inhibitor QPX7728 in Combination with Meropenem against Clinical Isolates of Carbapenem-Resistant Acinetobacter baumannii. In: Antimicrobial Agents and Chemotherapy. Band 64, Nr. 11, 20. Oktober 2020, ISSN 0066-4804, doi:10.1128/AAC.01406-20, PMID 32868334, PMC 7577151 (freier Volltext).
- ↑ Grit Ackermann: Antibiotika und Antimykotika Substanzen, Krankheitsbilder, erregerspezifische Therapie. 4., überarb. und erw. Auflage. Stuttgart 2014, ISBN 978-3-8047-2940-7, S. 49.
- ↑ Alessandra Oliva, Giancarlo Ceccarelli, Massimiliano De Angelis, Federica Sacco, Maria Claudia Miele: Cefiderocol for compassionate use in the treatment of complicated infections caused by extensively and pan-resistant Acinetobacter baumannii. In: Journal of Global Antimicrobial Resistance. Band 23, Dezember 2020, S. 292–296, doi:10.1016/j.jgar.2020.09.019 (elsevier.com [abgerufen am 17. September 2021]).
- ↑ Acinetobacter baumannii. UC San Diego School of Medicine, abgerufen am 5. Dezember 2023 (englisch).
- ↑ a b Christine Starostzik: Problemkeim fordert Klinikpersonal heraus. In: Ärzte Zeitung. 2. Februar 2015, abgerufen am 4. April 2018.
- ↑ Nicola Siegmund-Schultze: Acinetobacter baumanii: Bei diesem Keim in der Klinik ist Feuer unterm Dach. In: Ärzte Zeitung. 29. Juni 2010, abgerufen am 4. April 2018.
- ↑ Marianne Abele-Horn: Antimikrobielle Therapie. Entscheidungshilfen zur Behandlung und Prophylaxe von Infektionskrankheiten. Unter Mitarbeit von Werner Heinz, Hartwig Klinker, Johann Schurz und August Stich, 2., überarbeitete und erweiterte Auflage. Peter Wiehl, Marburg 2009, ISBN 978-3-927219-14-4, S. 260.
- ↑ Julia Merlot, DER SPIEGEL: Kampf gegen Resistenzen: Künstliche Intelligenz entdeckt vielversprechendes Antibiotikum - DER SPIEGEL - Wissenschaft. Abgerufen am 22. Februar 2020.
- ↑ Vishvanath Tiwari, Moganty R. Rajeswari, Monalisa Tiwari: Proteomic analysis of iron-regulated membrane proteins identify FhuE receptor as a target to inhibit siderophore-mediated iron acquisition in Acinetobacter baumannii. In: International Journal of Biological Macromolecules. Band 125, März 2019, S. 1156–1167, doi:10.1016/j.ijbiomac.2018.12.173 (elsevier.com [abgerufen am 16. Dezember 2023]).
- ↑ Arthur Hinton Jr., J. A. Cason, Kimberly D. Ingram: Tracking spoilage bacteria in commercial poultry processing and refrigerated storage of poultry carcasses. In: International Journal of Food Microbiology. Band 91, Nr. 2, März 2004, S. 155–165, doi:10.1016/S0168-1605(03)00377-5.