Nichtproteinogene Aminosäuren
Nichtproteinogene Aminosäuren sind Aminosäuren, die nicht in Proteinen während der Translation eingebaut werden.[1] Sie wirken im Aminosäuren-Stoffwechsel und der Proteinbiosynthese daher oftmals als Aminosäureantagonisten.[2] Von den Aminocarbonsäuren sind die Aminoheterooxosäuren zu unterscheiden. Weitere Unterscheidungskriterien sind die Ständigkeit (Isomerie) der Amino- relativ zur Säuregruppe sowie ggf. die Konfiguration dieser funktionellen Gruppen, insbesondere in α-Aminosäuren.
Eigenschaften[Bearbeiten | Quelltext bearbeiten]


Es sind bisher über 400 nichtproteinogene Aminosäuren bekannt, die in Organismen vorkommen.[3] Diese natürlich gebildeten nichtproteinogenen Aminosäuren erfüllen verschiedene Funktionen. Manche entstehen als posttranslationale Modifikation in Proteinen (z. B. Phosphotyrosin, Hydroxyprolin, Formylmethionin, Cystin, Lanthionin, Djenkolsäure, 2,6-Diaminopimelinsäure). Andere sind Metaboliten im Stoffwechsel (z. B. Ornithin, Citrullin, Argininosuccinat, Sarcosin, Homoserin, Homocystein, L-DOPA, 5-Hydroxytryptophan). Weitere dienen als Hormone (L-Thyroxin) und Neurotransmitter (z. B. GABA, D-Serin) oder auch als Toxine (β-Methylamino-Alanin, Ibotensäure, L-Azetidin-2-carbonsäure, Quisqualat, Canavanin).[4]
Über 250 unterschiedliche nichtproteinogene Aminosäuren wurden als Bausteine der Oligopeptide oder kurzen Polypeptide gefunden, die durch nichtribosomale Peptidsynthetasen verschiedener Arten von Archaeen, Bakterien, Pilzen und Nacktkiemern aufgebaut werden.[5]
Künstlich erzeugte Aminosäuren sind z. B. die Inhibitoren Isoserin, 2-Amino-5-phosphonovaleriansäure und der Synthesegrundstoff für β-Lactam-Antibiotika D-Phenylglycin. Im Miller-Urey-Experiment wurden unter anderem die Aminosäuren Norvalin und Norleucin künstlich erzeugt.
Struktur[Bearbeiten | Quelltext bearbeiten]
Im Gegensatz zu den proteinogenen Aminosäuren sind nicht alle nichtproteinogenen Aminosäuren L-α-Aminosäuren, z. B. die Gruppe der D-Aminosäuren oder die nicht-α-Aminosäuren wie β-Alanin, GABA, δ-Aminolävulinsäure oder 4-Aminobenzoesäure. Weitere nichtproteinogene Aminosäuren besitzen ein verändertes Peptid-Rückgrat wie 3-Aminoisobuttersäure oder Dehydroalanin. Es existieren ebenso nichtproteinogene Aminosäuren mit zwei Chiralitätszentren wie z. B. Cystathionin, Lanthionin, Djenkolsäure oder 2,6-Diaminopimelinsäure. Schwefel-enthaltende nichtproteinogene Aminosäuren sind z. B. Homocystein, Ethionin und Felinin. Selen-enthaltende nichtproteinogene Aminosäuren sind Methylselenocystein, Selenoethionin und Selenomethionin. Manche Arten wie Aspergillus fumigatus, Aspergillus terreus und Penicillium chrysogenum sind in Abwesenheit von Schwefel in der Lage, Tellurocystein und Telluromethionin zu produzieren und in Proteine einzubauen,[6] während diese Aminosäuren für die meisten Arten nichtproteinogen sind.
Beispiele[Bearbeiten | Quelltext bearbeiten]
| Aminosäure | Funktion | Struktur | Strukturformel |
|---|---|---|---|
| Taurin | 
| ||
| L-Thyroxin (T4) | Hormon der Schilddrüse | L-α-Aminosäure | 
|
| 2,6-Diaminopimelinsäure (DAP) | Bestandteil in bakteriellen Zellwänden | α-Aminosäure mit 2 Stereozentren | 
|
| Azetidin-2-carbonsäure | Toxin der Maiglöckchen | L-α-Aminosäure, sekundäres Amin in einem Ring | 
|
| Sarkosin | Stoffwechselzwischenprodukt im Aminosäurestoffwechsel | N-Methyl-α-aminosäure | 
|
| Homoserin | Abbauprodukt von Methionin | L-α-Aminosäure | 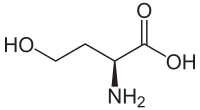
|
| Lanthionin | in Strukturproteinen | α-Aminosäure mit 2 Stereozentren | 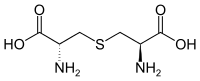
|
| Djenkolsäure | Fraßgift von Archidendron jiringa (Jengkol), einer Hülsenfrucht | α-Aminosäure mit 2 Stereozentren | 
|
| Cystathionin | Intermediat der Biosynthese von Cystein | α-Aminosäure mit 2 Stereozentren | 
|
| L-Homocystein | Abbauprodukt von Methionin | L-α-Aminosäure | 
|
| Ethionin | Methionin-Antagonist | L-α-Aminosäure | 
|
| 5-Aminolävulinsäure (5-ALA) | Vorstufe des Häms | δ-Aminosäure | 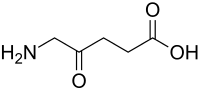
|
| p-Aminobenzoesäure (PABA) | bakterielle Folsäuresynthese | aromatisches Rückgrat | 
|
| Dehydroalanin (DHA) | Abbauprodukt von Cystein | Doppelbindung am α-C-Atom | 
|
| γ-Aminobuttersäure (GABA) | inhibitorischer Neurotransmitter | γ-Aminosäure | 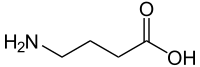
|
| 3-Aminoisobuttersäure | ist in den Fettstoffwechsel involviert | β-Aminosäure | 
|
| L-Ornithin | Stoffwechselzwischenprodukt im Harnstoffzyklus | L-α-Aminosäure | 
|
| L-Citrullin | Stoffwechselzwischenprodukt im Harnstoffzyklus | L-α-Aminosäure | 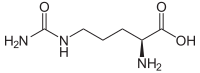
|
| Argininosuccinat | Stoffwechselzwischenprodukt im Harnstoffzyklus | L-α-Aminosäure | 
|
| L-3,4-Dihydroxyphenylalanin (L-DOPA) | Stoffwechselzwischenprodukt bei der Synthese von Katecholaminen | L-α-Aminosäure | 
|
| L-5-Hydroxytryptophan (5-HTP) | Stoffwechselzwischenprodukt bei der Serotoninsynthese | L-α-Aminosäure | 
|
| β-Alanin | Baustein von Coenzym A | β-Aminosäure | 
|
| β-N-Methylamino-Alanin | Neurotoxin in Cyanobakterien | L-α-Aminosäure | 
|
| Ibotensäure | Pilzgift | L-α-Aminosäure | 
|
| D-Valin | Bestandteil des Antibiotikums Valinomycin | D-Aminosäure | 
|
| D-Alanin | Bestandteil in bakteriellen Zellwänden | D-Aminosäure | 
|
| D-Glutaminsäure | Bestandteil in bakteriellen Zellwänden | D-Aminosäure | 
|
| Hypoglycin | Toxin der Akee-Pflanze | L-α-Aminosäure | 
|
| L-4-Hydroxyprolin | Stabilisiert die Struktur des Kollagens | L-α-Aminosäure, sekundäres Amin in einem Ring | 
|
| Pipecolinsäure | Abbauprodukt von Lysin | L-α-Aminosäure, sekundäres Amin in einem Ring |  |
| Glufosinat | Herbizid, enthält Phosphinsäure-Gruppe | L-α-Aminosäure | 
|
Anwendungen[Bearbeiten | Quelltext bearbeiten]
Nichtproteinogene Aminosäuren können mit nichtribosomalen Peptidsynthetasen,[7] durch Proteinligation,[8] durch eine Peptidsynthese oder durch genetische Reprogrammierung in Proteine eingebaut werden.[9][10] Mit Peptiden, die nichtproteinogene Aminosäuren enthalten, können die Substratspezifitäten von Peptidasen untersucht und Proteaseinhibitoren entwickelt werden.[11] β-Peptide sind aus β-Aminosäuren aufgebaut.
Einzelnachweise[Bearbeiten | Quelltext bearbeiten]
- ↑ Jan Koolman, Klaus Heinrich Roehm: Color Atlas of Biochemistry. 3. Ausgabe, Thieme 2012. ISBN 978-3-13-169693-9.
- ↑ Gerhard Habermehl, Peter E. Hammann: Naturstoffchemie: Eine Einführung. Springer-Verlag, 2013, ISBN 978-3-662-08929-3 (Google Books).
- ↑ Peter Nuhn: Naturstoffchemie, S. Hirzel Wissenschaftliche Verlagsgesellschaft Stuttgart 1990, ISBN 3-7776-0473-9. S. 70.
- ↑ K. Dasuri, P. J. Ebenezer, R. M. Uranga, E. Gavilán, L. Zhang, S. O. Fernandez-Kim, A. J. Bruce-Keller, J. N. Keller: Amino acid analog toxicity in primary rat neuronal and astrocyte cultures: implications for protein misfolding and TDP-43 regulation. In: Journal of neuroscience research. Band 89, Nummer 9, September 2011, S. 1471–1477, doi:10.1002/jnr.22677, PMID 21608013, PMC 3175609 (freier Volltext).
- ↑ siehe Einträge Monomere in der Datenbank NORINE.
- ↑ S. E. Ramadan, A. A. Razak, A. M. Ragab, M. el-Meleigy: Incorporation of tellurium into amino acids and proteins in a tellurium-tolerant fungi. In: Biological trace element research. Band 20, Nummer 3, Juni 1989, S. 225–232, doi:10.1007/BF02917437, PMID 2484755.
- ↑ G. H. Hur, C. R. Vickery, M. D. Burkart: Explorations of catalytic domains in non-ribosomal peptide synthetase enzymology. In: Natural product reports. Band 29, Nummer 10, Oktober 2012, S. 1074–1098, doi:10.1039/c2np20025b, PMID 22802156.
- ↑ U. Arnold: Incorporation of non-natural modules into proteins: structural features beyond the genetic code. In: Biotechnology letters. Band 31, Nummer 8, August 2009, S. 1129–1139, doi:10.1007/s10529-009-0002-9, PMID 19404746.
- ↑ A. Ohta, Y. Yamagishi, H. Suga: Synthesis of biopolymers using genetic code reprogramming. In: Current opinion in chemical biology. Band 12, Nummer 2, April 2008, S. 159–167, doi:10.1016/j.cbpa.2007.12.009, PMID 18249198.
- ↑ Y. Lu, S. Freeland: On the evolution of the standard amino-acid alphabet. In: Genome biology. Band 7, Nummer 1, 2006, S. 102, doi:10.1186/gb-2006-7-1-102, PMID 16515719, PMC 1431706 (freier Volltext).
- ↑ P. Kasperkiewicz, A. D. Gajda, M. Drąg: Current and prospective applications of non-proteinogenic amino acids in profiling of proteases substrate specificity. In: Biological chemistry. Band 393, Nummer 9, September 2012, S. 843–851, doi:10.1515/hsz-2012-0167, PMID 22944686.