Jingmenvirus
| „Jingmenvirus“ | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
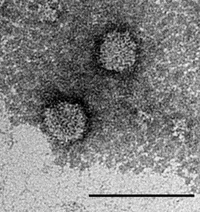
Elektronenmikroskopische Aufnahme von Alongshan-Viruspartikeln. Maßstabsbalken entspricht 100 nm.[3] | ||||||||||||||||
| Systematik | ||||||||||||||||
| ||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||
| ||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||
| „Jingmenvirus“ | ||||||||||||||||
| Links | ||||||||||||||||
|

„Jingmenvirus“ oder Jingmenvirus-Gruppe ist der Name einer vorgeschlagenen Gattung (ViralZone) bzw. Klade (NCBI-Taxonomie) von Viren aus der Familie der Flaviviridae. Eine Besonderheit ist, dass das RNA-Genom segmentiert ist: Es besteht nicht wie bei den meisten anderen Viren aus einem durchgehenden Nukleinsäuremolekül, sondern aus vier – in Einzelfällen auch aus fünf – getrennten Abschnitten.[4] Die Gruppe und ihre Mitglieder sind derzeit (Stand September 2023) noch nicht vom International Committee on Taxonomy of Viruses (ICTV) offiziell bestätigt.
Der erste Vertreter der Jingmenviren, JMTV (Jingmen Tick Virus; Tick ist das englische Wort für Zecke), wurde 2010 in China aus Zecken der Art Rhipicephalus microplus isoliert und 2014 in einer wissenschaftlichen Veröffentlichung beschrieben. Bei einem Vergleich der Sequenz der vier Genome mit Datenbanken ergab sich, dass ähnliche RNA-Sequenzen bereits bekannt waren: diese wurden einige Jahre zuvor aus Larven des Hundespulwurms isoliert, in denen sie als „reichlich vorhanden“ beschrieben (18 % der isolierten cDNA-Klone) und fälschlicherweise für entwicklungsbiologisch wichtige mRNA gehalten wurden, statt für eine Virusinfektion.[5][6][7]
Zahlreiche weitere Vertreter oder deren Erbgut wurden in den folgenden Jahren auf allen Kontinenten entdeckt und in verschiedenen Tiergruppen nachgewiesen, darunter neben Zecken und unterschiedlichen Insektenarten auch verschiedene Säugetiere, Schildkröten, Flusskrebse, Skorpione, Nematoden, Mehltau, Erbsen und einer Bodenprobe. Das Alongshan-Virus wurde aus Menschen mit einer fiebrigen Erkrankung in China isoliert und in Zecken (meist der Gattung Ixodes) in China und Europa nachgewiesen, auch in der Schweiz[8] und in Niedersachsen[9]. Da die Jingmenvirus-Gruppe erst vergleichsweise kurz bekannt ist, ist sie noch nicht ausreichend untersucht. Eine Vermehrung im Labor gelang nur manchmal.[10]
Übertragungswege[Bearbeiten | Quelltext bearbeiten]
Eine Übertragung von blutsaugenden oder pflanzensaugenden Zecken oder Insekten auf deren Wirte ist bei manchen Vertretern wahrscheinlich oder erscheint möglich, aber auch andere Mechanismen wie direkte Ansteckung von Viruswirt zu Viruswirt, etwa auf die Nachkommen, ist in manchen Fällen denkbar: JMTV wurde auch in Zeckenlarven gefunden, die noch kein Blut gesaugt hatten, so dass sie sich nicht bei einem Wirtstier angesteckt haben können. Hinzu kommt, dass der Hauptwirt von JMTV, die Zecke Rhipicephalus microplus, ihren Wirt während der individuellen Entwicklung nicht wechselt: Von der Larve bis zum Erwachsenenstadium bleibt die Zecke auf demselben Tier. Dies erschwert die Verbreitung des Virus von einem Wirbeltier zum nächsten. Anders der Hauptwirt des Alongshan-Virus, Ixodes persulcatus. Diese Zeckenart lässt sich nach einer Blutmahlzeit zu Boden fallen und sucht sich in jedem Entwicklungsstadium ein neues Wirtstier. In vielen Fällen gibt es keine Hinweise auf den Übertragungsweg (Stand 2022) und er könnte von Virustyp zu Virustyp stark variieren.[10]
Virale Gene und Proteine[Bearbeiten | Quelltext bearbeiten]

Das Genom der Viren der Jingmenvirus-Gruppe besteht meist aus vier Segmenten, die je nach Virus zusammen für bis zu sieben Strukturproteine und zwei Nicht-Struktur-Proteine codieren. Die Segmente 1 und 3 enthalten jeweils ein Gen, das große Ähnlichkeit mit den Proteinen NS3 beziehungsweise NS5 der Viren der Gattung Flavivirus hat. Daher wird die Gruppe in die Familie Flaviviridae eingeordnet. In Veröffentlichungen wird manchmal die Bezeichnungen der beiden Proteine aus den Flaviren übernommen[11], manchmal werden eigene Namen verwendet[10]. Das NS5-ähnliche Protein wird dann als NSP1 bezeichnet. „NSP“ steht für Nicht-Struktur-Protein, also ein Protein das kein Bestandteil des Kapsids oder der Virushülle ist. Es handelt sich um eine RNA-abhängige RNA-Polymerase. Das NS3-ähnliche Protein wird auch als NSP2 bezeichnet. Es ist eine Helikase und Serinprotease.[11][10]
Für die Gene der anderen Segmente sind keine Verwandten in anderen Virusgruppen bekannt. Die Genomsegmente 2 und 4 enthalten mehrere offene Leseraster (ORFs), von denen angenommen wird, dass sie Kapsid-Proteine, Hüllenproteine und Membranproteine codieren. Ihre Bezeichnung ist VP für Virusprotein mit einer anschließenden Zahl. Biochemische Analysen zeigten, dass die Viruspartikel eine Hülle haben.[11]
Für das Guaico Culex virus (GCXV), einen in Trinidad, Peru und Panama in Stechmücken der Gattung Culex gefundenen Virus, wurde gezeigt, dass die Genomsegmente in getrennten Partikel verpackt sind. Außerdem wurde für manche Isolate dieses Virus gezeigt, dass ein fünftes Genomsegment vorhanden sein kann.[11]
Während die RNAs von JMTV und ALSV polyadenyliert sind, ist dies bei den Insekten--assoziierten Jingmenviren nicht der Fall.[10]
| Segmenta | Längeb bei JMTV | Anzahl ORFsc | Named bei JMTV | Flavivirus-Homolog | Funktion(en) |
|---|---|---|---|---|---|
| 1 | 3072 | 1 | NSP1 | NS5 | RNA-abhängige RNA-Polymerase und Methyltransferase |
| 2 | 2785 | 2–3 | VP1, nuORF | - | vermutetes Hüllprotein, unbekannt |
| 3 | 2793 | 1 | NSP2 | NSB2/NS3 | Transmembrandomäne, Serinprotease und Helikase |
| 4 | 2751 | 2–3 | VP2, VP3 | - | vermutetes Capsidprotein, vermutetes Membranprotein |
| 5 (nur bei GCXV) | - (1260 bei GCXV) | 1 | VP7 | - | |
| a Bei manchen Viren, z. B. GCXV, sind die Segmente, die für die entsprechenden Proteine codieren in einer anderen Reihenfolge nummeriert. b Länge des Segments in Nukleotiden. c ORF, Open Reading Frame, offenes Leseraster. Hier ist die Zahl der möglichen Proteine angegeben, die von diesem Segment codiert werden können. d Name der codierten Proteine oder des ORFs. | |||||
Systematik und Vertreter[Bearbeiten | Quelltext bearbeiten]
Seit der Veröffentlichung des JMTV der Gruppe 2014 sind zahlreiche weitere Vertreter bekannt geworden. Die meisten publizierten Sequenzdaten stammen aus metagenomischen Studien, also nicht aus der Untersuchung reiner Virupartikel, sondern aus der Untersuchung aller in einem Tier, einer Pflanze oder einer Bodenprobe vorhandenen Nukleinsäuresequenzen. Vorhandene Stammbaumanalysen wurden generell an der Sequenz der beiden Nicht-Struktur-Proteine durchgeführt, die auch in verwandten Virengruppen vorkommen. Nach diesen Sequenzdaten lassen sich Jingmenviren in zwei Hauptgruppen unterteilen, die vorläufig als „Zecken- und Wirbeltier-assoziierte Jingmenviren“ und als „Insekten-assoziierte Jingmenviren“ bezeichnet werden. Diese Bezeichnungen sind jedoch ungenau: Vertreter der ersten Gruppe wurden auch in Stechmücken gefunden, die zu den Insekten gehören, Vertreter der zweiten auch in Krebsen, Pflanzen und Pilzen.[10]
Zecken- und Wirbeltier-assoziierte Jingmenviren[Bearbeiten | Quelltext bearbeiten]

JMTV – „Jingmen Tick Virus“[Bearbeiten | Quelltext bearbeiten]
JMTV ist der am längsten bekannte Vertreter der Gruppe. Alternative Namen sind Mogiana Tick Virus (MGTV), Guangxi tick virus, Amblyomma virus und Manych virus. Seit oder kurz vor der veröffentlichten Erstbeschreibung 2014 in China wurde er auch gefunden in Brasilien, Laos, Japan, Türkei, Italien, Kosovo, Rumänien, Russland, Trinidad und Tobago, Französische Antillen, Kolumbien, Uganda, Guinea und Kenia.[10]
JMTV wurde gefunden in 26 Zeckenarten aus mehreren Gattungen, Rindern, Primaten, elf Fledermausarten aus sechs Gattungen, elf Nagetierarten aus sieben Gattungen sowie kenianische Pantherschildkröten (Stigmochelys pardalis), und Menschen. Darunter waren drei Patienten im Kosovo und zwölf in China. Bei einigen der letzteren wurde ein juckendes oder schmerzhaftes Wundschorf und eine histologisch nachweisbare Nekrose an der Einstichstelle sowie häufig geschwollene Lymphknoten beschrieben, bei manchen hohes Fieber. Ob diese Symptome tatsächlich auf die JMTV-Infektion zurückzuführen sind oder andere Ursachen haben blieb jedoch unklar. Es liegt jedoch nahe, dass sich JMTV auch in Wirbeltieren und Menschen vermehren kann, auch wenn unklar ist in welchen Zelltypen und welche pathologischen Folgen dies hat.[12][10]
Alongshan-Virus[Bearbeiten | Quelltext bearbeiten]
Das „Alongshan-Virus“ oder ALSV wurde erstmals 2017 isoliert, aus der Blutprobe eines chinesischen Patienten mit einem Zeckenbiss. Die wissenschaftliche Veröffentlichung erfolgte 2019.[13] Das Genom unterscheidet sich insofern von JMTV, als dass der ORF des Genomsegments 2 kein durchgehendes VP1 kodiert, sondern zwei Proteine VP1a und VP1b. Ein potentieller „nuORF“ wie bei JMTV wurde hier ebenfalls gefunden. Die Nichtstrukturproteine NSP1 und NSP2 haben bei der Aminosäuresequenz 80 % Ähnlichkeit mit JMTV, die Strukturproteine nur 25–75 %. Auf Grund dieser Unterschiede wird ALSV als eigene Art angesehen.[10]
Eine nachträgliche Untersuchung von Blutproben von Patienten mit unklaren Symptomen wie Kopfschmerzen und Fieber und vorherigen Zeckenbissen aus der Region des ersten Nachweises ergab in 23 % der Fälle Hinweise auf ALSV. Alle Patienten waren vollständig genesen.[13][10] Wie bei JMTV ist unklar, ob die Symptome tatsächlich durch ALSV hervorgerufen wurden oder womöglich durch eine gleichzeitige Infektion mit einem anderen Erreger.[14]
ALSV wurde nachgewiesen in mehreren Zecken- und Stechmükenarten, in Schafen, Rindern, und einem Hirsch in den Ländern China, Russland, Finland, Deutschland[9], Schweiz[15], Frankreich und Serbien. In Nordostchina wiesen 9 % der Schafe und 5 % der Rinder Antikörper gegen ALSV auf. Hauptüberträger sind wohl Zecken der Gattung Ixodes, zu der der in Deutschland verbreitete Gemeine Holzbock gehört. In Europa sind Ixodes-Arten auch Hauptursache für die Übertragung von FSME.[10]
„Yanggou Tick Virus“[Bearbeiten | Quelltext bearbeiten]
Das „Yanggou Tick Virus“ (YGTV) wurde ebenfalls zuerst in China gefunden. Die Entdeckung von 2014 wurde nicht als wissenschaftlicher Artikel veröffentlicht, sondern nur in der Datenbank GenBank hochgeladen. Die RNA-Sequenzen konnten aus Zecken isoliert werden, von denen drei als Dermacentor nutalli bestimmt wurden. Eine Untersuchung an russischen Zecken, die zwischen 2011 und 2019 gesammelt wurden, fand YGTV ebenfalls in Dermacentor nutalli sowie in Dermacentor marginatus.[10]
Wie bei ALSV kodiert das zweite Segment für getrennte VP1a und VP1b-Proteine. Die Aminosäuresequenzen von NSP1 und NSP2 haben eine Ähnlichkeit von 80 % zu beiden, ALSV und JMTV. Bei den Strukturproteinen liegt die Ähnlichkeit bei 50–60 %.[10]
„Takachi-Virus“[Bearbeiten | Quelltext bearbeiten]
Die RNA-Sequenzen des „Takachi-Virus“ (engl. „Takachi virus“, TAKV) wurden in Japan aus Nymphen der Zeckenart Haemaphysalis formosensis isoliert, die 2019 und 2020 gesammelt wurden. Die Ähnlichkeiten der Aminosäuresequenzen zu JMTV und ALSV liegt bei 55–85 %.[10]
„Xinjiang Tick Virus 1“[Bearbeiten | Quelltext bearbeiten]
RNA des „Xinjiang Tick Virus 1“ (XJTV1) wurde aus nicht näher bestimmten Zecken aus China isoliert und die Sequenz 2021 direkt in GenBank eingegeben, ohne das nähere Informationen bekannt gemacht wurden (Stand 2022). Die Sequenzähnlichkeit ist zu YGTV am größten.[10]
„Pteropus lylei Jingmenvirus“[Bearbeiten | Quelltext bearbeiten]
Aus Urinproben von Fledermäusen der Art Pteropus lylei in Kambodscha würde eine vollständige Jingmenvirus-Sequenz isoliert und 2019 veröffentlicht. Die Sequenz des „Pteropus lylei Jingmenvirus“ (PLJV) wies eine Ähnlichkeit von 70–80 % bei NSP1 und NSP2 zu JMTV und ALSV auf sowie 40–50 % bei den Strukturproteinen. Wie bei JMTV (und anders als bei ALSV) kodiert Segment 2 für ein durchgehendes VP1.[10]
Guandong Jingmen-like Virus und Hainan Jingmen-like Virus[Bearbeiten | Quelltext bearbeiten]
Zwei weitere vollständige Virussequenzen der Jingmenvirus-Gruppe konnten in China aus Rinderkot und aus einer Bodenprobe isoliert werden. Diese wurden „Guandong Jingmen-like Virus“ (GDJLV) respektive „Hainan Jingmen-like Virus“ (HJLV) genannt. Aus welchem Wirt diese 2022 veröffentlichten Viren stammen ist unklar.
Teilsequenzen[Bearbeiten | Quelltext bearbeiten]
RNA-Sequenzen des Newport Tick Virus (NTV) oder Ixodes holocylus Jingmenvirus wurden in einer metagenomischen Untersuchung an Zecken aus New South Wales in Australien gefunden und 2022 veröffentlicht. Diese Zecken der Art Ixodes holocyclus wurden in Kioloa und Sydney gesammelt. Bisher sind nur Sequenzen von Strukturproteinen bekannt. Die längste, 2087 Nukleotid lange Sequenz zeigt zwischen 27 und 55 % Homologie mit den anderen Jingmenviren. Eine weitere Teilsequenz wurde aus der Weißfußmaus (Peromyscus leucopus) in den USA isoliert und 2020 veröffentlicht (FLSV). Sie beinhaltet Teile von NSP1 und NSP2. Die Ähnlichkeit der Aminosäuresequenzen zu JMTV und ALSV liegt unter 70 %.[10]
Insekten-assoziierte Jingmenviren[Bearbeiten | Quelltext bearbeiten]

Zu den Insekten-assoziierten Jingmenviren zählen mit Stand 2022 etwa 40 Vertreter, von denen die RNA-Sequenz isoliert wurde. Nur in zwei Fällen, GCXV und MoCV, wurden tatsächlich Virenpartikel beschrieben, die sich auch in Zellkulturen vermehren ließen. Die anderen Vertreter gelten daher vorerst als „mutmaßliche Viren“. Die Zuordnung zu bestimmten Wirten ist häufig nicht gesichert: Eine Sequenz, die aus einer Stechmücke isoliert wurde, kann auch aus dem Blut ihres letzten Opfers stammen.[10]
Bei einer kleinen Gruppe mit nur drei Vertretern ähnelt die Sequenz stärker den Zecken- und Wirbeltier-assoziierte Jingmenviren als den anderen Insektenassoziierten Jingmenviren (siehe Abbildung). Hierzu gehört Toxoplasmacanis larvae agent, TCLA, dessen RNA wie bereits beschrieben in Larven des Hundespulwurms gefunden wurde. Plasmopara viticola lesion associated Jingmen-like virus 1 wurde 2018 in Italien aus einem Pilz isoliert, genauer Plasmopara viticola, dem falschen Mehltau der Weinrebe. Der dritte Vertreter ist Histiostoma Jingmenvirus, dessen Sequenz aus einer 2014 in Deutschland gesammelten Milbe (siehe Histiostomatidae) isoliert wurde.[10]
Die Sequenzen der restlichen Vertreter der Insekten-assoziierte Jingmenviren sind stärker miteinander verwandt als mit den Zecken- und Wirbeltier-assoziierte Jingmenviren und der genannten Dreiergruppe. Die Ähnlichkeit der Sequenzen folgt dabei nicht der Verwandschaftsnähe der Wirte, in denen sie gefunden wurden, anders als dies bei insektenspezifischen Flaviviren der Fall ist. So finden sich mitten in der Gruppe auch Sequenzen, die aus Pflanzen, Skorpion und Krebs isoliert wurden. Die nächst-ähnlichen bekannten Jingmenviurs-Sequenzen wurden jeweils aus Insekten isoliert. Dies legt nahe, dass sich die Viren nicht parallel zu den Wirten, in denen sie gefunden wurden, evolutionär entwickelt haben.[10]
Guaico Culex Virus, GCXV[Bearbeiten | Quelltext bearbeiten]
GCXV wurde in verschiedenen Stechmückenarten der Gattung Culex aus Trinidad, Peru, Panama und Brasilien gefunden. Das Genom besteht aus vier oder fünf RNA-Segmenten, die nicht polyadenyliert sind. Segment 1 codiert auch bei GCXV NSP1. Abweichend von JMTV und ALSV wird das NSP2-codierende Segment hier als Segment 2 bezeichnet, Segment 3 codiert für VP1, VP2 und VP3, und Segment 4 für VP4, VP5 und VP6. Wenn vorhanden kodiert Segment 5 für VP7. Dabei überlappen die ORFs von VP1 und VP5 jeweils mit den beiden anderen ORFs des Segments. Von VP1-7 wird angenommen, dass es sich um Strukturproteine handelt.[10]
GCXV konnte in drei Zelllinen aus Culex und Aedes vermehrt werden, und auch in erwachsenen Tieren dieser Gattungen. Eine Vermehrung in Hirschzecken und der zu den Schmetterlingsmücken gehörenden Sandfliege Lutzomyia longipalpis gelang jedoch nicht. Eine wesentliche Weitergabe von erwachsenen Stechmücken an deren Nachkommen konnte nicht beobachtet werden. Laborversuche zeigten, dass eine Virusvermehrung mit und ohne Segment 5 möglich ist. GCXV ist insofern bemerkenswert, als seine Segmente in getrennte Viruspartikel verpackt werden können, die 30 - 35 nm Durchmesser haben. Die Übertragung erfolgt somit über ein multi-komponenten-System. Im Vergleich zu den Zecken- und Wirbeltier-assoziierte Jingmenviren kommt GCXV mit relativ niedriger Anzahl im Wirt vor. Es gibt keine Hinweise auf eine Verbindung zu Wirbeltieren.[10]
Mole Culex Virus, MoCV[Bearbeiten | Quelltext bearbeiten]
MoCV wurde 2016 im westafrikanischen Ghana aus Culex-Arten isoliert und in Zellkulturen von Stechmückenzellen vermehrt. Die Genomsequenz ist mit GCXV nah verwandt, bei NSP1 und NSP2 liegt die Ähnlichkeit auf Aminosäure-Ebene bei 80 und 70 %. Bei den Strukturproteinen liegt sie zwischen 50 und 80 %. Wie bei GCXV ist die Anzahl der Viren im Wirt im Vergleich zu den Zecken- und Wirbeltier-assoziierte Jingmenviren niedrig, auch bei MoCV gibt es keine Hinweise auf eine Verbindung zu Wirbeltieren.[10]
Wuhan aphid virus 2, WHAV2[Bearbeiten | Quelltext bearbeiten]
Eine umfangreiche Studie an verschiedenartigen Wirtsorganismen fand etliche Sequenzen von Flaviviridae, darunter mehrere Jingmenviren und speziell die des „Wuhan aphid virus 2“ bei zwei Blattlausarten, Hyalopterus pruni und Aulacorthum magnoliae.[16] Erstaunlicherweise wurde das WHAV2-Genom später auch aus Pflanzen in Europa isoliert. Eine französische Arbeitsgruppe infizierte unter anderem Erbsenpflanzen mit verschiedenen Viren um Nachweismethoden zu testen. Neben den künstlich zugegebenen Viren wurden aber zwei weitere isoliert, darunter ein Jingmenvirus, WHAV2. Die Sequenzähnlichkeit der Aminosäuren zum in Wuhan gefundenen Virus betrug je nach Segment zwischen 90 und 97 %. Diese starke Ähnlichkeit legt nahe, dass das Virus zwischen Blattläusen und Pflanzen übertragen werden kann. Die Autoren der 2020 erschienen Arbeit geben an, dass WHAV2-Segmente auch in drei weiteren Erbsenpflanzen aus Deutschland und Österreich gefunden wurden, die 2012 und 2013 gesammelt wurden.[17][10]
Weitere Vertreter[Bearbeiten | Quelltext bearbeiten]
Zahlreiche weitere Vertreter wurden in Insekten gefunden, darunter drei Drosophila-Arten inklusive Drosophila melanogaster, Florfliegen, Clogmia albipunctata, dem Katzenfloh, der Röhrenblattlaus Hyalopterus pruni, den Blattläusen Rhopalosiphum maidis, Rhopalosiphum padi und Sitobion avenae, dem Fransenflügler Neohydatothrips variabilis, einer Schwertschrecke der Gattung Conocephalus, einer Gnitze der Gattung Culicoides. In Deutschland wurden Jingmenviren-Sequenzen im Wasserzwerg, in Ulopa reticulata, Tachycixus pilosus, im Doppelschwanz Campodea silvestrii und in einer Hornmilbe der Gattung Histiostoma gefunden, in Österreich in der Florfliege Pseudomallada ventralis.[10]
Weitere Jingmenvirus-Sequenzen wurden aus dem Roten Amerikanischen Sumpfkrebs (Changiang Jingmen-like virus), dem Skorpion Euscorpius sicanus (siehe Euscorpius; Arachnidian jingmen-related virus) und dem Echten Mehltau der Weinrebe (Erysiphe necator associated ssRNA virus) isoliert.[10]
Äußere Systematik[Bearbeiten | Quelltext bearbeiten]

Einzelnachweise[Bearbeiten | Quelltext bearbeiten]
- ↑ a b c d ICTV: ICTV Taxonomy history: Yellow fever virus, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- ↑ ICTV Master Species List 2018b.v2. MSL #34, März 2019
- ↑ Ivan S. Kholodilov, Alexander G. Litov, Alexander S. Klimentov, Oxana A. Belova, Alexandra E. Polienko, Nikolai A. Nikitin, Alexey M. Shchetinin, Anna Y. Ivannikova, Lesley Bell-Sakyi, Alexander S. Yakovlev, Sergey V. Bugmyrin, Liubov A. Bespyatova, Larissa V. Gmyl, Svetlana V. Luchinina, Anatoly P. Gmy, Vladimir A. Gushchin, Galina G. Karganova: Isolation and Characterisation of Alongshan Virus in Russia. In: MDPI: Viruses, Band 12, Nr. 4, April 2020, S. 362; doi:10.3390/v12040362, PMID 32224888, PMC 7232203 (freier Volltext), Epub26. März 2020 (englisch).
- ↑ a b Suvi Kuivanen, Lev Levanov, Lauri Kareinen, Tarja Sironen, Anne J. Jääskeläinen, Ilya Plyusnin, Fathiah Zakham, Petra Emmerich, Jonas Schmidt-Chanasit, Jussi Hepojoki, Teemu Smura, Olli Vapalahti: Detection of novel tick-borne pathogen, Alongshan virus, in Ixodes ricinus ticks, south-eastern Finland, 2019. In: Eurosurveillance, Band 24, Nr. 27, 4. Juli 2019, S. 1900394; doi:10.2807/1560-7917.ES.2019.24.27.1900394, PMID 31290392, PMC 6628756 (freier Volltext) (englisch).
- ↑ Xin-Cheng Qin, Mang Shi, Jun-Hua Tian, Xian-Dan Lin, Dong-Ya Gao, Jin-Rong He, Jian-Bo Wang, Ci-Xiu Li, Yan-Jun Kang, Bin Yu, Dun-Jin Zhou, Jianguo Xu, Alexander Plyusnin, Edward C. Holmes, and Yong-Zhen Zhang: A tick-borne segmented RNA virus contains genome segments derived from unsegmented viral ancestors. In: Proceedings of the National Academy of Sciences of the United States of America. Band 111, Nr. 18, 21. April 2014, S. 6744–6749, doi:10.1073/pnas.132419411.
- ↑ Hayley M. Bennett: Split reality for novel tick virus. In: Nature Reviews Microbiology. Band 12, 9. September 2014, S. 464, doi:10.1038/nrmicro3297.
- ↑ Deborah M Callister, Alan D Winter, Antony P Page, Rick M Maizels: Four abundant novel transcript genes from Toxocara canis with unrelated coding sequences share untranslated region tracts implicated in the control of gene expression. In: Molecular and Biochemical Parasitology. Band 162, Nr. 1, 25. Juli 2008, S. 60–70, doi:10.1016/j.molbiopara.2008.07.004.
- ↑ Stefanie Stegmüller, Cornel Fraefel, Jakub Kubacki: Genome Sequence of Alongshan Virus from Ixodes ricinus Ticks Collected in Switzerland. In: Microbiology Resource Announcements. Band 12, Nr. 3, 16. März 2023, S. e0128722, doi:10.1128/mra.01287-22, PMID 36779723, PMC 10019301 (freier Volltext).
- ↑ a b Cara Leonie Ebert, Lars Söder, Mareike Kubinski, Julien Glanz, Eva Gregersen, Katrin Dümmer, Domenic Grund, Ann-Sophie Wöhler, Laura Könenkamp, Katrin Liebig, , Steffen Knoll, Fanny Hellhammer, Anna-Katharina Topp, Paul Becher, Andrea Springer, Christina Strube, Uschi Nagel-Kohl, Marcel Nordhoff, Imke Steffen, Benjamin Ulrich Bauer, Martin Ganter, Karsten Feige, Stefanie C. Becker, Mathias Boelke: Detection and Characterization of Alongshan Virus in Ticks and Tick Saliva from Lower Saxony, Germany with Serological Evidence for Viral Transmission to Game and Domestic Animals. In: Microorganisms. Band 11, Nr. 3, 21. Februar 2023, 543, doi:10.3390/microorganisms11030543, PMID 36985117.
- ↑ a b c d e f g h i j k l m n o p q r s t u v w x y z aa ab Agathe M. G. Colmant, Rémi N. Charrel, Bruno Coutard: Jingmenviruses: Ubiquitous, understudied, segmented flavi-like viruses. In: Front. Microbiol. Band 13, 10. Oktober 2022, 997058, doi:10.3389/fmicb.2022.997058.
- ↑ a b c d Naomi Attar: Animal viruses in pieces. In: Nature Reviews Microbiology. Band 14, 6. September 2016, S. 607, doi:10.1038/nrmicro.2016.133 (englisch).
- ↑ Na Jia, Hong-Bo Liu, Xue-Bing Ni, Lesley Bell-Sakyi, Yuan-Chun Zheng, Ju-Liang Song, Jie Li, Bao-Gui Jiang, Qian Wang, Yi Sun, Ran Wei, Ting-Ting Yuan, Luo-Yuan Xia, Yan-Li Chu, Wei Wei, Lian-Feng Li, Jin-Ling Ye, Qing-Yu Lv, Xiao-Ming Cui, Yi Guan, Yi-Gang Tong, Jia-Fu Jiang, Tommy Tsan-Yuk Lam, Wu-Chun Cao: Emergence of human infection with Jingmen tick virus in China: A retrospective study. In: EBioMedicine. Band 43, 17. April 2019, S. 317–324, doi:10.1016/j.ebiom.2019.04.004, PMID 31003930, PMC 6557783 (freier Volltext) – (englisch).
- ↑ a b Ze-Dong Wang, Bo Wang, M.D., Feng Wei, Shu-Zheng Han, M.S., Li Zhang, B.Sc., Zheng-Tao Yang, Yan Yan, Xiao-Long Lv, M.S., Liang Li, Shu-Chao Wang, Ming-Xin Song, Hao-Ji Zhang, Shu-Jian Huang, Jidang Chen, Fu-Qiang Huang, Shuang Li, B.Sc., Huan-Huan Liu, B.Sc., Jian Hong, Yu-Lan Jin, Wei Wang, M.S., Ji-Yong Zhou, and Quan Liu, Ph.D.: A New Segmented Virus Associated with Human Febrile Illness in China. In: New England Journal of Medicine. Band 380, 30. Mai 2019, S. 2116–2125, doi:10.1056/NEJMoa1805068.
- ↑ Cornel Fraefel: Fakten zum ALS-Virus. Kasten im Artikel „Neues Virus in Schweizer Zecken“ von Eva Mell. In: Schweizerische Ärztezeitung. Band 104, Nr. 03, 7. Januar 2023, S. 8–9, doi:10.4414/saez.2023.21434 ([1]).
- ↑ Stefanie Stegmüller, Cornel Fraefel, Jakub Kubacki: Genome Sequence of Alongshan Virus from Ixodes ricinus Ticks Collected in Switzerland. In: Am journals: Microbiology Resource Announcements, Band 12, Nr. 3, 16. März 2023, S. e0128722; doi:10.1128/mra.01287-22, PMID 36779723, PMC 10019301 (freier Volltext), Epub 13. Februar 2023 (englisch).
- ↑ Mang Shi, Xian-Dan Lin, Nikos Vasilakis, Jun-Hua Tian, Ci-Xiu Li, Liang-Jun Chen, Gillian Eastwood, Xiu-Nian Diao, Ming-Hui Chen, Xiao Chen, Xin-Cheng Qin, Steven G. Widen, Thomas G. Wood, Robert B. Tesh, Jianguo Xu, Edward C. Holmes, Yong-Zhen Zhang: Divergent Viruses Discovered in Arthropods and Vertebrates Revise the Evolutionary History of the Flaviviridae and Related Viruses. In: J Virol. Band 90, Nr. 2, 30. Dezember 2015, S. 659-69, doi:10.1128/JVI.02036-15, PMID 26491167, PMC 4702705 (freier Volltext) – (englisch).
- ↑ Yahya Zakaria Abdou Gaafar, Heiko Ziebell: Comparative study on three viral enrichment approaches based on RNA extraction for plant virus/viroid detection using high-throughput sequencing. In: PLoS One. Band 15, Nr. 8, 25. August 2020, S. e0237951, doi:10.1371/journal.pone.0237951, PMID 32841302, PMC 7447037 (freier Volltext) – (englisch).