Synthetische Biologie
Die synthetische Biologie ist ein Fachgebiet im Grenzbereich von Molekularbiologie, organischer Chemie, Ingenieurwissenschaften, Nanobiotechnologie und Informationstechnik. Sie wird von einigen ihrer Vertreter als die neueste Entwicklung der modernen Biologie beschrieben.[1][2]
Im Fachgebiet synthetische Biologie arbeiten Biologen, Chemiker und Ingenieure zusammen, um biologische Systeme zu erzeugen, die in der Natur nicht vorkommen. Der Biologe wird so zum Designer von einzelnen Molekülen, Zellen und Organismen, mit dem Ziel, biologische Systeme mit neuen Eigenschaften zu erzeugen.[1][3][4]
Dabei werden verschiedene Strategien verfolgt:
- Künstliche, biochemische Systeme werden in Lebewesen integriert, die dadurch neue Eigenschaften erhalten.
- Entsprechend den biologischen Vorbildern werden schrittweise chemische Systeme so aufgebaut, dass sie bestimmte Eigenschaften von Lebewesen aufweisen (biomimetische Chemie).
- Organismen werden auf ihre allernotwendigsten Systemkomponenten reduziert (Minimalgenom), die als eine Art „Gerüst“ dienen, um durch den Einbau von sogenannten bioparts biologische Schaltkreise zu erzeugen.
Im Unterschied zur Gentechnik werden nicht nur z. B. einzelne Gene von Organismus A zu Organismus B transferiert, sondern das Ziel der synthetischen Biologie ist es, komplette künstliche biologische Systeme zu erzeugen. Diese Systeme sind der Evolution unterworfen, sollen aber bis zu einem gewissen Grad „mutationsrobust“ gemacht werden.[5]
Geschichte der synthetischen Biologie
[Bearbeiten | Quelltext bearbeiten]Bereits 1912 veröffentlichte der Franzose Stéphane Leduc (1853–1939) eine Arbeit mit dem Titel „La Biologie Synthétique“, die sich mit der Bildung von pflanzenartigen Formen beschäftigte, wenn man Schwermetallsalze in eine wässrige Lösung von Natriumsilicat gibt, ein so genannter Chemischer Garten. Im Unterschied dazu formulierte der Chemiker Emil Fischer bereits Ende des 19. Jahrhunderts das Programm einer gezielten chemischen Veränderung von Organismen für technische Zwecke, für das er 1915 erstmals den Ausdruck „chemisch-synthetische Biologie“ verwendete.[6]
1978 schrieb Waclaw Szybalski in seinem Kommentar zum Nobelpreis in Medizin für die Arbeiten von Werner Arber, Daniel Nathans und Hamilton Smith[7] über Restriktionsenzyme: “The work on restriction nucleases not only permits us easily to construct recombinant DNA molecules and to analyze individual genes but also has led us into ‚the new era of synthetic biology‘ where not only existing genes are described and analyzed but also new gene arrangements can be constructed and evaluated.”[8]
1980 verwendete die Biologin und Wissenschaftsjournalistin Barbara Hobom den Begriff bei der Beschreibung rekombinanter Bakterien als Synonym für die Anwendung gentechnologischer Methoden.[9]
2000 bezeichnete Eric Kool auf dem Jahrestreffen der American Chemical Society in San Francisco die Integration künstlicher chemischer Systeme in Lebewesen als synthetische Biologie.[10] und etablierte damit das heutige Verständnis dieses Begriffs.
2004 fand die erste internationale wissenschaftliche Konferenz zur synthetischen Biologie am Massachusetts Institute of Technology (MIT) in Cambridge, Massachusetts/USA statt, die SB 1.0. Darauf folgten im Jahr 2006 die SB 2.0 (Berkeley, USA), 2007 die SB 3.0 (Zürich, Schweiz) sowie 2008 die SB 4.0 (Hongkong, China). Die SB 5.0 fand im Juni 2011 auf dem Gelände der Stanford University in Kalifornien, USA, statt.[11]
2007 beantragten Forscher des J. Craig Venter Institute (Rockville, Maryland) in den USA Patentschutz für ca. 400 Gene, die ein im Labor konstruiertes Bakterium „betreiben“ sollen. Laut Patentantrag sollen die eigenen Gene des Bakteriums zunächst entfernt und anschließend die im Labor zusammengestellten Gene eingebracht werden, mit dem möglichen Ziel, Wasserstoff oder Ethanol zu produzieren.[12]
Im Januar 2008 berichtete eine Forschergruppe um Craig Venter, es sei erstmals gelungen, das Erbmaterial eines Bakteriums komplett synthetisch herzustellen. Vorbild für den Nachbau des Genoms sei Mycoplasma genitalium gewesen, als Name des synthetischen Nachbaus wurde Mycoplasma genitalium JCVI-1.0 gewählt.[13]
Im Mai 2010 veröffentlichten Forscher des J. Craig Venter Institute, ihnen sei es gelungen, das vollständige, im Labor synthetisierte Genom des Bakteriums Mycoplasma mycoides in eine DNA-freie Zelle von Mycoplasma capricolum einzuführen. Das synthetische Genom habe in der Zelle für Eiweiße von Mycoplasma mycoides codiert, so dass die Bakterienzellen sich normal vermehrten.[14]
Konstruktion von DNA
[Bearbeiten | Quelltext bearbeiten]Die Abwandlung des Konstruktionsprinzips der natürlichen DNA und ihr Verhalten in Bakterien führte einerseits zur Revision einiger Modellvorstellungen und ergab andererseits eine neue Diagnosemöglichkeit für Aids und Hepatitis (branched DNA diagnostic assay).
Ein neues Rückgrat
[Bearbeiten | Quelltext bearbeiten]Um die Möglichkeiten zu erforschen, Gene mit Hilfe der Antisensetechnik gezielt auszuschalten, wurde in den 1980er Jahren mit modifizierter DNA experimentiert. Da das Rückgrat eines Polynukleotidstranges aus stark wasseranziehenden (hydrophilen) Bausteinen (Phosphat und – bei der DNA – Desoxyribose-Zucker) besteht, wurden diese durch fettanziehende (lipophile) Bausteine ersetzt, um die Moleküle durch die innere, lipophile Schicht der Zellmembran hindurchschleusen zu können. Man ging dabei davon aus, dass das veränderte Rückgrat keine Auswirkung auf die Erkennung der komplementären Basen durch die Polymerasen hat. Der Ersatz der Rückgratbausteine führte aber zu vielen fehlerhaften Basenpaarungen bei der Verdopplung (Replikation) der DNA. So ergab der Ersatz der Nukleotide durch PNAs (polyamide-linked nucleic-acid analogues, den natürlichen Nukleinsäuren analoge Moleküle, deren Basen durch Polyamide verknüpft sind) Ketten, die nicht mehr als 15 bis 20 Bausteine enthielten. Der Ersatz der Ribose durch andere, weniger hydrophile Moleküle ergab ähnliche Ergebnisse.
Allerdings verhielten sich Nukleinsäuren mit Threose stabiler als ihre natürlichen Vorbilder. In natürlichen Systemen ist dies unter Umständen nicht sinnvoll, da eine zu hohe Stabilität der Basenpaarungen die Variationsmöglichkeiten und damit die Anpassungsfähigkeit durch Evolution einschränken.
Inzwischen ist gesichert, dass das Phosphat-Ribose-Gerüst eine wichtige Rolle für das Erkennungssystem der Polymerasen spielt.
Erweiterung des genetischen Codes
[Bearbeiten | Quelltext bearbeiten]Zusätzliche Nukleinbasen
[Bearbeiten | Quelltext bearbeiten]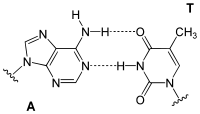
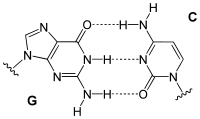
Der genetische Code beruht auf Triplett-Codons von vier verschiedenen Basen, von denen jeweils zwei zueinander komplementär sind und ein Basenpaar bilden können. Basis der Komplementarität sind Zahl und räumliche Ausrichtung der funktionellen Gruppen, welche die Wasserstoffbrückenbindungen zwischen den Purin- und Pyrimidinbasen ermöglichen. Dabei fungiert ein Wasserstoff der Aminogruppe (–NH2) oder der Wasserstoff des Stickstoffs im Heterozyklus als Donor (D), Carbonylsauerstoff oder heterozyklischer Stickstoff mit einem nichtbindenden Elektronenpaar als Akzeptor (A).
Theoretisch ergeben sich für die Kombination von Purinen (pu) mit komplementären Pyrimidinen (py) 12 Kombinationsmöglichkeiten für drei Wasserstoffbrückenbindungen:
| py | pu | py | pu |
|---|---|---|---|
| AAD | DDA | DDA | AAD |
| ADA (wie bei Thymin) | DAD (wie bei Adenin) | DAD | ADA |
| ADD | DAA | DAA (wie bei Cytosin, hier allerdings DA-) | ADD (wie bei Guanin) |
Damit ist es möglich, den genetischen Code um vier neue Basenpaare zu erweitern, was zu 448 neuen Codons führt. Mit einer modifizierten Polymerase lässt sich eine DNA, die auch diese Basenpaare enthält, replizieren.
Untersuchungen mit diesem modifizierten Replikationssystem haben gezeigt, dass die DNA-Reparaturenzyme nicht, wie das bis dahin gültige Modell vorschlug, die kleine Furche der DNA-Doppelhelix entlangwandern und den Nukleotidstrang nach nichtbindenden Elektronenpaaren der Wasserstoffbrücken-Donatoren absuchen.
Quadruplett- und Pentaplett-Codons
[Bearbeiten | Quelltext bearbeiten]Eine andere Möglichkeit ist bzw. wäre durch eine Verwendung von Basen-Quadrupletts[15][16] oder gar Pentapletts als Codons gegeben.[17]
Konstruktion von RNA
[Bearbeiten | Quelltext bearbeiten]Ein Anwendungsbeispiel ist die Verwendung von modifizierter mRNS als Impfstoff gegen den Erreger von COVID-19, SARS-CoV-2,[18][19] wie sie von der Firmen-Kooperation Biontech/Pfizer/Fosun (Tozinameran, Handelsname Comirnaty)[20][21] und Moderna (mRNA-1273),[22][23] sowie von anderen Unternehmen[24] entwickelt wurden. Der Impfstoff Zorecimeran des Herstellers Curevac verwendet dagegen unmodifizierte RNA.
Konstruktion von Enzymen
[Bearbeiten | Quelltext bearbeiten]Bereits 1983 begann Kevin Ulmer mit den Grundzügen des Proteindesigns die Aminosäuresequenzen von Enzymen zu verändern, um neue katalytische Eigenschaften erzeugen zu können. Ziel war es bestimmte, in verschiedenen Enzymen immer wieder vorkommende, Domänen wie α-Helix oder β-Faltblatt als Grundbausteine zu isolieren und sie so zu modifizieren, dass sie als Module wie bei einem Baukastensystem zu verschiedenen neuen Enzymen mit vorbestimmten Eigenschaften zusammengesetzt werden können.[25] Da die Wechselwirkungen vor allem zwischen weiter entfernten Aminosäureresten zurzeit kaum bekannt sind, konnte dieses Ziel nicht erreicht werden. Allerdings wurden bei diesen Forschungen Enzyme entwickelt, die als Polymerase für die DNA-Sequenzierung, als reverse Transkriptase für die Vermehrung künstlicher genetischer Systeme und als Enzyme in Waschmitteln Verwendung finden.
Konstruktion von Stoffwechselwegen
[Bearbeiten | Quelltext bearbeiten]Mit Hilfe der seit 1973 verfügbaren, von Herbert W. Boyer und Stanley N. Cohen entwickelten Rekombinationstechnik[26] können transgene Organismen bestimmte Stoffe synthetisieren, die sie mit ihrer natürlichen Genausstattung nicht hätten herstellen können. Dabei wird in der Regel ein Strukturgen mit dem entsprechenden Promotor und Steuergenen in das Wirtsgenom integriert. Mit Hilfe des vorhandenen Stoffwechselweges wird dieses Gen exprimiert, der gewünschte Stoff hergestellt (Beispiel: Synthese von Insulin durch das Bakterium Escherichia coli).
Die synthetische Biologie geht bei der Konstruktion neuer Stoffwechselwege noch einen Schritt weiter. Ziel ist ein modulares System von exprimierbaren Genen, die in einem Wirtsorganismus je nach Bedarf zusammengefügt werden und nicht nur die vorhandenen, natürlichen Stoffwechselwege nutzen, sondern auch neue in der Zelle etablieren.
Da auch hier die zahlreichen Wechselwirkungen zwischen den zahlreichen Stoffwechselwegen und ihrer Regulation nur unzureichend bekannt sind, konnte bis jetzt ein Modulares System nicht aufgebaut werden.

Es konnte aber in einem Escherichia-coli-Stamm der Stoffwechselweg von Acetyl-CoA zu Amorphadien eingerichtet werden. Dieser Stoff ist eine Vorstufe zu Artemisinin, das als Medikament gegen Malaria eingesetzt werden kann. Für diesen Stoffwechselweg wurden Gene aus der Blutregenalge (Haematococcus pluvialis) und Hefe (Saccharomyces cerevisiae) verwendet.
Artemisin wird vom Einjährigen Beifuß (Artemisia annua), allerdings nur in ungenügenden Mengen, hergestellt.
Konstruktion von biochemischen Signalwegen
[Bearbeiten | Quelltext bearbeiten]Grundlage ist die Regulation der Genexpression, bei der zum Beispiel ein Signalmolekül ein die Transkription blockierendes Protein (Repressor) dergestalt verändert, dass es den Weg für die RNA-Polymerase und damit für die Enzymsynthese eines bestimmten Stoffwechselweges freigibt. Dieser Stoffwechselweg produziert wieder Moleküle, die ihrerseits als Signalmoleküle für Induktion oder Repression der Expression bestimmter Gene dienen können.
Die erste Kombination von zwei verschiedenen Signalwegen gelang mit der Verknüpfung von zwei in der Natur nicht voneinander abhängigen Mechanismen: So konnte durch ein Signal, das eigentlich Wachstum fördert, der Zelltod ausgelöst werden.[27]
Der Zugang von Tom Knight, Andrew (Drew) David Endy und Randy Rettberg (MIT Cambridge, USA) ist die Schaffung von modularen genetischen Einheiten, den sogenannten „BioBricks“ („biologische Bausteine“). Diese Biobricks sollen, wenn sie in das Genom z. B. eines Bakteriums eingefügt werden, vorher-definierte Aufgaben erfüllen, analog etwa den elektronischen Schaltkreisen, wie sie in Mikroprozessoren (Computer) zu finden sind, daher auch die Bezeichnung „biological circuits“. Die Idee von Biobricks ist die, dass die Biobricks in dem biologischen Schaltkreis immer eine ganz bestimmte Aufgabe erfüllen, unabhängig von der (zellulären oder genetischen) Umgebung. Allerdings ist nicht sicher, ob die Kontext-Unabhängigkeit in einem komplexen biologischen Schaltkreis hinreichend gewährleistet werden kann, Biobricks befindet sich nach wie vor in einer experimentellen Phase.[28]
Endy konstruierte einen genetischen Schaltkreis, der Bakterien periodisch aufleuchteten ließ. Dieser sogenannte „Repressilator“ besteht aus drei verschiedenen BioBricks, die sich gegenseitig hemmen. Ein Baustein produziert auch ein Fluoreszenz-Protein. Die zeitliche Verzögerung bewirkt in diesem Schaltkreis eine Oszillation:[4]

Zwar ahmen diese genetischen Schaltkreise technische nach, es bestehen aber grundsätzliche Unterschiede, die eine technologische Anwendung erschweren:
- Genetische Schaltkreise sind in das Genom von Lebewesen integriert, die sich vermehren und mit der Umwelt in Wechselwirkung stehen. Die Fremdgene sind damit der Evolution ausgesetzt. So konnte man beobachten, dass nach einer Stunde bereits 58 % der manipulierten Zellen nicht mehr das gewünschte Verhalten zeigten.
- Die Bausteine stellen Fremdkörper in den Wirtsorganismen dar. Sie entziehen für ihre Funktion den Zellen Nährstoffe, so dass die Vitalität der Wirte vermindert ist.
- Die Bausteine erhöhen die Komplexität der Wirtszelle. Diese Komplexität erlaubt in der Regel keine exakten Voraussagen wie in den entsprechenden einfachen, ausschließlich auf Naturgesetzen beruhenden technischen Systemen.
- In technischen Systemen können Signale auf Grund der Verdrahtung gezielt an ihren Bestimmungsort gebracht werden. In biologischen Systemen wirkt sich ein Signalmolekül auf alle Systemelemente aus, die den entsprechenden Rezeptor aufweisen. Wenn also die gleichen Module an verschiedenen Stellen eines Schaltkreises oder in verschiedenen Schaltkreisen verwendet werden, kommt es zu unerwünschten Wechselwirkungen. Dieses Problem wird dadurch umgangen, dass für jeden Schaltkreis in einem Organismus verschiedene BioBricks für dieselbe Funktion eingebaut werden müssen. Dies führt zu einer unerwünschten Vervielfachung der Zahl der Module. In manchen Systemen wird deshalb statt eines Signalmoleküls die Transkriptionsrate der Module (TIPS, transcription initialisings per second) benutzt.
- Die Entwicklung des „Repressilators“ dauerte zwei Jahre. Für die Entwicklung eines E. coli Stammes mit einer Flipflop-Schaltung benötigte James J. Collins ein Jahr.
Die Nachahmung elektronischer Schaltkreise erscheint zunächst wie eine Spielerei. Neben dem Erkenntnisgewinn über intrazelluläre Informationswege bestehen aber auch Möglichkeiten zu technologischen Anwendungen. So ist es denkbar, dass mit entsprechenden genetischen Schaltkreisen versehene Bakterien die Anwesenheit von Landminen anzeigen, wobei das Signal eine bestimmte Konzentration von TNT im Boden und die Reaktion ein Aufleuchten in von der Konzentration des TNT abhängigen Farben ist.
Konstruktion von Zellen
[Bearbeiten | Quelltext bearbeiten]Forscher arbeiten daran, künstliche Chloroplasten, Komponenten von Pflanzenzellen, die Photosynthese betreiben, zu entwickeln. Ein Team kombinierte dazu ein Photosynthesesystem aus Spinat-Chloroplasten mit einem Bakterienenzym und einem künstlichen Enzym-Netzwerk. Das System ist energieeffizienter als natürliche Chloroplasten und könnte diverse Anwendungsmöglichkeiten haben.[29][30] Auch an der Entwicklung synthetischer roter Blutzellen wird geforscht. Diese ermöglichen etwa das Aufladen funktioneller Frachten wie Hämoglobin, Nanopartikel, Biosensoren und Medikamente.[31][32]
Genom-Komplettsynthese
[Bearbeiten | Quelltext bearbeiten]Die ersten Genomsynthesen basierten auf der Fusions-PCR, einer Methode zur Erzeugung von Genen aus Oligonukleotiden.[33]
- 2002 setzten Jeronimo Cello, Aniko V. Paul und Eckard Wimmer infektiöse Polioviren in vitro zusammen.[34]
- 2003 gelang Hamilton O. Smith und Craig Venter die Komplettsynthese des Bakteriophagen PhiX174 mit 5386 Basenpaaren.[35]
- 2005: Teilsynthese des Influenza-Virus durch die Arbeitsgruppe von Jeffery Taubenberger, welches um 1918 für die als Spanische Grippe bekannte Pandemie verantwortlich war.[36][37]
- Ende 2006 war es möglich, synthetische DNA bis zu etwa 35.000 Basenpaaren zu produzieren.[38] Obwohl die Größe lebensfähiger Minimalorganismen damals auf nur ca. 110.000 Basenpaare geschätzt wurde,[39] gelang es zunächst nicht, ein komplettes Prokaryoten-Genom (Bakterien-Genom) zu synthetisieren.
- 2010 gaben Forscher um Craig Venter die Herstellung des künstlichen Bakteriums Mycoplasma mycoides JCVI-syn1.0 bekannt. Zuvor hatten sie das 1,08 Millionen Basenpaare umfassende Erbgut eines Laborstammes von Mycoplasma mycoides aus chemischem Rohmaterial synthetisiert und in ein zuvor von der DNA befreites Bakterium von Mycoplasma capricolum übertragen.[14] Der Publikation in Science zufolge erwiesen sich die mit dem synthetischen Genom ausgestatteten Zellen als selbstreplizierend und zu exponentiellem Wachstum fähig.
- Im März 2016 stellte das Team am J. Craig Venter Institute im kalifornischen La Jolla die lebensfähige Version Syn 3.0 vor, die wiederum auf dem Bakterium Mycoplasma mycoides beruht. Die Bioingenieure verkürzten das ursprüngliche DNA-Molekül mit 1079 Kilobasen-Paaren (kbp) auf 531 kbp, damit verringerten sie das Gen-Repertoire um 57, nämlich auf 473 Gene.[40] Überraschenderweise blieb die zelluläre Funktion von 149 Genen des Minimalgenoms unklar. Bleibt also weiter zu fragen, was das Leben molekular ausmacht. Von diesem grundsätzlichen Problem unabhängig, wird sich ein solcher programmierbarer Minimalorganismus zur industriellen Herstellung von Biomolekülen einsetzen lassen.
- Im Mai 2019 berichteten Forscher über die Schaffung einer neuen synthetischen Variante Syn61 des Bakteriums Escherichia coli. Dieses unterscheidet sich von allen natürlichen Stämmen durch die Reduzierung der Anzahl von 64 Codons im Genom auf 61. Entfernt wurden bei der Unkodierung des 4 Megabasen großen Genoms zwei der sechs Codons für Serin und eines der drei Stopcodons. Zusätzlich zum Wegfall der Verwendung seltener Codons muss dabei die Spezifität des Systems erhöht werden, da viele tRNAs mehrere Codons erkennen.[41][42][43] Derartige synthetische Bakterien sind potentiell immun gegen jegliche Bakteriophagen.[44]
- Das Synthetic Yeast Project (Sc2.0) hat das Ziel, schrittweise eine Version der Backhefe (Bierhefe, Saccharomyces cerevisiae mit komplett künstlichem Genom herzustellen. Dieser Mikropliz wäre dann der erste Eukaryot, bei dem dies gelang. Im Herbst 2023 waren bereits über 50 % des Genoms ersetzt. Dabei wurden auch die Gene sämtlicher tRNAs in einem zusätzlichen künstlichen Chromosom zusammengefasst.[45][46][47]
Genom-Rekonstruktion
[Bearbeiten | Quelltext bearbeiten]Endy zerlegte mit seinem Team das Genom des Bakteriophagen T7 in seine bekannten funktionellen Einheiten, die stückweise wieder zusammengefügt wurden. Jede Kombination wurde auf ihre Funktion hin geprüft. Dadurch erhoffte man sich, die essentiellen Bestandteile von den überflüssigen, redundanten zu unterscheiden und damit zu erfahren, welches Minimalgenom für den Übergang von unbelebten, chemischen Systemen zu Lebewesen notwendig sind und damit Erkenntnisse über die frühe Evolution der Lebewesen zu erlangen.[4]
Durch Austausch der Gene für Rezeptor-bindende Proteine (RPB) von Bakteriophagen können diese Mutanten andere Bakterienwirte infizieren als die Wildtypen.[48]
Experimente in silico
[Bearbeiten | Quelltext bearbeiten]Kapazitäten und Rechnerleistungen sowie spezielle Algorithmen ermöglichen die Simulation komplexer biologischer Netzwerke im Computer. Dadurch werden die Methoden in vivo (im lebenden Organismus) und in vitro (im Reagenzglas) um die Möglichkeit in silico (wörtlich „im Silicium“) erweitert. Mit dem Computer können Selbstorganisationsprozesse, wie die Bildung von Membranen aus amphiphilen Molekülen in Wasser oder die Faltung von Proteinen, untersucht und mit den Beobachtungen an natürlichen Systemen verglichen werden. Ein weiteres Anwendungsgebiet ist die Erforschung der Bedeutung der Basensequenzen des menschlichen Genoms (siehe Humangenomprojekt). Dabei werden isolierte Basensequenzen der DNA mit Hilfe des Computers in Aminosäuresequenzen übersetzt. Diese werden mit Sequenzen bereits bekannter Proteine von Modellorganismen wie Taufliege (Drosophila melanogaster), Fadenwurm (Caenorhabditis elegans) oder Darmbakterium (Escherichia coli) verglichen. Anhand der gefundenen Eiweißstruktur wird im Computer das menschliche Proteinmolekül in seiner räumlichen Gestalt konstruiert und untersucht. So lassen sich zum Beispiel noch im Computer Wirkstoffe und Medikamente finden und konstruieren, welche die Funktion dieses Proteins beeinflussen können.
Einen großen Zuwachs an neuen Fragestellungen, Hypothesen und Erkenntnissen erwartet man sich von der Simulation regulatorischer Netzwerke einer Zelle im Computer. So ist es bereits gelungen, das Kammerflimmern des Herzens anhand von vier Genen, die dabei ihre Aktivität ändern, im Computer zu reproduzieren.
Fragen zur Biosicherheit, Ethik und zum geistigen Eigentum
[Bearbeiten | Quelltext bearbeiten]Neben den potenziell positiven Verwendungsmöglichkeiten der synthetischen Biologie müssen auch die potenziell negativen Auswirkungen berücksichtigt werden.[49][50] In den USA und teilweise in Großbritannien wird diesbezüglich vor allem das mögliche Missbrauchspotenzial (Dual Use) durch (Bio-)Terroristen diskutiert, was vor allem im Zusammenhang mit dem sogenannten Kampf gegen den Terror gesehen wird. Beispielsweise hat die Sequenzierung pathogener Viren (Spanische Grippe, Polio) zu einer heftigen Diskussion geführt,[51] ob und wie die Synthese pathogener Genome eingeschränkt werden kann, um einen Missbrauch zu verhindern. Warnungen einiger Wissenschaftler, NGOs und Journalisten[52] vor Bioterrorismus sorgten für eine erste Sensibilisierung einer breiteren Öffentlichkeit.[53] Eine von Gensynthese- und Bioinformatik-Firmen gegründete internationale Vereinigung, die International Association of Synthetic Biology, hat auf einem Workshop zu technischen Maßnahmen für die Gewährleistung der Biosicherheit in der synthetischen Biologie 2008 in München die Grundlage für einen Report mit einem detaillierten Maßnahmenkatalog gelegt.[54]
In Europa wird sich eine künftige Diskussion vermutlich weniger um Bioterroristen drehen[55] als um Fragen zu den unbeabsichtigten Auswirkungen auf die Gesundheit und Umwelt sowie um Fragen der Ethik und des geistigen Eigentums.[56] Vergangene und aktuelle öffentliche Debatten wie etwa zu gentechnisch verändertem Saatgut oder über Stammzellen zeigen, dass neue Technologien auch Konfliktpotenzial beinhalten können.
Vernetzung der synthetischen Biologie in Deutschland
[Bearbeiten | Quelltext bearbeiten]Die German Association for Synthetic Biology e. V. (GASB), der größte einschlägige Fachverband in Deutschland, widmet sich der Vernetzung von Wissenschaftlern und der allgemeinen Information über das Fachgebiet.[57][58] Neben wissenschaftlichen Konferenzen organisiert der Verband auch Veranstaltungen für die breite Öffentlichkeit.
Forschungsaktivitäten in Deutschland werden auch in der Studiengruppe 'Synthetische Biologie'[59] der Gesellschaft für Biochemie und Molekularbiologie (GBM) vorangetrieben, die den Austausch von Experten und Nachwuchswissenschaftlern ermöglicht.[60]
Siehe auch
[Bearbeiten | Quelltext bearbeiten]Literatur
[Bearbeiten | Quelltext bearbeiten]- Y. Ding, F. Wu, C. Tan: Synthetic Biology: A Bridge between Artificial and Natural Cells. In: Life. Band 4, Nr. 4, 2014, S. 1092–1116, doi:10.3390/life4041092, PMID 25532531, PMC 4284483 (freier Volltext) (Review).
- S. A. Benner, M. A. Sismour: Synthetic Biology. In: Nature Reviews Genetics. Band 6, 2005, S. 533–543 (siehe PDF)
- W. W. Gibbs: Künstliche Biomaschinen. In Spektrum der Wissenschaft Oktober 2004, S. 68 ff; ISSN 0170-2971
- K. Köchy, A. Hümpel (Hrsg.): Synthetische Biologie. Entwicklung einer neuen Ingenieurbiologie? Dornburg, 2012. ISBN 978-3-940647-07-8.
- M. Petter, in Laborjournal, Ausgabe 03, 2004 (siehe Stichwort – Synthetische Biologie)
- J. Schummer: Das Gotteshandwerk: Die künstliche Herstellung von Leben im Labor. Berlin: Suhrkamp (edition unseld), 2011, ISBN 978-3-518-26039-5.
- A. Pühler, B. Müller-Röber, M.-D. Weitze: Synthetische Biologie : die Geburt einer neuen Technikwissenschaft (Hrsg. im Auftrag der Deutschen Akademie der Technikwissenschaften (Acatech)), Berlin ; Heidelberg : Springer 2011, ISBN 978-3-642-22353-2 Seite der Acatech über den Band, mit Download-Möglichkeit
- D. Lanzerath, B. Giese, L. Jaeckel: Synthetische Biologie. Naturwissenschaftliche, rechtliche und ethische Aspekte, Freiburg, München: Verlag Karl Alber 2020, ISBN 978-3-495-49053-2.
- Hannah Depmeier and Stephanie Kath-Schorr: Expanding the Horizon of the Xeno Nucleic Acid Space: Threose Nucleic Acids with Increased Information Storage. In: ACS Publications: J. Am. Chem. Soc., 5. März 2024; doi:10.1021/jacs.3c14626 (englisch). Dazu:
- Forscherinnen entwickeln künstliche Bausteine des Lebens. Auf: EurekAlert! vom 8. März 2024. Quelle: Universität Köln.
- Unlocking TNA: Researchers Develop Artificial Building Blocks of Life. Auf: SciTechDaily vom 25. März 2024. Quelle: Universität Köln.
Presseartikel
[Bearbeiten | Quelltext bearbeiten]- Projekt Genesis. In: Die Zeit vom 16. Februar 2006.
- Terrorgefahr DNS-Synthese? In: Technology Review vom 14. Juli 2006.
- Organismen vom Reissbrett. In: Neue Zürcher Zeitung vom 23. August 2006.
- Synthetische Biologie: Baukasten für Biomaschinen. In: Spektrum der Wissenschaft vom 26. Januar 2007.
- „Bio-Ingenieure“ auf dem Vormarsch. In: APA-Zukunftswissen vom 31. Mai 2007 (PDF, 534 kB).
- Konkurrenz für Gott. In: Der Spiegel vom 4. Januar 2010.
- Künstliche Aminosäuren in Genom eingeschleust, auf: scinexx vom 10. Dezember 2004.
Weblinks
[Bearbeiten | Quelltext bearbeiten]- Synthetic genomics: options for governance (pdf; 1,68 MB)
- BioBricks-Datenbank
- Übersicht über SynBio-Labore in Deutschland
- Registry of Standard Biological Parts
- Seite der Max-Planck-Gesellschaft über Synthetische Biologie
- German Association for Synthetic Biology e. V.
- Expert Interviews Video-Interviews mit Experten und NGOs (englisch).
- Michael Le Page: Tiny axles and rotors made of protein could drive molecular machines – Researchers have designed proteins that self-assemble into tiny machine parts for use in molecular engines. Auf: New Scientist vom 21. April 2022 (englisch).
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ a b Synthetic Biology: Applying Engineering to Biology Report of a NEST High-Level Expert Group. Published by the European Commission 2005.
- ↑ L. Serrano: Synthetic biology: promises and challenges. In: Molecular Systems Biology. 3, 2007, S. 158 (synbiosafe.eu PDF; 113 kB).
- ↑ Technology Review. Juli 2006.
- ↑ a b c Drew Endy: Foundations for engineering biology. In: Nature. Band 438, Nr. 7067, 24. November 2005, S. 449–453, doi:10.1038/nature04342 (englisch).
- ↑ Nozomu Yachie et al.: Alignment-Based Approach for Durable Data Storage into Living Organisms. In: Biotechnology Progress. Band 23, Nr. 2, 2007, S. 501–505, doi:10.1021/bp060261y, yachie-lab.org (PDF, Volltext).
- ↑ Emil Fischer: Die Kaiser-Wilhelm-Institute und der Zusammenhang von organischer Chemie und Biologie. (1915). In: M. Bergmann (Hrsg.): Gesammelte Werke: Untersuchungen aus verschiedenen Gebieten: Vorträge und Abhandlungen allgemeinen Inhalts. J. Springer, Berlin 1924, S. 796–809.
- ↑ Nobelpreis für Medizin 1978. Auf: nobelprize.org
- ↑ Waclaw Szybalski, A. Skalka: Nobel prizes and restriction enzymes. In: Gene. Band 4, Nr. 3. Elsevier, November 1978, ISSN 0378-1119, S. 181 f., doi:10.1016/0378-1119(78)90016-1 (englisch).
- ↑ Barbara Hobom: Surgery of genes. At the doorstep of synthetic biology. In: Medizinische Klinik. Band 75, 1980, S. 14–21 (englisch).
- ↑ Rebecca L. Rawls: ‘Synthetic Biology’ makes its debut. In Chemical & Engineering News. Band 78, Nr. 17, 2000, S. 49–53; doi:10.1021/cen-v078n017.p049 (englisch).
- ↑ The Fifth International Meeting on Synthetic Biology. ( vom 1. Februar 2011 im Internet Archive)
- ↑ Peter Aldhous: Angry reception greets patent for synthetic life. In: New Scientist vom 16. Juni 2007, S. 13.
- ↑ Venter präsentiert künstliches Bakterien-Genom. In: Netzeitung. 24. Januar 2008, archiviert vom (nicht mehr online verfügbar) am 6. Januar 2010; abgerufen am 8. November 2019.
- ↑ a b Daniel G. Gibson et al.: Creation of a Bacterial Cell Controlled by a Chemically Synthesized Genome. In: Science, Band 329, Nr. 5987, 2010, S. 52–56, doi:10.1126/science.1190719 (englisch).
- ↑
Alan Costello, Alexander A. Peterson, David L. Lanster, Zhiyi Li, Gavriela D. Carver, Ahmed H. Badran: Efficient genetic code expansion without host genome modifications. In: Nature Biotechnology, 11. September 2024; doi:10.1038/s41587-024-02385-y (englisch). Dazu:
- Scripps Research scientists expand the genetic alphabet to create new proteins. Auf: EurekAlert! vom 11. September 2024.
- Elena Bernard: Erweiterter Gen-Code für künstliche Aminosäuren: Codons aus vier DNA-Basen ermöglichen Zellen den Einbau synthetischer Aminosäuren in Proteine. Auf: scinexx.de vom 24. September 2024.
- ↑ J. Christopher Anderson, Ning Wu, Stephen W. Santoro, Vishva Lakshman, David S. King, Peter G. Schultz: An expanded genetic code with a functional quadruplet codon. In: Proceedings of the National Academy of Sciences. Band 101, Nr. 20, 18. Mai 2004, S. 7566–7571, doi:10.1073/pnas.0401517101, PMID 15138302 (englisch).
- ↑ Takahiro Hohsaka, Masahiko Sisido: Incorporation of non-natural amino acids into proteins. In: Current Opinion in Chemical Biology. Band 6, Nr. 6, 1. Dezember 2002, S. 809–815, doi:10.1016/S1367-5931(02)00376-9 (englisch).
- ↑ Elie Dolgin: Der verschlungene Weg zum RNA-Impfstoff, Spektrum 3/2022 S. 28–37, Epub 16. Februar 2022.
- ↑ Christina Hohmann-Jeddi: Hoffnungsträger BNT162b2: Wie funktionieren mRNA-Impfstoffe? In: Pharmazeutische Zeitung. 10. November 2020, abgerufen am 28. November 2020.
- ↑ n-tv NACHRICHTEN: Paul-Ehrlich-Institut gibt Corona-Impfstoff frei. Abgerufen am 25. Dezember 2020.
- ↑ Annette B. Vogel, Isis Kanevsky, Ye Che, Kena A. Swanson, Alexander Muik: A prefusion SARS-CoV-2 spike RNA vaccine is highly immunogenic and prevents lung infection in non-human primates. In: bioRxiv. 8. September 2020, S. 2020.09.08.280818, doi:10.1101/2020.09.08.280818.
- ↑ COVID-19 Vaccine Moderna. (PDF) EMA - European Medicines Agency, archiviert vom (nicht mehr online verfügbar) am 14. Januar 2021; abgerufen am 9. Januar 2021 (englisch).
- ↑ Moderna's Pipeline. In: modernatx.com. Moderna, abgerufen am 28. November 2020 (englisch).
- ↑ Florian Krammer: SARS-CoV-2 vaccines in development. In: Nature. Band 586, Nr. 7830, Oktober 2020, ISSN 1476-4687, S. 516–527, doi:10.1038/s41586-020-2798-3.
- ↑ Kevin Michael Ulmer: Protein engineering. In: Science. Band 219, Nr. 4585, 1983, S. 666–671, doi:10.1126/science.6572017.
- ↑ Hans-Peter Kröner: Humangenetik. In: Werner E. Gerabek, Bernhard D. Haage, Gundolf Keil, Wolfgang Wegner (Hrsg.): Enzyklopädie Medizingeschichte. De Gruyter, Berlin 2005, ISBN 3-11-015714-4, S. 635–641, hier: S. 640 (Humangenetik in der molekularbiologischen Epoche).
- ↑ Perry L. Howard, Marie C. Chia, Suzanne Del Rizzo, Fei-Fei Liu und Tony Pawson: Redirecting tyrosine kinase signaling to an apoptotic caspase pathway through chieric adaptor proteins. In: PNAS. Band 100, Nr. 20, 2003, S. 11267–11272, doi:10.1073/pnas.1934711100.
- ↑ Deborah Halber: Teams lay BioBrick foundation for genetic engineering. Auf: MIT News, 8. November 2005 (englisch).
- ↑ Researchers develop an artificial chloroplast. Pressemeldung der Max-Planck-Gesellschaft. In: phys.org. 13. Juli 2021, abgerufen am 13. Juli 2021 (englisch).
- ↑ Tarryn E. Miller, Thomas Beneyton, Thomas Schwander, Christoph Diehl, Mathias Girault, Richard McLean, Tanguy Chotel, Peter Claus, Niña Socorro Cortina, Jean-Christophe Baret, Tobias J. Erb: Light-powered CO2 fixation in a chloroplast mimic with natural and synthetic parts. In: Science. 368. Jahrgang, Nr. 6491, 2020, S. 649–654, doi:10.1126/science.aaz6802, PMID 32381722.
- ↑ Synthetic red blood cells mimic natural ones, and have new abilities In: phys.org (englisch).
- ↑ Jimin Guo, Jacob Ongudi Agola, Rita Serda, Stefan Franco, Qi Lei, Lu Wang, Joshua Minster, Jonas G. Croissant, Kimberly S. Butler, Wei Zhu, C. Jeffrey Brinker: Biomimetic Rebuilding of Multifunctional Red Blood Cells: Modular Design Using Functional Components. In: ACS Nano. 14. Jahrgang, Nr. 7, 2020, S. 7847–7859, doi:10.1021/acsnano.9b08714, PMID 32391687.
- ↑ W. P. Stemmer, A. Crameri, K. D. Ha, T. M. Brennan, H. L. Heyneker: Single-step assembly of a gene and entire plasmid from large numbers of oligodeoxyribonucleotides. In: Gene. Band 164, Nr. 1, 1995, S. 49–53. PMID 7590320.
- ↑ Jeronimo Cello, Aniko V. Paul, Eckard Wimmer: Chemical Synthesis of Poliovirus cDNA: Generation of Infectious Virus in the Absence of Natural Template. In: Science. Band 297, 2002, S. 1016–1018, doi:10.1126/science.1072266
- ↑ H. O. Smith, C. A. 3rd Hutchison, C. Pfannkoch, J. C. Venter: Generating a synthetic genome by whole genome assembly: phiX174 bacteriophage from synthetic oligonucleotides. In: PNAS. Band 100, Nr. 26, 2003, S. 15440–5, doi:10.1073/pnas.2237126100. PMID 14657399; PMC 307586 (freier Volltext).
- ↑ Jeffery K. Taubenberger et al.: Characterization of the 1918 influenza virus polymerase genes. In: Nature. Band 437, 2005, S. 889–893, doi:10.1038/nature04230
- ↑ T. M. Tumpey, C. F. Basler, P. V. Aguilar, Zeng, H., Solórzano, A., Swayne, D.E., Cox, N.J., Katz, J.M., Taubenberger, J.K., Palese, P., and García-Sastre, A. (2005): Characterization of the reconstructed 1918 Spanish influenza pandemic virus. In: Science. Band 310, Nr. 5745, 2005, S. 77–80, doi:10.1126/science.1119392
- ↑ Matthew Herper: The Biggest DNA Ever Made. In: forbes.com. 13. Juli 2006, abgerufen am 12. Dezember 2019 (englisch).
- ↑ Philip Ball: Smallest genome clocks in at 182 genes. In: news@nature. 2006, doi:10.1038/news061009-10.
- ↑ Clyde A Hutchison III, Ray-Yuan Chuang, Vladimir N Noskov et al.: Design and synthesis of a minimal bacterial genome. In: Science. Band 351, 2016, doi:10.1126/science.aad6253 Freier Artikel.
- ↑ J. Fredens, K. Wang, D. de la Torre, L. F. Funke, W. E. Robertson, Y. Christova, T. Chia, W. H. Schmied, D. L. Dunkelmann, V. Beránek, C. Uttamapinant, A. G. Llamazares, T. S. Elliott, J. W. Chin: Total synthesis of Escherichia coli with a recoded genome. In: Nature. 569. Jahrgang, Nr. 7757, Mai 2019, S. 514–518, doi:10.1038/s41586-019-1192-5, PMID 31092918, PMC 7039709 (freier Volltext), bibcode:2019Natur.569..514F.
- ↑ Carl Zimmer: Scientists Created Bacteria With a Synthetic Genome. Is This Artificial Life? - In a milestone for synthetic biology, colonies of E. coli thrive with DNA constructed from scratch by humans, not nature. In: The New York Times, 15. Mai 2019
- Scientists Created Bacteria With a Synthetic Genome. Is This Artificial Life?, Richard Dawkins Foundation for Reason and Science, 22. Mai 2019.
- Scientists Created Bacteria With a Synthetic Genome. Is This Artificial Life?, auf: Big World Tale, 15. Mai 2019
- ↑ R. Krishnakumar, J. Ling: Experimental challenges of sense codon reassignment: an innovative approach to genetic code expansion. In: FEBS Letters. 588. Jahrgang, Nr. 3, Januar 2014, S. 383–388, doi:10.1016/j.febslet.2013.11.039, PMID 24333334 (englisch).
- ↑ Labor – Künstliche Zellen sind immun gegen Viren, auf: orf.at vom 4. Juni 2021.
- ↑
Katherine Bourzac: Engineered yeast breaks new record: a genome with over 50% synthetic DNA. Highly edited strain survives and replicates despite containing 7.5 artificial chromosomes. In: Nature, Band 623, News Nr. 469, 8. November 2023; doi:10.1038/d41586-023-03495-4 (englisch). Dazu:
- Geoffrey J. Taghon, Elizabeth A. Strychalski: Rise of synthetic yeast: Charting courses to new applications. In: Cell Genomics, Bend 3, Nr. 11, 8. November 2023; S. 100438; doi:10.1016/j.xgen.2023.100438 (englisch).
- Erster Eukaryot mit halbsynthetischem Genom. Forschungsteam erzeugt Hefezellen mit siebeneinhalb künstlich im Labor hergestellten Chromosomen. Auf: scinexx.de vom 9. November 2023.
- ↑
Daniel Schindler, Roy S. K. Walker, Shuangying Jiang; Lars M. Steinmetz, Jef D. Boeke, Yizhi Cai et al.: Design, construction, and functional characterization of a tRNA neochromosome in yeast. In: Cell, 8. November 2023; doi:10.1016/j.cell.2023.10.015, ResearchGate, ScienceDirect (englisch). Dazu:
- Ordnung für die Bierhefe: Ein künstliches Chromosom vereint alle t-RNA-Gene für die Produktion von Proteinen. Presseveröffentlichung der Max-Planck-Gesellschaft vom 10. November 2023.
- Kay Sanders: Eine neue Ordnung für die Bierhefe: Max-Planck-Institut für terrestrische Mikrobiologie. Auf: scinexx.de vom 14. November 2023.
- ↑ Yu Zhao, Camila Coelho, Amanda L. Hughes; Lars M. Steinmetz, Yizhi Cai, Jef D. Boeke et al.: Debugging and consolidating multiple synthetic chromosomes reveals combinatorial genetic interactions. In: Cell, 8. November 2023; doi:10.1016/j.cell.2023.09.025 (englisch).
- ↑ Matthew Dunne, Nikolai S. Prokhorov, Martin J. Loessner, Petr G. Leiman: Reprogramming bacteriophage host range: design principles and strategies for engineering receptor binding proteins. In: Current Opinion in Biotechnology, Band 68, April 2021, S. 272–281; doi:10.1016/j.copbio.2021.02.006. (englisch).
- ↑ Bhutkar, A. 2005. Synthetic Biology: Navigating the Challenges Ahead. J. BIOLAW & BUS., Band 8, Nr. 2, S. 19–29.
- ↑ Frontiers 2018/19: Emerging Issues of Environmental Concern. In: unenvironment.org. 4. März 2019, abgerufen am 3. Januar 2021 (englisch).
- ↑ P. A. Sharp: 1918 Flu and Responsible Science. In: Science. Band 310, Nr. 5745, 2005, S. 17, doi:10.1126/science.310.5745.17.
- ↑ z. B. in The Guardian ( vom 14. Juni 2006 im Internet Archive)
- ↑ Newspaper investigation highlights bioterror fears. In: Nature News. 2006, doi:10.1038/news060612-5 (nature.com).
- ↑ Report der IASB Technical solutions for biosecurity in synthetic biology. (PDF; 381 kB), München, April 2008.
- ↑ Synthetic Biology & Biosecurity Awareness In Europe (PDF; 408 kB).
- ↑ Markus Schmidt: Public will fear biological accidents, not just attacks. In: Nature. Band 441, Nr. 7097, 2006, S. 1048, doi:10.1038/4411048d.
- ↑ A platform for Synthetic Biology in Germany. Abgerufen am 8. Dezember 2019.
- ↑ Helene Richter: Nachrichten des Verbandes Biologie, Biowissenschaften & Biomedizin in Deutschland. In: Biologie in unserer Zeit. Band 48, Nr. 1, 2018, ISSN 1521-415X, S. 23–26, doi:10.1002/biuz.201870111.
- ↑ Homepage der Studiengruppe Synthetische Biologie der GBM Auf: gbm-online.de.
- ↑ Vorstellung – GBM-Studiengruppe „Synthetische Biologie“. In: BIOspektrum. 24. Jahrgang. Springer, 2018, ISSN 0947-0867, S. 732 f. (biospektrum.de [PDF; 6,5 MB; abgerufen am 10. Dezember 2019]).