„SARS-CoV-2“ – Versionsunterschied
| [gesichtete Version] | [gesichtete Version] |
K Änderungen von 46.245.148.225 (Diskussion) auf die letzte Version von Ernsts zurückgesetzt Markierung: Zurücksetzung |
Quelle WHCV (biorxiv) geprüft, dort ist nicht die Bezeichnung WH-Human-1 coronavirus zu finden; Artikel nun in Nature veröffentlicht, weitere Infos daraus (ELISA) erg.; Infos zum ACE2-Rezeptor und dem damit verbunden S-Protein erg.; Inkubationszeit, Antikörpernachweis und Anteil bestimmter Bevölkerungsgruppen erg.; weitere Kleinigkeiten; Belege im Artikeltext |
||
| Zeile 32: | Zeile 32: | ||
== Benennung == |
== Benennung == |
||
Die vom 13. Januar 2020 bis zum 11. Februar 2020 von der WHO verwendete Bezeichnung „2019-nCoV“ galt laut WHO als „vorläufig“.<ref>{{Webarchiv |url=https://www.who.int/emergencies/diseases/novel-coronavirus-2019 |wayback=20200128165840 |text=''Novel Coronavirus (2019-nCoV).''}}</ref> Das erste [[DNA-Sequenzierung|sequenzierte]] Virusisolat wurde in der Erstbeschreibung als ''WH-Human-1 coronavirus (WHCV)'' bezeichnet (WH = Wuhan)< |
Die vom 13. Januar 2020 bis zum 11. Februar 2020 von der WHO verwendete Bezeichnung „2019-nCoV“ galt laut WHO als „vorläufig“.<ref>{{Webarchiv |url=https://www.who.int/emergencies/diseases/novel-coronavirus-2019 |wayback=20200128165840 |text=''Novel Coronavirus (2019-nCoV).''}}</ref> Das erste [[DNA-Sequenzierung|sequenzierte]] Virusisolat wurde in der Erstbeschreibung als ''WH-Human-1 coronavirus (WHCV)'' bezeichnet (WH = Wuhan)<--! Quelle WHCV (biorxiv) geprüft, im Artikel ist nicht die Bezeichnung WH-Human-1 coronavirus zu finden --> und als ''Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1'' in die NCBI [[Taxonomie]]-Datenbank (die für Virusnamen und -klassifikationen nicht maßgebend ist) aufgenommen. Das Virus wurde dort danach – ebenfalls vorläufig – als ''Wuhan seafood market pneumonia virus'' geführt; als [[Synonym (Taxonomie)|Synonyme]] wurden ''2019-nCoV'' und ''Wuhan coronavirus'' erwähnt.<ref>{{Webarchiv |url=https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=2697049 |wayback=20200203071823 |text=Taxonomy ID: 2697049 ''Wuhan seafood market pneumonia virus.''}}</ref> Namensvorschläge, das Virus in Anlehnung an [[MERS-CoV]] (''Middle East respiratory syndrome coronavirus'') nach dem Ort seiner Erstidentifikation, der Stadt Wuhan, als ''Wuhan respiratory syndrome coronavirus'' (WRS-CoV) zu benennen, wurden von der WHO nicht aufgegriffen. Ein von Fachleuten genannter Grund waren Beschwerden in der Vergangenheit, als Viren ihren Namen nach einzelnen Ländern oder Regionen erhalten hatten<ref>{{Internetquelle |autor=Ching-Tse Cheng |url=https://www.taiwannews.com.tw/en/news/3856862 |titel=''WHO declines to name new pneumonia after 'China' or 'Wuhan'.'' |hrsg=Taiwan News |datum=2020-01-14 |sprache=en |abruf=2020-01-14}}</ref> (beispielsweise das [[Marburg-Virus]]). Die WHO hatte daher 2015 Empfehlungen herausgegeben, wie neue Krankheitserreger und Erkrankungen zu benennen seien. Eine Benennung nach dem Entdeckungsort wurde dabei explizit für unerwünscht erklärt.<ref>{{Internetquelle |url=https://www.who.int/mediacentre/news/notes/2015/naming-new-diseases/en/ |titel=WHO issues best practices for naming new human infectious diseases |hrsg=Weltgesundheitsorganisation |datum=2015-05-08 |sprache=en |abruf=2020-02-06}}</ref> In der NCBI Taxonomie-Datenbank aufgeführte Synonyme sind ''2019-nCoV'', ''COVID-19'', ''COVID-19 virus'', ''Wuhan coronavirus'' und ''Wuhan seafood market pneumonia virus'' (Stand 16. Februar 2020).<ref name="taxo">{{Internetquelle |url=https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=2697049 |titel=Taxonomy Browser Severe acute respiratory syndrome coronavirus 2, Taxonomy ID: 2697049 |werk=Website [[National Center for Biotechnology Information]] (NCBI) |sprache=en |abruf=2020-02-16}}</ref> |
||
Am 11. Februar 2020 gab die WHO bekannt, die durch das Virus verursachte Erkrankung als „COVID-19“ bzw. „Covid-19“ (''Corona virus disease 2019'') benannt zu haben.<ref name="WHO_report_22" /><ref name="Fischer_Spektrum_20200210" /> Am selben Tag wurde von der ''Coronavirus Study Group'' (CSG) des ''[[International Committee on Taxonomy of Viruses]]'' (ICTV) auf dem Preprint-Server ''[[bioRxiv]]'' für das Virus die Bezeichnung SARS-CoV-2 (''Severe Acute Respiratory Syndrome Coronavirus 2'') vorgeschlagen.<ref name="CSG20200211" /> Dem widersprach eine Woche später eine Gruppe chinesischer Virologen, die stattdessen als Bezeichnung „Humanes Coronavirus 2019“ (''human coronavirus 2019'', HCoV-19) vorschlug. Als Begründung nannten sie die Ähnlichkeit dieser Virusbezeichnung mit der Bezeichnung der von der WHO als COVID-19 benannten Krankheit und die Gefahr der Verwechslung von SARS-CoV-2 mit SARS-CoV in der Öffentlichkeit. Sie betonten, dass sich „2019-nCoV“ von dem SARS-Virus in biologischer und epidemiologischer Hinsicht unterscheidet, ebenso wie die klinischen Symptome von COVID-19 und SARS verschieden sind.<ref name="Guo_Lancet_20200219">{{Literatur |Autor=Shibo Jiang, Zhengli Shi, Yuelong Shu, Jingdong Song, George F. Gao, Wenjie Tan, Deyin Guo |Titel=A distinct name is needed for the new coronavirus |Sammelwerk=[[The Lancet]] |Band= |Datum=2020-02-19 |Sprache=en |DOI=10.1016/S0140-6736(20)30419-0}}</ref> Zur Unterscheidung wird der Erreger von SARS auch als SARS-CoV-1 bezeichnet.<ref name="armbaut2019">Kristian G. Andersen, Andrew Rambaut, W. Ian Lipkin, Edward C. Holmes, Robert F. Garry: [http://virological.org/t/the-proximal-origin-of-sars-cov-2/398 The Proximal Origin of SARS-CoV-2], auf: virologica.org, Quelle: ARTIC Network, 17. Februar 2020</ref> |
Am 11. Februar 2020 gab die WHO bekannt, die durch das Virus verursachte Erkrankung als „COVID-19“ bzw. „Covid-19“ (''Corona virus disease 2019'') benannt zu haben.<ref name="WHO_report_22" /><ref name="Fischer_Spektrum_20200210" /> Am selben Tag wurde von der ''Coronavirus Study Group'' (CSG) des ''[[International Committee on Taxonomy of Viruses]]'' (ICTV) auf dem Preprint-Server ''[[bioRxiv]]'' für das Virus die Bezeichnung SARS-CoV-2 (''Severe Acute Respiratory Syndrome Coronavirus 2'') vorgeschlagen.<ref name="CSG20200211" /> Dem widersprach eine Woche später eine Gruppe chinesischer Virologen, die stattdessen als Bezeichnung „Humanes Coronavirus 2019“ (''human coronavirus 2019'', HCoV-19) vorschlug. Als Begründung nannten sie die Ähnlichkeit dieser Virusbezeichnung mit der Bezeichnung der von der WHO als COVID-19 benannten Krankheit und die Gefahr der Verwechslung von SARS-CoV-2 mit SARS-CoV in der Öffentlichkeit. Sie betonten, dass sich „2019-nCoV“ von dem SARS-Virus in biologischer und epidemiologischer Hinsicht unterscheidet, ebenso wie die klinischen Symptome von COVID-19 und SARS verschieden sind.<ref name="Guo_Lancet_20200219">{{Literatur |Autor=Shibo Jiang, Zhengli Shi, Yuelong Shu, Jingdong Song, George F. Gao, Wenjie Tan, Deyin Guo |Titel=A distinct name is needed for the new coronavirus |Sammelwerk=[[The Lancet]] |Band= |Datum=2020-02-19 |Sprache=en |DOI=10.1016/S0140-6736(20)30419-0}}</ref> Zur Unterscheidung wird der Erreger von SARS auch als SARS-CoV-1 bezeichnet.<ref name="armbaut2019">Kristian G. Andersen, Andrew Rambaut, W. Ian Lipkin, Edward C. Holmes, Robert F. Garry: [http://virological.org/t/the-proximal-origin-of-sars-cov-2/398 The Proximal Origin of SARS-CoV-2], auf: virologica.org, Quelle: ARTIC Network, 17. Februar 2020</ref> |
||
| Zeile 40: | Zeile 40: | ||
[[Datei:SARS-CoV-2 genome.svg|mini|Putative Genomorganisation von SARS-CoV-2 ([[Offener Leserahmen|offene Leserahmen]])]] |
[[Datei:SARS-CoV-2 genome.svg|mini|Putative Genomorganisation von SARS-CoV-2 ([[Offener Leserahmen|offene Leserahmen]])]] |
||
Das Virusgenom besteht, wie in Coronaviren üblich, aus einzelsträngiger RNA ([[ssRNA]]) mit [[Polarität (Virologie)|positiver Polarität]]. Das Isolat Wuhan-Hu-1 (NCBI GenBank-Nummer MN908947<ref name="nucleotide" />) umfasst 29.903 nt ([[Nukleotide]]) mit zwei 265 nt bzw. 229 nt langen [[Untranslatierter Bereich|untranslatierten Bereichen]] am [[Nukleinsäure-Nomenklatur|5′-Ende]] bzw. am 3′-Ende.<ref name="MN908947" /> Die [[Vermutetes Gen|putativen (vermuteten) Gene]] könnten für zehn [[Protein]]e [[Genetischer Code|codieren]]: ein 7096 [[Aminosäuren]] (AS) langes [[Offener Leserahmen|ORF]]1ab-[[Polyprotein]] (Replikase-Komplex), ein 1273 AS langes Oberflächen-[[Glykoprotein]] (S für englisch ''spikes'', vergleiche [[Peplomer]]), ein 75 AS langes [[Hüllprotein]] (E für engl. ''envelope'', vergleiche [[Virushülle]]), ein 222 AS langes [[Membranprotein|Membran-Glykoprotein]] (M), ein 419 AS langes [[Kapsid#Kapsid und Nukleokapsid|Nukleokapsid]]-[[Phosphoprotein]] (N) und weitere fünf Proteine (ORF3a, ORF6, ORF7a, ORF8 und ORF10).<ref name="MN908947" /> |
Das Virusgenom besteht, wie in Coronaviren üblich, aus einzelsträngiger RNA ([[ssRNA]]) mit [[Polarität (Virologie)|positiver Polarität]]. Das Isolat Wuhan-Hu-1 (NCBI GenBank-Nummer MN908947<ref name="nucleotide" />) umfasst 29.903 nt ([[Nukleotide]]) mit zwei 265 nt bzw. 229 nt langen [[Untranslatierter Bereich|untranslatierten Bereichen]] am [[Nukleinsäure-Nomenklatur|5′-Ende]] bzw. am 3′-Ende.<ref name="MN908947" /> Die [[Vermutetes Gen|putativen (vermuteten) Gene]] könnten für zehn [[Protein]]e [[Genetischer Code|codieren]]: ein 7096 [[Aminosäuren]] (AS) langes [[Offener Leserahmen|ORF]]1ab-[[Polyprotein]] (Replikase-Komplex), ein 1273 AS langes Oberflächen-[[Glykoprotein]] (S für englisch ''spikes'', vergleiche [[Peplomer]]), ein 75 AS langes [[Hüllprotein]] (E für engl. ''envelope'', vergleiche [[Virushülle]]), ein 222 AS langes [[Membranprotein|Membran-Glykoprotein]] (M), ein 419 AS langes [[Kapsid#Kapsid und Nukleokapsid|Nukleokapsid]]-[[Phosphoprotein]] (N) und weitere fünf Proteine (ORF3a, ORF6, ORF7a, ORF8 und ORF10).<ref name="MN908947" /><!-- Infos zu den Proteinen sind auf der als Quelle genannten Website verlinkt --> Die Abfolge der Gene entspricht jener des [[SARS-assoziiertes Coronavirus|SARS-Virus]] und der aller Coronaviren.<ref>{{Literatur |Autor=Matthew Frieman, Ralph Baric |Titel=Mechanisms of Severe Acute Respiratory Syndrome Pathogenesis and Innate Immunomodulation |Sammelwerk=[[Microbiology and Molecular Microbiology Reviews]] |Band=Band 72 |Datum=2008-12 |Seiten=672–685 |Sprache=en |Kommentar=open access |DOI=10.1128/MMBR.00015-08 |PMID=19052324}}</ref> |
||
Mittlerweile (Stand 16. Februar 2020) gibt es mehr als 40 vollständige Genomanalysen von SARS-CoV-2-Isolaten. Die Genomgröße liegt zwischen 29.825 und 29.903 nt.<ref name="nucleotide" /> Der [[GC-Gehalt]] (der Anteil der [[Nukleinbasen]] [[Guanin]] und [[Cytosin]]) liegt bei 38,0 Mol-Prozent.<ref name="genome">{{Internetquelle |url=https://www.ncbi.nlm.nih.gov/genome/?term=txid2697049 |titel=Wuhan seafood market pneumonia virus |werk=Website Genome des [[National Center for Biotechnology Information]] (NCBI) |abruf=2020-02-16}}</ref><ref name="Chan_Lancet_20200124" /> Die beiden Virusisolate HKU-SZ-002a (NCBI GenBank-Nummer MN938384<ref name="nucleotide" />) und HKU-SZ-005b (NCBI GenBank-Nummer MN975262<ref name="nucleotide" />) stammen von Patienten einer Familie aus [[Shenzhen]] und unterscheiden sich lediglich durch zwei Nukleotide. Die Genomanalyse dieser beiden Isolate ergab, dass sie nahe verwandt mit den bei [[Fledermäuse]]n (englisch ''bat'') auftretenden SARS-ähnlichen Coronaviren bat-SL-CoVZXC21 (NCBI GenBank-Nummer MG772934) und bat-SL-CoVZC45 (NCBI GenBank-Nummer MG772933) sind, zu letzterem besteht eine Übereinstimmung in der Nukleotidabfolge von 89 %. Das Genom der beiden Fledermaus-Coronaviren wurde 2018 sequenziert, bat-SL-CoVZC45 wurde bei der [[Chinesische Hufeisennase|Chinesischen Hufeisennase]] (''Rhinolophus sinicus'') aus der Familie der [[Hufeisennasen]] (Rhinolophidae) gefunden, die Wirtstiere wurden in [[Zhoushan]] in der ostchinesischen Provinz [[Zhejiang]] in den Jahren 2015 und 2017 untersucht.<ref name="Chan_Lancet_20200124" /> |
Mittlerweile (Stand 16. Februar 2020) gibt es mehr als 40 vollständige Genomanalysen von SARS-CoV-2-Isolaten. Die Genomgröße liegt zwischen 29.825 und 29.903 nt.<ref name="nucleotide" /> Der [[GC-Gehalt]] (der Anteil der [[Nukleinbasen]] [[Guanin]] und [[Cytosin]]) liegt bei 38,0 Mol-Prozent.<ref name="genome">{{Internetquelle |url=https://www.ncbi.nlm.nih.gov/genome/?term=txid2697049 |titel=Wuhan seafood market pneumonia virus |werk=Website Genome des [[National Center for Biotechnology Information]] (NCBI) |abruf=2020-02-16}}</ref><ref name="Chan_Lancet_20200124" /> Die beiden Virusisolate HKU-SZ-002a (NCBI GenBank-Nummer MN938384<ref name="nucleotide" />) und HKU-SZ-005b (NCBI GenBank-Nummer MN975262<ref name="nucleotide" />) stammen von Patienten einer Familie aus [[Shenzhen]] und unterscheiden sich lediglich durch zwei Nukleotide. Die Genomanalyse dieser beiden Isolate ergab, dass sie nahe verwandt mit den bei [[Fledermäuse]]n (englisch ''bat'') auftretenden SARS-ähnlichen Coronaviren bat-SL-CoVZXC21 (NCBI GenBank-Nummer MG772934) und bat-SL-CoVZC45 (NCBI GenBank-Nummer MG772933) sind, zu letzterem besteht eine Übereinstimmung in der Nukleotidabfolge von 89 %. Das Genom der beiden Fledermaus-Coronaviren wurde 2018 sequenziert, bat-SL-CoVZC45 wurde bei der [[Chinesische Hufeisennase|Chinesischen Hufeisennase]] (''Rhinolophus sinicus'') aus der Familie der [[Hufeisennasen]] (Rhinolophidae) gefunden, die Wirtstiere wurden in [[Zhoushan]] in der ostchinesischen Provinz [[Zhejiang]] in den Jahren 2015 und 2017 untersucht.<ref name="Chan_Lancet_20200124" /> |
||
Ein weiteres Virusisolat (WIV04, NCBI GenBank-Nummer MN996528<ref name="nucleotide" />) von SARS-CoV-2 aus der [[Bronchoalveoläre Lavage|bronchoalveolären Spülflüssigkeit]] eines der ersten Patienten zeigt ebenfalls [[Phylogenese|phylogenetisch]] größte Ähnlichkeit mit |
Ein weiteres Virusisolat (WIV04, NCBI GenBank-Nummer MN996528<ref name="nucleotide" />) von SARS-CoV-2 aus der [[Bronchoalveoläre Lavage|bronchoalveolären Spülflüssigkeit]] eines der ersten Patienten zeigt ebenfalls [[Phylogenese|phylogenetisch]] größte Ähnlichkeit mit einem bei Fledermäusen (''[[Rhinolophus affinis]]'') in China isolierten Coronavirus BatCoV RaTG13, die Genomsequenzen stimmen zu 96,2 % überein.<ref name="Zhou_Nature_20200203" /> Auch eine am 27. Januar 2020 publizierte genetische Analyse verwies auf Fledermäuse als mutmaßlicher Ursprungswirt des Virus.<ref>{{Literatur |Autor=D. Paraskevis, E. G. Kostaki, G. Magiorkinis, G. Panayiotakopoulos, G. Sourvinos, S. Tsiodras |Titel=Full-genome evolutionary analysis of the novel corona virus (2019-nCoV) rejects the hypothesis of emergence as a result of a recent recombination event |Sammelwerk=bioRxiv |Datum=2020-01-27 |Sprache=en |DOI=10.1101/2020.01.26.920249}}</ref> Am 29. Januar 2020 wurde in der Fachzeitschrift ''[[The Lancet]]'' eine genetische Analyse von zehn Virusproben publiziert, die bei neun Erkrankten gewonnen worden waren. Demnach war die Genomsequenz aller zehn Proben zu 99,98 Prozent identisch, was darauf hinweist, dass die neu entdeckte Coronavirusvariante erst vor Kurzem auf den Menschen übergegangen ist.<ref name="Lu_Lancet_20200129" /><ref name="DEAEB_20200130">{{Internetquelle |url=https://www.aerzteblatt.de/nachrichten/109051/Genom-Analysen-klaeren-Herkunft-von-2019-nCoV |titel=Genom-Analysen klären Herkunft von 2019-nCoV |werk=Website [[Deutsches Ärzteblatt]] |datum=2020-01-30 |abruf=2020-02-09}}</ref><ref name="DEAEB_20200127">{{Internetquelle |url=https://www.aerzteblatt.de/nachrichten/108909/2019-nCoV-Erste-Bilder-vom-Virus-und-Erkenntnisse-zum-klinischen-Verlauf |titel=2019-nCoV: Erste Bilder vom Virus und Erkenntnisse zum klinischen Verlauf |werk=Website [[Deutsches Ärzteblatt]] |datum=2020-01-27 |abruf=2020-02-11}}</ref> Die Genomsequenz stimmt zu 88 bzw. 87 % Prozent mit den Genomsequenzen der bei Fledermäusen auftretenden bat-SL-CoVZC45 und bat-SL-CoVZXC21 überein. Die zehn Proben zeigen hingegen nur rund 79 Prozent Übereinstimmung in der Genomsequenz zu [[SARS-assoziiertes Coronavirus|SARS-CoV]] und rund 50 Prozent zu [[MERS-CoV]]. Die Ergebnisse der phylogenetischen Untersuchungen werden auch als [[phylogenetischer Baum]] veranschaulicht.<ref name="Chan_Lancet_20200124" /><ref name="Lu_Lancet_20200129" /> Eine darauf basierende Darstellung ist im Artikel ''[[Betacoronavirus]]'' zu finden. |
||
Der Aufbau des Genoms sowohl der SARS-CoV-2-Isolate wie auch der genannten Fledermaus-Coronaviren ist typisch für Viren der ''Lineage B'' (Untergattung ''[[Sarbecovirus]]'', {{enS|SARS-like Betacoronavirus}}) der Gattung ''Betacoronavirus''. Aufgrund der genetischen Distanzen zu SARS-CoV und zu MERS-CoV wurde SARS-CoV-2 zunächst als eine in Bezug auf den Menschen neue, ihn infizierende ''Betacoronavirus''-Spezies angesehen.<ref name="Chan_Lancet_20200124" /><ref name="Lu_Lancet_20200129" /> Aufgrund der großen genetischen Übereinstimmung mit dem ursprünglichen ''SARS-Coronavirus'' hatte am 11. Februar 2020 die ''Coronavirus Study Group'' des ICTV jedoch vorgeschlagen, das neue Virus derselben Spezies ''Severe acute respiratory syndrome-related coronavirus'' zuzuordnen wie das bisherige.<ref name="CSG20200211" /> |
Der Aufbau des Genoms sowohl der SARS-CoV-2-Isolate wie auch der genannten Fledermaus-Coronaviren ist typisch für Viren der ''Lineage B'' (Untergattung ''[[Sarbecovirus]]'', {{enS|SARS-like Betacoronavirus}}) der Gattung ''Betacoronavirus''. Aufgrund der genetischen Distanzen zu SARS-CoV und zu MERS-CoV wurde SARS-CoV-2 zunächst als eine in Bezug auf den Menschen neue, ihn infizierende ''Betacoronavirus''-Spezies angesehen.<ref name="Chan_Lancet_20200124" /><ref name="Lu_Lancet_20200129" /> Aufgrund der großen genetischen Übereinstimmung mit dem ursprünglichen ''SARS-Coronavirus'' hatte am 11. Februar 2020 die ''Coronavirus Study Group'' des ICTV jedoch vorgeschlagen, das neue Virus derselben Spezies ''Severe acute respiratory syndrome-related coronavirus'' zuzuordnen wie das bisherige.<ref name="CSG20200211" /> |
||
Das S-Protein (S für englisch ''spikes'') ist für die [[Viren#Vermehrung und Verbreitung|Bindung an die Wirtszelle]] verantwortlich, funktionell wird es in die S1-Domäne und die S2-Domäne unterschieden. Die S1-Domäne vermittelt die Bindung an den [[Rezeptor (Biochemie)|Oberflächenrezeptor]] der Wirtszelle, die S2-Domäne vermittelt die Fusion der [[Zellmembran]], durch [[Endozytose]] erfolgt dann der Eintritt des Virus in die Zelle. Das S-Gen von SARS-CoV-2 zeigt mit 75 % eine eher geringe Übereinstimmung in der Nukleotidsequenz mit den beiden Fledermausisolaten bat-SL-CoVZC45 und bat-SL-CoVZXC21 im Vergleich zur Genomanalyse. Insbesondere die Nukleotidsequenz, die für die S1-Domäne codiert, unterscheidet sich von diesen deutlich (68 % Übereinstimmung) und weist eine größere Ähnlichkeit mit der entsprechenden Nukleotidsequenz von SARS-CoV auf. Daraus lässt sich schließen, dass SARS-CoV und das neuartige Coronavirus den gleichen Zellrezeptor nutzen, das [[Angiotensin-konvertierendes Enzym 2|Angiotensin-konvertierende Enzym 2]] (ACE2).<ref name="Lu_Lancet_20200129" /> Dies konnte experimentell bewiesen werden, verleiche [[#Pathogenese|Abschnitt Pathogenese]]. |
|||
=== Morphologie === |
=== Morphologie === |
||
| Zeile 58: | Zeile 60: | ||
== Virulenz und Pathogenese == |
== Virulenz und Pathogenese == |
||
=== Risikogruppe === |
=== Risikogruppe === |
||
Für Beschäftigte, die durch ihre berufliche Tätigkeit mit Infektionserregern in Kontakt kommen können, gilt in Deutschland die [[Biostoffverordnung]] |
Für Beschäftigte, die durch ihre berufliche Tätigkeit mit Infektionserregern in Kontakt kommen können, gilt in Deutschland die [[Biostoffverordnung]] (BioStoffV). Der bei der [[Bundesanstalt für Arbeitsschutz und Arbeitsmedizin]] (BAuA) eingerichtete Ausschuss für [[Biologische Arbeitsstoffe]] (ABAS) hat SARS-CoV-2 am 19. Februar 2020 vorläufig in die [[Biologische Schutzstufe#Risikogruppe|Risikogruppe]] 3 nach der BioStoffV eingeordnet.<ref name="abas_info" /> Grundsätzlich erfolgt die Einstufung in Risikogruppen in den Technischen Regeln für Biostoffe (TRBA), die von der BAuA veröffentlicht werden, für Viren ist dies die ''TRBA 462: Einstufung von Viren in Risikogruppen''. Beim Auftreten neuartiger, noch nicht zugeordneter Krankheitserreger erfolgt zunächst eine vorläufige Einstufung durch den ABAS. In der Begründung wird auf die Ähnlichkeit von SARS-CoV-2 mit dem SARS-CoV-1 hingewiesen, der die SARS-Epidemie 2002/2003 ausgelöst hat und auch die Ähnlichkeit in geringerem Umfang mit dem MERS-CoV wird erwähnt. Diese beiden Viren wurden ebenfalls der Risikogruppe 3 zugeordnet. Der ABAS nennt die „derzeit fehlenden Möglichkeiten zu Impfprävention und Therapie sowie die großen Verbreitungsmöglichkeit in der Bevölkerung“ als Begründung für die vorläufige Zuordnung zur Risikogruppe 3.<ref name="abas_einstuf" /> |
||
Außerdem werden Empfehlungen zur Arbeit mit dem Virus bei der Diagnostik im Labor gegeben: Nicht gezielte Tätigkeiten (vergleiche {{§|5|biostoffv_2013|juris|text=§ 5 BioStoffV}}) – ausgehend vom Untersuchungsmaterial, also beispielsweise die Probenvorbereitung, Probenaufbereitung und die Inaktivierung, um den Nachweis mittels RT-PCR (siehe [[#Nachweismethoden|Abschnitt Nachweismethoden]]) durchzuführen – können unter den Bedingungen der [[Biologische Schutzstufe|Schutzstufe]] 2 durchgeführt werden. Dabei sind alle Tätigkeiten, bei denen mit [[Aerosol]]bildung zu rechnen ist, in einer mikrobiologischen [[Sicherheitswerkbank]] der Klasse II durchzuführen. Außerdem ist die entsprechende [[persönliche Schutzausrüstung]] zu tragen. Gezielte Tätigkeiten nach § 5 BioStoffV dürfen nur in Laboratorien der Schutzstufe 3 durchgeführt werden, dies betrifft z. B. die Vermehrung des Virus in einer [[Zellkultur]].<ref name="abas_einstuf" /> Die US-amerikanische Seuchenkontrollbehörde ''[[Centers for Disease Control and Prevention]]'' (CDC) hatte zuvor ähnliche Empfehlungen herausgegeben.<ref name="CDC_4" /> |
Außerdem werden Empfehlungen zur Arbeit mit dem Virus bei der Diagnostik im Labor gegeben: Nicht gezielte Tätigkeiten (vergleiche {{§|5|biostoffv_2013|juris|text=§ 5 BioStoffV}}) – ausgehend vom Untersuchungsmaterial, also beispielsweise die Probenvorbereitung, Probenaufbereitung und die Inaktivierung, um den Nachweis mittels RT-PCR (siehe [[#Nachweismethoden|Abschnitt Nachweismethoden]]) durchzuführen – können unter den Bedingungen der [[Biologische Schutzstufe|Schutzstufe]] 2 durchgeführt werden. Dabei sind alle Tätigkeiten, bei denen mit [[Aerosol]]bildung zu rechnen ist, in einer mikrobiologischen [[Sicherheitswerkbank]] der Klasse II durchzuführen. Außerdem ist die entsprechende [[persönliche Schutzausrüstung]] zu tragen. Gezielte Tätigkeiten nach § 5 BioStoffV dürfen nur in Laboratorien der Schutzstufe 3 durchgeführt werden, dies betrifft z. B. die Vermehrung des Virus in einer [[Zellkultur]].<ref name="abas_einstuf" /> Die US-amerikanische Seuchenkontrollbehörde ''[[Centers for Disease Control and Prevention]]'' (CDC) hatte zuvor ähnliche Empfehlungen herausgegeben.<ref name="CDC_4" /> |
||
| Zeile 70: | Zeile 72: | ||
=== Basisreproduktionszahl === |
=== Basisreproduktionszahl === |
||
Die Auswertung der Daten der ersten 425 Fälle in Wuhan ergab eine [[Basisreproduktionszahl]] von 2,2.<ref name="Li_NEJM_20200129">{{Literatur |Autor=Li Q, Guan X, Wu P, Wang X, Zhou L, Tong Y, Ren R, Leung KSM, Lau EHY, Wong JY, Xing X, Xiang N, Wu Y, Li C, Chen Q, Li D, Liu T, Zhao J, Li M, Tu W, Chen C, Jin L, Yang R, Wang Q, Zhou S, Wang R, Liu H, Luo Y, Liu Y, Shao G, Li H, Tao Z, Yang Y, Deng Z, Liu B, Ma Z, Zhang Y, Shi G, Lam TTY, Wu JTK, Gao GF, Cowling BJ, Yang B, Leung GM, Feng Z. |Titel=Early Transmission Dynamics in Wuhan, China, of Novel Coronavirus–Infected Pneumonia |Sammelwerk=[[The New England Journal of Medicine]] |Datum=2020-01-29 |Sprache=en |DOI=10.1056/NEJMoa2001316}}</ref> Eine Modellrechnung mit chinesischen und ausländischen Patientendaten vom 31. Dezember 2019 bis zum 28. Januar 2020 ergab einen Wert von 2,68.<ref name="Wu_Lancet_20200131">{{Literatur |Autor=Joseph T. Wu, Kathy Leung, Gabriel M. Leung |Titel=Nowcasting and forecasting the potential domestic and international spread of the 2019-nCoV outbreak originating in Wuhan, China: a modelling study |Sammelwerk=[[The Lancet]] |Datum=2020-01-31 |Sprache=en |DOI=10.1016/S0140-6736(20)30260-9}}</ref> Im Vergleich hierzu wurde für SARS eine Basisreproduktionsrate von 2,3 bis 2,6 berechnet.<ref name="Liu_bioRxiv_20200213">{{Literatur |Autor=Tao Liu, Jianxiong Hu, Jianpeng Xiao, Guanhao He, Min Kang, Zuhua Rong, Lifeng Lin, Haojie Zhong, Qiong Huang, Aiping Deng, Weilin Zeng, Xiaohua Tan, Siqing Zeng, Zhihua Zhu, Jiansen Li, Dexin Gong, Donghua Wan, Shaowei Chen, Lingchuan Guo, Yan Li, Limei Sun, Wenjia Liang, Tie Song, Jianfeng He, Wenjun Ma |Titel=Time-varying transmission dynamics of Novel Coronavirus Pneumonia in China |Sammelwerk=[[bioRxiv]] |Datum=2020-02-13 |Sprache=en |DOI=10.1101/2020.01.25.919787}}</ref> Eine vergleichende Auswertung von 12 Studien, die bis zum 7. Februar 2020 veröffentlicht wurden, kommt zu dem Ergebnis, dass die Basisreproduktionszahl höher liegt, als bisher von der WHO angenommen, deren Schätzung bei 1,4 bis 2,5 liegt. Die Wissenschaftler aus Schweden, China und Deutschland ermitteln, dass die Basisreproduktionszahl im [[Mittelwert|Mittel]] bei 3,28, im [[Median]] bei 2,79 liegt und somit höher als der Wert bei SARS ist, den sie mit 2 bis 5 angeben.<ref name="Rocklöv_Lancet_20200131">{{Literatur |Autor=Ying Liu, Albert A. Gayle, Annelies Wilder-Smith, Joacim Rocklöv |Titel=The reproductive number of COVID-19 is higher compared to SARS coronavirus |Sammelwerk=[[Journal of Travel Medicine]] |Datum=2020-02-13 |Sprache=en |DOI=10.1093/jtm/taaa021 |Seiten=taaa021}}</ref> |
Die Auswertung der Daten der ersten 425 Fälle in Wuhan ergab eine [[Basisreproduktionszahl]] von 2,2.<ref name="Li_NEJM_20200129">{{Literatur |Autor=Li Q, Guan X, Wu P, Wang X, Zhou L, Tong Y, Ren R, Leung KSM, Lau EHY, Wong JY, Xing X, Xiang N, Wu Y, Li C, Chen Q, Li D, Liu T, Zhao J, Li M, Tu W, Chen C, Jin L, Yang R, Wang Q, Zhou S, Wang R, Liu H, Luo Y, Liu Y, Shao G, Li H, Tao Z, Yang Y, Deng Z, Liu B, Ma Z, Zhang Y, Shi G, Lam TTY, Wu JTK, Gao GF, Cowling BJ, Yang B, Leung GM, Feng Z. |Titel=Early Transmission Dynamics in Wuhan, China, of Novel Coronavirus–Infected Pneumonia |Sammelwerk=[[The New England Journal of Medicine]] |Datum=2020-01-29 |Sprache=en |DOI=10.1056/NEJMoa2001316}}</ref> Eine Modellrechnung mit chinesischen und ausländischen Patientendaten vom 31. Dezember 2019 bis zum 28. Januar 2020 ergab einen Wert von 2,68.<ref name="Wu_Lancet_20200131">{{Literatur |Autor=Joseph T. Wu, Kathy Leung, Gabriel M. Leung |Titel=Nowcasting and forecasting the potential domestic and international spread of the 2019-nCoV outbreak originating in Wuhan, China: a modelling study |Sammelwerk=[[The Lancet]] |Datum=2020-01-31 |Sprache=en |DOI=10.1016/S0140-6736(20)30260-9}}</ref> Im Vergleich hierzu wurde für SARS eine Basisreproduktionsrate von 2,3 bis 2,6 berechnet.<ref name="Liu_bioRxiv_20200213">{{Literatur |Autor=Tao Liu, Jianxiong Hu, Jianpeng Xiao, Guanhao He, Min Kang, Zuhua Rong, Lifeng Lin, Haojie Zhong, Qiong Huang, Aiping Deng, Weilin Zeng, Xiaohua Tan, Siqing Zeng, Zhihua Zhu, Jiansen Li, Dexin Gong, Donghua Wan, Shaowei Chen, Lingchuan Guo, Yan Li, Limei Sun, Wenjia Liang, Tie Song, Jianfeng He, Wenjun Ma |Titel=Time-varying transmission dynamics of Novel Coronavirus Pneumonia in China |Sammelwerk=[[bioRxiv]] |Datum=2020-02-13 |Sprache=en |DOI=10.1101/2020.01.25.919787}}</ref> Eine vergleichende Auswertung von 12 Studien, die bis zum 7. Februar 2020 veröffentlicht wurden, kommt zu dem Ergebnis, dass die Basisreproduktionszahl höher liegt, als bisher von der WHO angenommen, deren Schätzung bei 1,4 bis 2,5 liegt.<ref name="Cheng_Infection_20200218" /> Die Wissenschaftler aus Schweden, China und Deutschland ermitteln, dass die Basisreproduktionszahl im [[Mittelwert|Mittel]] bei 3,28, im [[Median]] bei 2,79 liegt und somit höher als der Wert bei SARS ist, den sie mit 2 bis 5 angeben.<ref name="Rocklöv_Lancet_20200131">{{Literatur |Autor=Ying Liu, Albert A. Gayle, Annelies Wilder-Smith, Joacim Rocklöv |Titel=The reproductive number of COVID-19 is higher compared to SARS coronavirus |Sammelwerk=[[Journal of Travel Medicine]] |Datum=2020-02-13 |Sprache=en |DOI=10.1093/jtm/taaa021 |Seiten=taaa021}}</ref> |
||
=== Anteil bestimmter Bevölkerungsgruppen === |
|||
Eine Mitte Februar 2020 veröffentliche Auswertung der englischsprachigen und chinesischen Fachartikel kommt zu dem Ergebnis, dass alle Bevölkerungsgruppen infiziert werden können. Von den Infizierten waren 72 % 40 Jahre alt oder älter, 64 % waren männlich. 40 % der Patienten hatten [[Krankheitsverlauf|chronische Erkrankungen]] wie [[Diabetes mellitus]] und [[Arterielle Hypertonie|Bluthochdruck]].<ref name="Cheng_Infection_20200218" /> |
|||
=== Anteil schwerer Verläufe und Sterblichkeit === |
=== Anteil schwerer Verläufe und Sterblichkeit === |
||
| Zeile 82: | Zeile 87: | ||
=== Inkubationszeit === |
=== Inkubationszeit === |
||
Die [[Inkubationszeit]] kann laut Informationen des Robert Koch |
Die [[Inkubationszeit]] kann laut Informationen des [[Robert Koch-Institut]]s (RKI) bis zu 14 Tage betragen.<ref name="RKI_FAQ" /> Darüber hinaus gibt es Berichte chinesischer Forscher, welche die mögliche Inkubationszeit auf bis zu 24 Tage ausdehnen.<ref name="Bai_JAMA_20200221" /> Eine Analyse der ersten 425 in Wuhan gemeldeten Fälle ergibt eine Inkubationszeit von im Mittel 5,2 Tagen und ein Durchschnittsalter von 59 Jahren. Die Autoren gehen davon aus, dass bereits Mitte Dezember 2019 im Umfeld des Fischmarktes Übertragungen von Mensch zu Mensch stattfanden.<ref name="Li_NEJM_20200129" /> Eine statistische Auswertung mehrerer Berichte von Infektionen in einem Haushalt oder in anderer enger räumlicher Begrenzung (sogenannte Cluster) ergibt eine Inkubationszeit von im [[Median]] 5 – 6 Tagen.<ref name="WHO_report_30" /> |
||
Eine Ansteckung anderer Menschen während der Inkubationszeit ist trotz beschwerdefreien Gesundheitszustands möglich. Tests auf die [[Viruslast]] im Blut bei einzelnen Patienten legen den Verdacht nahe, dass manche Patienten auch während der Ausheilung bei klinischer Besserung weiterhin vorübergehend infektiös sein können.<ref name="Rothe_NEJM_20200130" /> |
Eine Ansteckung anderer Menschen während der Inkubationszeit ist trotz beschwerdefreien Gesundheitszustands möglich. Tests auf die [[Viruslast]] im Blut bei einzelnen Patienten legen den Verdacht nahe, dass manche Patienten auch während der Ausheilung bei klinischer Besserung weiterhin vorübergehend infektiös sein können.<ref name="Rothe_NEJM_20200130" /> Der Bericht dieser Publikation, welcher auf der Annahme einer asymptomatischen chinesischen Indexpatientin beruhen, wurde durch die Recherche der Fachzeitschrift ''[[Science]]'' widerlegt und vom RKI in Zweifel gezogen.<ref name="Kupferschmidt_Science_20200203">{{Literatur |Autor=Kai Kupferschmidt |Titel=Study claiming new coronavirus can be transmitted by people without symptoms was flawed |Sammelwerk=[[Science]] |Datum=2020-02-03 |Sprache=en |Online=https://www.sciencemag.org/news/2020/02/paper-non-symptomatic-patient-transmitting-coronavirus-wrong}}</ref> In einer Gruppe von 126 aus Wuhan nach Deutschland Evakuierten zeigten sich zwei Patienten in der RT-PCR des Rachenabstrichs positiv, welche keine oder nur sehr unspezifische Beschwerden aufwiesen.<ref>Sandra Ciesek et al.: ''Evidence of SARS-CoV-2 Infection in Returning Travelers from Wuhan, China.'' NEJM, 18. Februar 2018 [[doi:10.1056/NEJMc2001899]]</ref> Ebenso ist ein Fall eines subjektiv asymptomatischen zehnjährigen Jungen in [[Shenzhen]] beschrieben, dessen Blutbild und Entzündungszeichen im Labor unauffällig waren. In der weiteren Untersuchung zeigten sich jedoch radiologische Befunde vereinbar mit einer Pneumonie, und im Rachenabstrich ließ sich Virus-RNA nachweisen.<ref name="Chan_Lancet_20200124" /> |
||
Aufgrund von quantitativen Virusuntersuchungen im Sekret des Nasenrachenraums bei Patienten mit sehr leichten [[Symptom]]en schlossen die Forscher der [[Virologie]] der [[Charité]] und des [[Institut für Mikrobiologie der Bundeswehr|Instituts für Mikrobiologie der Bundeswehr]], dass auch bereits bei sehr milden Erkrankungssymptomen eine hohe Infektionsfähigkeit besteht.<ref name="DEAEB_20200204_1">{{Internetquelle |url=https://www.aerzteblatt.de/nachrichten/109159/2019-nCoV-offenbar-schon-bei-sehr-leichten-Symptomen-uebertragbar |titel=2019-nCoV offenbar schon bei sehr leichten Symptomen übertragbar |werk=Website [[Deutsches Ärzteblatt]] |datum=2020-02-04 |abruf=2020-02-05}}</ref><ref name="Fischer_Spektrum_20200210" /> Auch das |
Darüber hinaus existiert ein weiterer Fallbericht aus [[Guangzhou]] von zwei asymptomatisch Infizierten mit Virusnachweis im Nasenrachenraum. Die Autoren wiesen explizit auf die Verbreitungsgefahr des Virus durch beschwerdefreie Patienten in frühen Infektionsstadien hin.<ref>Jing Liu : ''Asymptomatic cases in a family cluster with SARS-CoV-2 infection.'' Lancet, 19. Februar 2020, [[doi:10.1016/S1473-3099(20)30114-6]]</ref> Messungen der Viruslast im Sekret des Nasenrachenraums ergeben eine ähnlich hohe Viruslast zwischen beschwerdefreien und symptomatisch kranken Patienten.<ref name="Zou_NEJM_20200219" /> Aufgrund von quantitativen Virusuntersuchungen im Sekret des Nasenrachenraums bei Patienten mit sehr leichten [[Symptom]]en schlossen die Forscher der [[Virologie]] der [[Charité]] und des [[Institut für Mikrobiologie der Bundeswehr|Instituts für Mikrobiologie der Bundeswehr]], dass auch bereits bei sehr milden Erkrankungssymptomen eine hohe Infektionsfähigkeit besteht.<ref name="DEAEB_20200204_1">{{Internetquelle |url=https://www.aerzteblatt.de/nachrichten/109159/2019-nCoV-offenbar-schon-bei-sehr-leichten-Symptomen-uebertragbar |titel=2019-nCoV offenbar schon bei sehr leichten Symptomen übertragbar |werk=Website [[Deutsches Ärzteblatt]] |datum=2020-02-04 |abruf=2020-02-05}}</ref><ref name="Fischer_Spektrum_20200210" /> Auch das Robert Koch-Institut berichtet über einzelne Fälle, bei denen sich Betroffene möglicherweise bei infizierten Personen angesteckt haben, die noch keine oder keine spezifischen Symptome gezeigt hatten.<ref name="RKI_FAQ" /> Zum gleichen Ergebnis kommt eine chinesische Fallstudie, die sechs Patienten einer Familie betrachtet. Patientin 1 hat ihre fünf Verwandten mit COVID-19 angesteckt, ohne selber Symptome zu zeigen, wegen der Krankheitsfälle wurde auch sie isoliert und ärztlich überwacht. Der Virusnachweis durch RT-PCR bei ihr war nach 17 Tagen negativ, nach 19 Tagen positiv und nach 25 bzw. 30 Tagen wieder negativ.<ref name="Bai_JAMA_20200221" /> |
||
=== Pathogenese === |
=== Pathogenese === |
||
Das Virus dringt wie bei SARS über den [[Angiotensin-konvertierendes Enzym 2|ACE2]]-Rezeptor in die menschliche Zelle ein.<ref name="Zhou_Nature_20200203" /> Im Versuch mit [[HeLa-Zellen]], die ACE2 des Menschen, der Chinesischen Hufeisennase (''Rhinolophus sinicus''), einer [[Schleichkatzen|Schleichkatzenart]] (engl. ''civet''), des [[Hausschwein]]s und der [[Mäuse|Maus]] [[Genexpression|exprimieren]], konnte SARS-CoV-2 das jeweilige ACE2-Protein als Rezeptor nutzen, um in die Zelle einzudringen, nur bei dem Maus-ACE2 gelang dies nicht, ebensowenig bei HeLa-Zellen, die kein ACE2 bildeten. An Rezeptoren, die von anderen Coronaviren genutzt werden, findet keine Bindung von SARS-CoV-2 statt.<ref name="Zhou_Nature_20200203" /> |
|||
Das Virus dringt wie bei SARS über den [[Angiotensin-konvertierendes Enzym 2|ACE2]]-Rezeptor in die menschliche Zelle ein.<ref name="WHCV" /> |
|||
In einem Informationsblatt des deutschen [[Auswärtiges Amt|Außenministeriums]] wird erwähnt, dass aus China Berichte „von [[Infektionskette]]n über die 4. Generationen hinaus“ vorliegen.<ref>{{Internetquelle |url=https://www.auswaertiges-amt.de/blob/2294930/c4ff6708cd7c37bac1cb198f53400b56/ncov-data.pdf |titel=Gesundheitsdienst, Stand 27. Januar 2020 |hrsg=Auswärtiges Amt |format=PDF |abruf=2020-01-27}}</ref> |
In einem Informationsblatt des deutschen [[Auswärtiges Amt|Außenministeriums]] wird erwähnt, dass aus China Berichte „von [[Infektionskette]]n über die 4. Generationen hinaus“ vorliegen.<ref>{{Internetquelle |url=https://www.auswaertiges-amt.de/blob/2294930/c4ff6708cd7c37bac1cb198f53400b56/ncov-data.pdf |titel=Gesundheitsdienst, Stand 27. Januar 2020 |hrsg=Auswärtiges Amt |format=PDF |abruf=2020-01-27}}</ref> |
||
| Zeile 136: | Zeile 141: | ||
==== Antikörpernachweis ==== |
==== Antikörpernachweis ==== |
||
Auch der [[Immunmarkierung|Antikörpernachweis]] als [[Serologie|serologische]] Untersuchung wird nach Angabe der WHO derzeit entwickelt (Stand: 17. Januar 2020). Dadurch wird es ein Assay (beispielsweise ein [[Immunassay]] wie [[Enzyme-linked Immunosorbent Assay|ELISA]]) geben, mit dem [[Antikörper]] aus Patienproben ([[Blutserum]]) durch [[Antigen-Antikörper-Reaktion]] nachweisbar sind. Die WHO und das Robert Koch-Institut (RKI) in Deutschland rufen dazu auf, Serumproben von bestätigten oder Verdachtsfällen in der Akutphase zu sammeln und zu [[Asservat|asservieren]].<ref name="RKI_20200123" /> Die WHO empfiehlt, die erste Probe in der ersten Krankheitswoche und die zweite Probe drei bis vier Wochen später zu nehmen. Damit lässt sich eine [[Serokonversion]] überprüfen.<ref name="who_lab_testing" /><ref name="RKI_20200205">{{Internetquelle |url=https://www.rki.de/DE/Content/InfAZ/N/Neuartiges_Coronavirus/Vorl_Testung_nCoV.html |titel=Hinweise zur Testung von Patienten auf Infektion mit dem in Wuhan entdeckten neuartigen Coronavirus (2019-nCoV) |werk=Website des [[Robert Koch-Institut]]s (RKI) |datum=2020-02-03 |abruf=2020-02-05}}</ref> |
Auch der [[Immunmarkierung|Antikörpernachweis]] als [[Serologie|serologische]] Untersuchung wird nach Angabe der WHO derzeit entwickelt (Stand: 17. Januar 2020). Dadurch wird es ein Assay (beispielsweise ein [[Immunassay]] wie [[Enzyme-linked Immunosorbent Assay|ELISA]]) geben, mit dem [[Antikörper]] aus Patienproben ([[Blutserum]]) durch [[Antigen-Antikörper-Reaktion]] nachweisbar sind. Die WHO und das Robert Koch-Institut (RKI) in Deutschland rufen dazu auf, Serumproben von bestätigten oder Verdachtsfällen in der Akutphase zu sammeln und zu [[Asservat|asservieren]].<ref name="RKI_20200123" /> Die WHO empfiehlt, die erste Probe in der ersten Krankheitswoche und die zweite Probe drei bis vier Wochen später zu nehmen. Damit lässt sich eine [[Serokonversion]] überprüfen.<ref name="who_lab_testing" /><ref name="RKI_20200205">{{Internetquelle |url=https://www.rki.de/DE/Content/InfAZ/N/Neuartiges_Coronavirus/Vorl_Testung_nCoV.html |titel=Hinweise zur Testung von Patienten auf Infektion mit dem in Wuhan entdeckten neuartigen Coronavirus (2019-nCoV) |werk=Website des [[Robert Koch-Institut]]s (RKI) |datum=2020-02-03 |abruf=2020-02-05}}</ref> |
||
In einem chinesischen Forschungslabor wurden im Januar 2020 erste ELISA-Tests durchgeführt, als Antigen wurde das Nukleokapsidprotein (N) eines Fledermaus-Coronavirus mit Ähnlichkeit zu SARS-CoV-2 verwendet. Damit ließen sich in Serumproben eines Patienten die Antikörper [[Immunglobulin G]] (IgG) und [[Immunglobulin M]] (IgM) nachweisen und deren [[Titer (Medizin)|Titer]] über mehrere Tage während des Krankheitsverlaufes bestimmen. In einem zweiten Test wurden Serumproben, die 20 Tage nach den ersten Symptomen entnommen wurden, untersucht. Alle Patientenseren, aber nicht die Seren von Gesunden zeigten eine stark positive IgG-Reaktion, einige Patientenseren zeigten zusätzlich eine IgM-Reaktion, was auf eine aktuelle Immunantwort, also eine momentane Infektion hindeutet.<ref name="Zhou_Nature_20200203" /> |
|||
=== Zellkultur === |
=== Zellkultur === |
||
Die Vermehrung des Virus zu Forschungszwecken in einer [[Zellkultur]] ist unter anderem in [[China]], [[Australien]], [[Frankreich]], [[Deutschland]] und den [[Vereinigte Staaten|USA]] gelungen.<ref name="Zhu_NEJM_20200124" /><ref name="the-scientist_20200129">{{Internetquelle |autor=Jef Akst |url=https://www.the-scientist.com/news-opinion/australian-lab-cultures-new-coronavirus-as-infections-climb-67031 |titel=Australian Lab Cultures New Coronavirus as Infections Climb |werk=Website The-Scientist |datum=2020-01-29 |sprache=en |abruf=2020-02-04}}</ref><ref>{{Internetquelle |url=https://eurekalert.org/pub_releases/2020-01/ip-ipi013120.php |titel=Institut Pasteur isolates strains of coronavirus 2019-nCoV detected in France |werk=Website EurekAlert! |datum=2020-01-31 |sprache=en |abruf=2020-02-04}}</ref><ref>{{Internetquelle |url=https://instmikrobiobw.de/aktuelles/ansicht/erstmals-ncov-in-deutschland-in-zellkultur-isoliert |titel=Erstmals nCoV in Deutschland in Zellkultur isoliert |werk=Website [[Institut für Mikrobiologie der Bundeswehr]] |datum=2020-01-31 |abruf=2020-02-03}}</ref><ref name="CDC_3" /> Die chinesischen Wissenschaftler verwenden hierbei [[Epithel]]zellen des menschlichen [[Atemtrakt]]s, die das mehrschichtige mukoziliäres Epithelgewebe ([[Flimmerepithel]]) simulieren, ebenso werden die [[Zellkultur#Zellkultur-Linien|Zelllinien]] Vero E6 und Huh-7 eingesetzt.<ref name="Zhu_NEJM_20200124" /> |
Die Vermehrung des Virus zu Forschungszwecken in einer [[Zellkultur]] ist unter anderem in [[China]], [[Australien]], [[Frankreich]], [[Deutschland]] und den [[Vereinigte Staaten|USA]] gelungen.<ref name="Zhu_NEJM_20200124" /><ref name="the-scientist_20200129">{{Internetquelle |autor=Jef Akst |url=https://www.the-scientist.com/news-opinion/australian-lab-cultures-new-coronavirus-as-infections-climb-67031 |titel=Australian Lab Cultures New Coronavirus as Infections Climb |werk=Website The-Scientist |datum=2020-01-29 |sprache=en |abruf=2020-02-04}}</ref><ref>{{Internetquelle |url=https://eurekalert.org/pub_releases/2020-01/ip-ipi013120.php |titel=Institut Pasteur isolates strains of coronavirus 2019-nCoV detected in France |werk=Website EurekAlert! |datum=2020-01-31 |sprache=en |abruf=2020-02-04}}</ref><ref>{{Internetquelle |url=https://instmikrobiobw.de/aktuelles/ansicht/erstmals-ncov-in-deutschland-in-zellkultur-isoliert |titel=Erstmals nCoV in Deutschland in Zellkultur isoliert |werk=Website [[Institut für Mikrobiologie der Bundeswehr]] |datum=2020-01-31 |abruf=2020-02-03}}</ref><ref name="CDC_3" /> Die chinesischen Wissenschaftler verwenden hierbei [[Epithel]]zellen des menschlichen [[Atemtrakt]]s, die das mehrschichtige mukoziliäres Epithelgewebe ([[Flimmerepithel]]) simulieren, ebenso werden die [[Zellkultur#Zellkultur-Linien|Zelllinien]] Vero E6 und Huh-7 eingesetzt.<ref name="Zhu_NEJM_20200124" /><ref name="Zhou_Nature_20200203" /> |
||
== Behandlung == |
== Behandlung == |
||
| Zeile 178: | Zeile 185: | ||
Am 27. Januar 2020 wurden erstmals auch in Deutschland Infektionen nachgewiesen.<ref name="stmgp_20200127">{{Internetquelle |url=https://www.stmgp.bayern.de/presse/bestaetigter-coronavirus-fall-in-bayern-infektionsschutzmassnahmen-laufen/ |titel=Pressemitteilung: Bestätigter Coronavirus-Fall in Bayern – Infektionsschutzmaßnahmen laufen |werk=Website [[Bayerisches Staatsministerium für Gesundheit und Pflege]] |datum=2020-01-27 |abruf=2020-02-09}}</ref> Zunächst bei einem 33-jährigen Mann aus dem [[Landkreis Landsberg am Lech]], der von einer Chinesin – der [[Indexpatient]]in – infiziert worden war, die sich vom 19. bis 22. Januar auf Dienstreise in dem Unternehmen [[Webasto|Webasto SE]] in Bayern aufgehalten hatte. Zunächst hatte es geheißen, bei ihr seien Anzeichen für eine Infektion erst während des Rückflugs nach China aufgetreten;<ref name="Rothe_NEJM_20200130">{{Literatur |Autor=Camilla Rothe, Mirjam Schunk, Peter Sothmann, Gisela Bretzel, Guenter Froeschl, Claudia Wallrauch, Thorbjörn Zimmer, Verena Thiel, Christian Janke, Wolfgang Guggemos, Michael Seilmaier, Christian Drosten, Patrick Vollmar, Katrin Zwirglmaier, Sabine Zange, Roman Wölfel, Michael Hoelscher |Titel=Transmission of 2019-nCoV Infection from an Asymptomatic Contact in Germany |Sammelwerk=[[The New England Journal of Medicine]] |Datum=2020-01-30 |Sprache=en |DOI=10.1056/NEJMc2001468}}</ref> tatsächlich hatte sie aber bereits zuvor aufgrund erster Symptome fiebersenkende Mittel eingenommen.<ref name="Kupferschmidt_Science_20200203" /> Bereits am Abend des 28. Januar wurde bekannt gegeben, dass sich auch drei Kollegen des 33-Jährigen mit SARS-CoV-2 infiziert hatten.<ref name="tagesschau_20200129">{{Internetquelle |url=https://www.tagesschau.de/inland/coronavirus-149.html |titel=Coronavirus: Drei weitere Fälle in Bayern |werk=Website [[tagesschau.de]] |datum=2020-01-29 |abruf=2020-02-09}}</ref> An den folgenden Tagen wurde das Virus bei mehreren Kontaktpersonen der Indexpatientin und bei Angehörigen der in Bayern infizierten Personen nachgewiesen, darunter ein fünfjähriges Kind.<ref name="tagesschau_20200131">{{Internetquelle |url=https://www.tagesschau.de/coronavirus-deutschland-115.html |titel=Sechste Infektion bestätigt: Kind in Bayern an Coronavirus erkrankt |werk=Website [[tagesschau.de]] |datum=2020-01-31 |abruf=2020-02-09}}</ref><ref name="stmgp_20200201">{{Internetquelle |url=https://www.stmgp.bayern.de/presse/aktuelle-informationen-zur-coronavirus-lage-in-bayern-bayerisches-gesundheitsministerium-7/ |titel=Pressemitteilung: 7. Fall in München Klinik Schwabing in stabilem Zustand |werk=Website [[Bayerisches Staatsministerium für Gesundheit und Pflege]] |datum=2020-01-27 |abruf=2020-02-09}}</ref> |
Am 27. Januar 2020 wurden erstmals auch in Deutschland Infektionen nachgewiesen.<ref name="stmgp_20200127">{{Internetquelle |url=https://www.stmgp.bayern.de/presse/bestaetigter-coronavirus-fall-in-bayern-infektionsschutzmassnahmen-laufen/ |titel=Pressemitteilung: Bestätigter Coronavirus-Fall in Bayern – Infektionsschutzmaßnahmen laufen |werk=Website [[Bayerisches Staatsministerium für Gesundheit und Pflege]] |datum=2020-01-27 |abruf=2020-02-09}}</ref> Zunächst bei einem 33-jährigen Mann aus dem [[Landkreis Landsberg am Lech]], der von einer Chinesin – der [[Indexpatient]]in – infiziert worden war, die sich vom 19. bis 22. Januar auf Dienstreise in dem Unternehmen [[Webasto|Webasto SE]] in Bayern aufgehalten hatte. Zunächst hatte es geheißen, bei ihr seien Anzeichen für eine Infektion erst während des Rückflugs nach China aufgetreten;<ref name="Rothe_NEJM_20200130">{{Literatur |Autor=Camilla Rothe, Mirjam Schunk, Peter Sothmann, Gisela Bretzel, Guenter Froeschl, Claudia Wallrauch, Thorbjörn Zimmer, Verena Thiel, Christian Janke, Wolfgang Guggemos, Michael Seilmaier, Christian Drosten, Patrick Vollmar, Katrin Zwirglmaier, Sabine Zange, Roman Wölfel, Michael Hoelscher |Titel=Transmission of 2019-nCoV Infection from an Asymptomatic Contact in Germany |Sammelwerk=[[The New England Journal of Medicine]] |Datum=2020-01-30 |Sprache=en |DOI=10.1056/NEJMc2001468}}</ref> tatsächlich hatte sie aber bereits zuvor aufgrund erster Symptome fiebersenkende Mittel eingenommen.<ref name="Kupferschmidt_Science_20200203" /> Bereits am Abend des 28. Januar wurde bekannt gegeben, dass sich auch drei Kollegen des 33-Jährigen mit SARS-CoV-2 infiziert hatten.<ref name="tagesschau_20200129">{{Internetquelle |url=https://www.tagesschau.de/inland/coronavirus-149.html |titel=Coronavirus: Drei weitere Fälle in Bayern |werk=Website [[tagesschau.de]] |datum=2020-01-29 |abruf=2020-02-09}}</ref> An den folgenden Tagen wurde das Virus bei mehreren Kontaktpersonen der Indexpatientin und bei Angehörigen der in Bayern infizierten Personen nachgewiesen, darunter ein fünfjähriges Kind.<ref name="tagesschau_20200131">{{Internetquelle |url=https://www.tagesschau.de/coronavirus-deutschland-115.html |titel=Sechste Infektion bestätigt: Kind in Bayern an Coronavirus erkrankt |werk=Website [[tagesschau.de]] |datum=2020-01-31 |abruf=2020-02-09}}</ref><ref name="stmgp_20200201">{{Internetquelle |url=https://www.stmgp.bayern.de/presse/aktuelle-informationen-zur-coronavirus-lage-in-bayern-bayerisches-gesundheitsministerium-7/ |titel=Pressemitteilung: 7. Fall in München Klinik Schwabing in stabilem Zustand |werk=Website [[Bayerisches Staatsministerium für Gesundheit und Pflege]] |datum=2020-01-27 |abruf=2020-02-09}}</ref> |
||
Am Abend des 30. Januar 2020 erklärte die [[Weltgesundheitsorganisation]] (WHO) die Epidemie zu einer [[Gesundheitliche Notlage internationaler Tragweite|Gesundheitlichen Notlage internationaler Tragweite]].<ref name="SZ_20200130">{{Internetquelle |url=https://www.sueddeutsche.de/gesundheit/coronavirus-who-notlage-1.4778794 |titel=WHO ruft wegen Coronavirus weltweite Notlage aus |hrsg=[[Süddeutsche Zeitung]] |datum=2020-01-30 |abruf=2020-01-30}}</ref> |
Am Abend des 30. Januar 2020 erklärte die [[Weltgesundheitsorganisation]] (WHO) die Epidemie zu einer [[Gesundheitliche Notlage internationaler Tragweite|Gesundheitlichen Notlage internationaler Tragweite]].<ref name="SZ_20200130">{{Internetquelle |url=https://www.sueddeutsche.de/gesundheit/coronavirus-who-notlage-1.4778794 |titel=WHO ruft wegen Coronavirus weltweite Notlage aus |hrsg=[[Süddeutsche Zeitung]] |datum=2020-01-30 |abruf=2020-01-30}}</ref> Am 9. Februar 2020 überstieg die Zahl der registrierten Todesfälle mit über 800 die Gesamtzahl der Todesfälle bei der [[SARS-Pandemie 2002/2003]].<ref name="Cheng_Infection_20200218" /> |
||
== Meldepflicht == |
== Meldepflicht == |
||
| Zeile 253: | Zeile 260: | ||
<ref name="abas_einstuf">{{Internetquelle |url=https://www.baua.de/DE/Aufgaben/Geschaeftsfuehrung-von-Ausschuessen/ABAS/pdf/SARS-CoV-2.pdf |titel=Beschluss 1/2020 des ABAS vom 19.2.2020 und Begründung zur vorläufigen Einstufung des Virus SARS-CoV-2 in Risikogruppe 3 und Empfehlungen zu nicht gezielten Tätigkeiten (Labordiagnostik) und gezielten Tätigkeiten mit SARS-CoV-2 |werk=Website der [[Bundesanstalt für Arbeitsschutz und Arbeitsmedizin]] |format=PDF; 140 kB |datum=2020-02-19|abruf=2020-02-23}}</ref> |
<ref name="abas_einstuf">{{Internetquelle |url=https://www.baua.de/DE/Aufgaben/Geschaeftsfuehrung-von-Ausschuessen/ABAS/pdf/SARS-CoV-2.pdf |titel=Beschluss 1/2020 des ABAS vom 19.2.2020 und Begründung zur vorläufigen Einstufung des Virus SARS-CoV-2 in Risikogruppe 3 und Empfehlungen zu nicht gezielten Tätigkeiten (Labordiagnostik) und gezielten Tätigkeiten mit SARS-CoV-2 |werk=Website der [[Bundesanstalt für Arbeitsschutz und Arbeitsmedizin]] |format=PDF; 140 kB |datum=2020-02-19|abruf=2020-02-23}}</ref> |
||
<ref name="abas_info">{{Internetquelle |url=https://www.baua.de/DE/Angebote/Aktuelles/Meldungen/2020/2020-02-19-Coronavirus.html |titel=Informationen der BAuA: Neuartiges Virus SARS-CoV-2 (bislang 2019-nCoV) durch den ABAS in Risikogruppe 3 eingestuft und Empfehlungen zur Labordiagnostik gegeben |werk=Website der [[Bundesanstalt für Arbeitsschutz und Arbeitsmedizin]] (BAuA) |datum=2020-02-19|abruf=2020-02-23}}</ref> |
<ref name="abas_info">{{Internetquelle |url=https://www.baua.de/DE/Angebote/Aktuelles/Meldungen/2020/2020-02-19-Coronavirus.html |titel=Informationen der BAuA: Neuartiges Virus SARS-CoV-2 (bislang 2019-nCoV) durch den ABAS in Risikogruppe 3 eingestuft und Empfehlungen zur Labordiagnostik gegeben |werk=Website der [[Bundesanstalt für Arbeitsschutz und Arbeitsmedizin]] (BAuA) |datum=2020-02-19|abruf=2020-02-23}}</ref> |
||
<ref name="Bai_JAMA_20200221">{{Literatur |Autor=Yan Bai, Lingsheng Yao, Tao Wei, Fei Tian, Dong-Yan Jin, Lijuan Chen, Meiyun Wang |Titel=Presumed Asymptomatic Carrier Transmission of COVID-19 |Sammelwerk=[[Journal of the American Medical Association]] |Datum=2020-02-21 |Sprache=en |DOI=10.1001/jama.2020.2565}}</ref> |
|||
<ref name="Chan_Lancet_20200124">{{Literatur |Autor=Jasper Fuk-Woo Chan, Shuofeng Yuan, Kin-Hang Kok, Kelvin Kai-Wang To, Hin Chu, Jin Yang, Fanfan Xing, Jieling Liu, Cyril Chik-Yan Yip, Rosana Wing-Shan Poon, Hoi-Wah Tsoi, Simon Kam-Fai Lo, Kwok-Hung Chan, Vincent Kwok-Man Poon, Wan-Mui Chan, Jonathan Daniel Ip, Jian-Piao Cai, Vincent Chi-Chung Cheng, Honglin Chen, Christopher Kim-Ming Hui, Kwok-Yung Yuen |Titel=A familial cluster of pneumonia associated with the 2019 novel coronavirus indicating person-to-person transmission: a study of a family cluster |Sammelwerk=[[The Lancet]] |Datum=2020-01-24 |Sprache=en |DOI=10.1016/S0140-6736(20)30154-9}}</ref> |
<ref name="Chan_Lancet_20200124">{{Literatur |Autor=Jasper Fuk-Woo Chan, Shuofeng Yuan, Kin-Hang Kok, Kelvin Kai-Wang To, Hin Chu, Jin Yang, Fanfan Xing, Jieling Liu, Cyril Chik-Yan Yip, Rosana Wing-Shan Poon, Hoi-Wah Tsoi, Simon Kam-Fai Lo, Kwok-Hung Chan, Vincent Kwok-Man Poon, Wan-Mui Chan, Jonathan Daniel Ip, Jian-Piao Cai, Vincent Chi-Chung Cheng, Honglin Chen, Christopher Kim-Ming Hui, Kwok-Yung Yuen |Titel=A familial cluster of pneumonia associated with the 2019 novel coronavirus indicating person-to-person transmission: a study of a family cluster |Sammelwerk=[[The Lancet]] |Datum=2020-01-24 |Sprache=en |DOI=10.1016/S0140-6736(20)30154-9}}</ref> |
||
<ref name="CDC_1">{{Internetquelle |url=https://www.cdc.gov/coronavirus/2019-ncov/about/transmission.html |titel=How COVID-19 Spreads |werk=Website der US-amerikanischen [[Centers for Disease Control and Prevention]] (CDC) |datum=2020-02-17 |sprache=en |abruf=2020-02-23}}</ref> |
<ref name="CDC_1">{{Internetquelle |url=https://www.cdc.gov/coronavirus/2019-ncov/about/transmission.html |titel=How COVID-19 Spreads |werk=Website der US-amerikanischen [[Centers for Disease Control and Prevention]] (CDC) |datum=2020-02-17 |sprache=en |abruf=2020-02-23}}</ref> |
||
| Zeile 262: | Zeile 270: | ||
<ref name="Fischer_Spektrum_20200210">{{Internetquelle |autor=Lars Fischer, Alina Schadwinkel |url=https://www.spektrum.de/wissen/verursacht-das-coronavirus-engpaesse-bei-medikamenten/1700384 |titel=Verursacht das Coronavirus Engpässe bei Medikamenten? |titelerg=Stammt das Virus aus dem Pangolin? |hrsg=Website [[Spektrum.de]] |datum=2020-02-10 |abruf=2020-02-15}}</ref> |
<ref name="Fischer_Spektrum_20200210">{{Internetquelle |autor=Lars Fischer, Alina Schadwinkel |url=https://www.spektrum.de/wissen/verursacht-das-coronavirus-engpaesse-bei-medikamenten/1700384 |titel=Verursacht das Coronavirus Engpässe bei Medikamenten? |titelerg=Stammt das Virus aus dem Pangolin? |hrsg=Website [[Spektrum.de]] |datum=2020-02-10 |abruf=2020-02-15}}</ref> |
||
<ref name="Huang_Lancet_20200124">{{Literatur |Autor=Huang C, Wang Y, Li X, Ren L, Zhao J, Hu Y, Zhang L, Fan G, Xu J, Gu X, Cheng Z, Yu T, Xia J, Wei Y, Wu W, Xie X, Yin W, Li H, Liu M, Xiao Y, Gao H, Guo L, Xie J, Wang G, Jiang R, Gao Z, Jin Q, Wang J, Cao B |Titel=Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China |Sammelwerk=[[The Lancet]] |Datum=2020-01-24 |Sprache=en |DOI=10.1016/S0140-6736(20)30183-5}}</ref> |
<ref name="Huang_Lancet_20200124">{{Literatur |Autor=Huang C, Wang Y, Li X, Ren L, Zhao J, Hu Y, Zhang L, Fan G, Xu J, Gu X, Cheng Z, Yu T, Xia J, Wei Y, Wu W, Xie X, Yin W, Li H, Liu M, Xiao Y, Gao H, Guo L, Xie J, Wang G, Jiang R, Gao Z, Jin Q, Wang J, Cao B |Titel=Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China |Sammelwerk=[[The Lancet]] |Datum=2020-01-24 |Sprache=en |DOI=10.1016/S0140-6736(20)30183-5}}</ref> |
||
<ref name="Lu_Lancet_20200129">{{Literatur |Autor=Roujian Lu, Xiang Zhao, Juan Li, Peihua Niu, Bo Yang, Honglong Wu, Wenling Wang, Hao Song, Baoying Huang, Na Zhu, Yuhai Bi, Xuejun Ma, Faxian Zhan, Liang Wang, Tao Hu, Hong Zhou, Zhenhong Hu, Weimin Zhou, Li Zhao, Jing Chen, Yao Meng, Ji Wang, Yang Lin, Jianying Yuan, Zhihao Xie, Jinmin Ma, William J Liu, Dayan Wang, Wenbo Xu, Edward C Holmes, George F Gao, Guizhen Wu, Weijun Chen, Weifeng Shi, Wenjie Tan |Titel=Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding |Sammelwerk=[[The Lancet]] |Datum=2020-01-29 |Sprache=en |DOI=10.1016/S0140-6736(20)30251-8}}</ref> |
|||
<ref name="MN908947">{{Internetquelle |autor=F. Wu, S. Zhao, B. Yu, Y.-M. Chen, W. Wang, Y. Hu, Z.-G. Song, Z.-W. Tao, J.-H. Tian, Y.-Y. Pei, M. L. Yuan, Y.-L. Zhang, F.-H. Dai, Y. Liu, Q.-M. Wang, J.-J. Zheng, L. Xu, E. C. Holmes, Y.-Z. Zhang |url=https://www.ncbi.nlm.nih.gov/nuccore/MN908947.3 |titel=Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome |werk=Website Nucleotide des [[National Center for Biotechnology Information]] (NCBI) |abruf=2020-02-04}}</ref> |
<ref name="MN908947">{{Internetquelle |autor=F. Wu, S. Zhao, B. Yu, Y.-M. Chen, W. Wang, Y. Hu, Z.-G. Song, Z.-W. Tao, J.-H. Tian, Y.-Y. Pei, M. L. Yuan, Y.-L. Zhang, F.-H. Dai, Y. Liu, Q.-M. Wang, J.-J. Zheng, L. Xu, E. C. Holmes, Y.-Z. Zhang |url=https://www.ncbi.nlm.nih.gov/nuccore/MN908947.3 |titel=Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome |werk=Website Nucleotide des [[National Center for Biotechnology Information]] (NCBI) |abruf=2020-02-04}}</ref> |
||
<ref name="nucleotide">{{Internetquelle |url=https://www.ncbi.nlm.nih.gov/nuccore/?term=txid2697049 |titel=NCBI Database Nucleotide, txid2697049 (Severe acute respiratory syndrome coronavirus 2) |werk=Website Nucleotide des [[National Center for Biotechnology Information]] (NCBI) |abruf=2020-02-16}}</ref> |
<ref name="nucleotide">{{Internetquelle |url=https://www.ncbi.nlm.nih.gov/nuccore/?term=txid2697049 |titel=NCBI Database Nucleotide, txid2697049 (Severe acute respiratory syndrome coronavirus 2) |werk=Website Nucleotide des [[National Center for Biotechnology Information]] (NCBI) |abruf=2020-02-16}}</ref> |
||
<ref name="RKI_FAQ">{{Internetquelle |autor= |url=https://www.rki.de/SharedDocs/FAQ/NCOV2019/FAQ_Liste.html |titel=Antworten auf häufig gestellte Fragen zum neuartigen Coronavirus (2019-nCoV) |werk=Website des [[Robert Koch-Institut]]s |datum=2020-02-21 |abruf=2020-02-23}}</ref> |
<ref name="RKI_FAQ">{{Internetquelle |autor= |url=https://www.rki.de/SharedDocs/FAQ/NCOV2019/FAQ_Liste.html |titel=Antworten auf häufig gestellte Fragen zum neuartigen Coronavirus (2019-nCoV) |werk=Website des [[Robert Koch-Institut]]s |datum=2020-02-21 |abruf=2020-02-23}}</ref> |
||
<ref name="who_lab_testing">{{Internetquelle |url=https://www.who.int/publications-detail/laboratory-testing-for-2019-novel-coronavirus-in-suspected-human-cases-20200117 |titel=Laboratory testing for 2019 novel coronavirus (2019-nCoV) in suspected human cases |werk=Website WHO |hrsg=[[Weltgesundheitsorganisation]] (WHO) |datum=2020-01-17 |sprache=en |abruf=2020-02-05}}</ref> |
<ref name="who_lab_testing">{{Internetquelle |url=https://www.who.int/publications-detail/laboratory-testing-for-2019-novel-coronavirus-in-suspected-human-cases-20200117 |titel=Laboratory testing for 2019 novel coronavirus (2019-nCoV) in suspected human cases |werk=Website WHO |hrsg=[[Weltgesundheitsorganisation]] (WHO) |datum=2020-01-17 |sprache=en |abruf=2020-02-05}}</ref> |
||
<ref name="WHO_report_22">{{Internetquelle |url=https://www.who.int/docs/default-source/coronaviruse/situation-reports/20200211-sitrep-22-ncov.pdf |
<ref name="WHO_report_22">{{Internetquelle |url=https://www.who.int/docs/default-source/coronaviruse/situation-reports/20200211-sitrep-22-ncov.pdf |titel=Novel Coronavirus (2019-nCoV) |titelerg=Situation Report – 22 |hrsg=[[WHO]] |datum=2020-02-11 |format=PDF; 1,0 MB |abruf=2020-02-13}}</ref> |
||
<ref name="WHO_report_30">{{Internetquelle |url=https://www.who.int/docs/default-source/coronaviruse/situation-reports/20200219-sitrep-30-covid-19.pdf |titel=Coronavirus disease 2019 (COVID-19) |titelerg=Situation Report – 30 |hrsg=[[WHO]] |datum=2020-02-19 |format=PDF; 1,1 MB |abruf=2020-02-23}}</ref> |
|||
<ref name="WHO-reports">{{Internetquelle |url=https://www.who.int/emergencies/diseases/novel-coronavirus-2019/situation-reports |titel=Novel Coronavirus (2019-nCoV) situation reports; Coronavirus disease (COVID-2019) situation reports |hrsg=WHO |sprache=en |abruf=2020-01 |abruf-verborgen=1 |kommentar=abgerufen im Januar bis Februar 2020}}</ref> |
<ref name="WHO-reports">{{Internetquelle |url=https://www.who.int/emergencies/diseases/novel-coronavirus-2019/situation-reports |titel=Novel Coronavirus (2019-nCoV) situation reports; Coronavirus disease (COVID-2019) situation reports |hrsg=WHO |sprache=en |abruf=2020-01 |abruf-verborgen=1 |kommentar=abgerufen im Januar bis Februar 2020}}</ref> |
||
<ref name="Zhou_Nature_20200203">{{Literatur |Autor=Peng Zhou, Xing-Lou Yang, Xian-Guang Wang, Ben Hu, Lei Zhang, Wei Zhang, Hao-Rui Si, Yan Zhu, Bei Li, Chao-Lin Huang, Hui-Dong Chen, Jing Chen, Yun Luo, Hua Guo, Ren-Di Jiang, Mei-Qin Liu, Ying Chen, Xu-Rui Shen, Xi Wang, Xiao-Shuang Zheng, Kai Zhao, Quan-Jiao Chen, Fei Deng, Lin-Lin Liu, Bing Yan, Fa-Xian Zhan, Yan-Yi Wang, Geng-Fu Xiao, Zheng-Li Shi |Titel=A pneumonia outbreak associated with a new coronavirus of probable bat origin |Sammelwerk=[[Nature]] |Datum=2020-02-03 |Sprache=en |DOI=10.1038/s41586-020-2012-7|Kommentar=dieser Artikel wurde am 23. Januar 2020 vorab ohne Peer-Review auf bioRxiv veröffentlicht}}</ref> |
|||
<ref name="Zhu_NEJM_20200124">{{Literatur |Autor=Na Zhu, Dingyu Zhang, Wenling Wang, Xingwang Li, Bo Yang, Jingdong Song, Xiang Zhao, Baoying Huang, Weifeng Shi, Roujian Lu, Peihua Niu, Faxian Zhan, Xuejun Ma, Dayan Wang, Wenbo Xu, Guizhen Wu, George F. Gao, Wenjie Tan for the China Novel Coronavirus Investigating and Research Team |Titel=A Novel Coronavirus from Patients with Pneumonia in China, 2019 |Sammelwerk=[[The New England Journal of Medicine]] |Datum=2020-01-24 |Sprache=en |DOI=10.1056/NEJMoa2001017}}</ref> |
<ref name="Zhu_NEJM_20200124">{{Literatur |Autor=Na Zhu, Dingyu Zhang, Wenling Wang, Xingwang Li, Bo Yang, Jingdong Song, Xiang Zhao, Baoying Huang, Weifeng Shi, Roujian Lu, Peihua Niu, Faxian Zhan, Xuejun Ma, Dayan Wang, Wenbo Xu, Guizhen Wu, George F. Gao, Wenjie Tan for the China Novel Coronavirus Investigating and Research Team |Titel=A Novel Coronavirus from Patients with Pneumonia in China, 2019 |Sammelwerk=[[The New England Journal of Medicine]] |Datum=2020-01-24 |Sprache=en |DOI=10.1056/NEJMoa2001017}}</ref> |
||
<ref name="Zou_NEJM_20200219">{{Literatur |Autor=Lirong Zou, Feng Ruan, Mingxing Huang, Lijun Liang, Huitao Huang, Zhongsi Hong, Jianxiang Yu, Min Kang, Yingchao Song, Jinyu Xia, Qianfang Guo, Tie Song, Jianfeng He, Hui-Ling Yen, Malik Peiris, Jie Wu |Titel=SARS-CoV-2 Viral Load in Upper Respiratory Specimens of Infected Patients |Sammelwerk=[[The New England Journal of Medicine]] |Datum=2020-02-19 |Sprache=en |DOI=10.1056/NEJMc2001737}}</ref> |
<ref name="Zou_NEJM_20200219">{{Literatur |Autor=Lirong Zou, Feng Ruan, Mingxing Huang, Lijun Liang, Huitao Huang, Zhongsi Hong, Jianxiang Yu, Min Kang, Yingchao Song, Jinyu Xia, Qianfang Guo, Tie Song, Jianfeng He, Hui-Ling Yen, Malik Peiris, Jie Wu |Titel=SARS-CoV-2 Viral Load in Upper Respiratory Specimens of Infected Patients |Sammelwerk=[[The New England Journal of Medicine]] |Datum=2020-02-19 |Sprache=en |DOI=10.1056/NEJMc2001737}}</ref> |
||
Version vom 24. Februar 2020, 12:01 Uhr
| SARS-CoV-2 | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ||||||||||||||||
| Systematik | ||||||||||||||||
| ||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||
| ||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||
| Severe Acute Respiratory Syndrome Coronavirus 2 | ||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||
| SARS-CoV-2 | ||||||||||||||||
| Links | ||||||||||||||||
|
SARS-CoV-2 (Sars-CoV-2; vormals 2019-nCoV, 2019-novel Corona virus, „neuartiges Coronavirus 2019“ sowie Wuhan-Virus[5]) ist die Bezeichnung eines im Januar 2020 in der chinesischen Stadt Wuhan in der Provinz Hubei neu identifizierten Coronavirus. Das Virus verursacht die Erkrankung namens COVID-19 (oder Covid-19, für Corona virus disease 2019)[6] und ist Auslöser der Coronavirus-Epidemie 2019/2020, die von der WHO als „gesundheitliche Notlage von internationaler Tragweite“ eingestuft wurde. In der Öffentlichkeit wird das Virus meist nur kurz (nach der Virus-Familie) als Coronavirus oder gelegentlich (nach der Krankheit) als Covid-19-Virus bezeichnet.[7]
Entdeckungsgeschichte
Im Dezember 2019 wurde in der Stadt Wuhan eine Häufung von schweren Lungenentzündungen unklarer Ursache festgestellt. Am 30. Dezember 2019 informierte der chinesische Arzt Li Wenliang in einer WeChat-Gruppe seine Arztkollegen über sieben Patienten, die mit Verdacht auf eine Infektion mit dem SARS-Virus im Zentralkrankenhaus Wuhan behandelt wurden.[8] Die chinesische Seuchenschutzbehörde CCDC (Chinese Center for Disease Control and Prevention) entsandte am 31. Dezember 2019 ein Team in die Stadt.[9] Am selben Tag wurde das China-Büro der Weltgesundheitsorganisation (WHO) durch die chinesischen Behörden offiziell darüber informiert, dass im Dezember 2019 in Wuhan mehrere Personen an einer schweren Lungenentzündung erkrankt waren und dass als deren Ursache ein bis dahin uncharakterisierter, infektiöser Erreger vermutet wurde. Bis zum 3. Januar 2020 wurden der WHO insgesamt 44 Erkrankte gemeldet, darunter mehrere Schwerkranke. Da mehrere Erkrankte auf dem örtlichen „Südchinesischen Markt für Fische und Meeresfrüchte“ (chinesisch 武汉华南海鲜批发市场, Pinyin Wǔhàn huánán hǎixiān pīfā shìchǎng – „Wuhan Huanan Großhandelsmarkt für Fische und Meeresfrüchte“) als Verkäufer oder Händler arbeiteten, wurde in diesem Markt der primäre Infektionsort vermutet.[10][11]
Am 7. Januar 2020 gab der mit der Virusidentifizierung befasste, leitende chinesische Virologe Xu Jianguo (徐建国) bekannt, dass es sich bei dem Krankheitserreger um ein bis dahin unbekanntes Coronavirus handle. Dies hätten Untersuchungen von Blutproben und Rachenabstrichen von 15 Erkrankten ergeben. In einer Stellungnahme der WHO am 9. Januar 2020 wurde diese Erkenntnis bestätigt.[12][13] Am 13. Januar 2020 wurde die komplette Genomsequenz eines Isolats des neuen Coronavirus in der NCBI-GenBank hinterlegt (GenBank-Nummer MN908947).[14] Nahezu gleichzeitig wurde ein erstes Nachweisverfahren publiziert.[15][16][17]
Benennung
Die vom 13. Januar 2020 bis zum 11. Februar 2020 von der WHO verwendete Bezeichnung „2019-nCoV“ galt laut WHO als „vorläufig“.[18] Das erste sequenzierte Virusisolat wurde in der Erstbeschreibung als WH-Human-1 coronavirus (WHCV) bezeichnet (WH = Wuhan)<--! Quelle WHCV (biorxiv) geprüft, im Artikel ist nicht die Bezeichnung WH-Human-1 coronavirus zu finden --> und als Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1 in die NCBI Taxonomie-Datenbank (die für Virusnamen und -klassifikationen nicht maßgebend ist) aufgenommen. Das Virus wurde dort danach – ebenfalls vorläufig – als Wuhan seafood market pneumonia virus geführt; als Synonyme wurden 2019-nCoV und Wuhan coronavirus erwähnt.[19] Namensvorschläge, das Virus in Anlehnung an MERS-CoV (Middle East respiratory syndrome coronavirus) nach dem Ort seiner Erstidentifikation, der Stadt Wuhan, als Wuhan respiratory syndrome coronavirus (WRS-CoV) zu benennen, wurden von der WHO nicht aufgegriffen. Ein von Fachleuten genannter Grund waren Beschwerden in der Vergangenheit, als Viren ihren Namen nach einzelnen Ländern oder Regionen erhalten hatten[20] (beispielsweise das Marburg-Virus). Die WHO hatte daher 2015 Empfehlungen herausgegeben, wie neue Krankheitserreger und Erkrankungen zu benennen seien. Eine Benennung nach dem Entdeckungsort wurde dabei explizit für unerwünscht erklärt.[21] In der NCBI Taxonomie-Datenbank aufgeführte Synonyme sind 2019-nCoV, COVID-19, COVID-19 virus, Wuhan coronavirus und Wuhan seafood market pneumonia virus (Stand 16. Februar 2020).[22]
Am 11. Februar 2020 gab die WHO bekannt, die durch das Virus verursachte Erkrankung als „COVID-19“ bzw. „Covid-19“ (Corona virus disease 2019) benannt zu haben.[6][23] Am selben Tag wurde von der Coronavirus Study Group (CSG) des International Committee on Taxonomy of Viruses (ICTV) auf dem Preprint-Server bioRxiv für das Virus die Bezeichnung SARS-CoV-2 (Severe Acute Respiratory Syndrome Coronavirus 2) vorgeschlagen.[1] Dem widersprach eine Woche später eine Gruppe chinesischer Virologen, die stattdessen als Bezeichnung „Humanes Coronavirus 2019“ (human coronavirus 2019, HCoV-19) vorschlug. Als Begründung nannten sie die Ähnlichkeit dieser Virusbezeichnung mit der Bezeichnung der von der WHO als COVID-19 benannten Krankheit und die Gefahr der Verwechslung von SARS-CoV-2 mit SARS-CoV in der Öffentlichkeit. Sie betonten, dass sich „2019-nCoV“ von dem SARS-Virus in biologischer und epidemiologischer Hinsicht unterscheidet, ebenso wie die klinischen Symptome von COVID-19 und SARS verschieden sind.[24] Zur Unterscheidung wird der Erreger von SARS auch als SARS-CoV-1 bezeichnet.[25]
Merkmale
Molekulargenetik und Phylogenetik
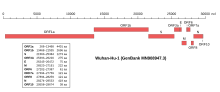
Das Virusgenom besteht, wie in Coronaviren üblich, aus einzelsträngiger RNA (ssRNA) mit positiver Polarität. Das Isolat Wuhan-Hu-1 (NCBI GenBank-Nummer MN908947[26]) umfasst 29.903 nt (Nukleotide) mit zwei 265 nt bzw. 229 nt langen untranslatierten Bereichen am 5′-Ende bzw. am 3′-Ende.[14] Die putativen (vermuteten) Gene könnten für zehn Proteine codieren: ein 7096 Aminosäuren (AS) langes ORF1ab-Polyprotein (Replikase-Komplex), ein 1273 AS langes Oberflächen-Glykoprotein (S für englisch spikes, vergleiche Peplomer), ein 75 AS langes Hüllprotein (E für engl. envelope, vergleiche Virushülle), ein 222 AS langes Membran-Glykoprotein (M), ein 419 AS langes Nukleokapsid-Phosphoprotein (N) und weitere fünf Proteine (ORF3a, ORF6, ORF7a, ORF8 und ORF10).[14] Die Abfolge der Gene entspricht jener des SARS-Virus und der aller Coronaviren.[27]
Mittlerweile (Stand 16. Februar 2020) gibt es mehr als 40 vollständige Genomanalysen von SARS-CoV-2-Isolaten. Die Genomgröße liegt zwischen 29.825 und 29.903 nt.[26] Der GC-Gehalt (der Anteil der Nukleinbasen Guanin und Cytosin) liegt bei 38,0 Mol-Prozent.[28][29] Die beiden Virusisolate HKU-SZ-002a (NCBI GenBank-Nummer MN938384[26]) und HKU-SZ-005b (NCBI GenBank-Nummer MN975262[26]) stammen von Patienten einer Familie aus Shenzhen und unterscheiden sich lediglich durch zwei Nukleotide. Die Genomanalyse dieser beiden Isolate ergab, dass sie nahe verwandt mit den bei Fledermäusen (englisch bat) auftretenden SARS-ähnlichen Coronaviren bat-SL-CoVZXC21 (NCBI GenBank-Nummer MG772934) und bat-SL-CoVZC45 (NCBI GenBank-Nummer MG772933) sind, zu letzterem besteht eine Übereinstimmung in der Nukleotidabfolge von 89 %. Das Genom der beiden Fledermaus-Coronaviren wurde 2018 sequenziert, bat-SL-CoVZC45 wurde bei der Chinesischen Hufeisennase (Rhinolophus sinicus) aus der Familie der Hufeisennasen (Rhinolophidae) gefunden, die Wirtstiere wurden in Zhoushan in der ostchinesischen Provinz Zhejiang in den Jahren 2015 und 2017 untersucht.[29]
Ein weiteres Virusisolat (WIV04, NCBI GenBank-Nummer MN996528[26]) von SARS-CoV-2 aus der bronchoalveolären Spülflüssigkeit eines der ersten Patienten zeigt ebenfalls phylogenetisch größte Ähnlichkeit mit einem bei Fledermäusen (Rhinolophus affinis) in China isolierten Coronavirus BatCoV RaTG13, die Genomsequenzen stimmen zu 96,2 % überein.[30] Auch eine am 27. Januar 2020 publizierte genetische Analyse verwies auf Fledermäuse als mutmaßlicher Ursprungswirt des Virus.[31] Am 29. Januar 2020 wurde in der Fachzeitschrift The Lancet eine genetische Analyse von zehn Virusproben publiziert, die bei neun Erkrankten gewonnen worden waren. Demnach war die Genomsequenz aller zehn Proben zu 99,98 Prozent identisch, was darauf hinweist, dass die neu entdeckte Coronavirusvariante erst vor Kurzem auf den Menschen übergegangen ist.[32][33][34] Die Genomsequenz stimmt zu 88 bzw. 87 % Prozent mit den Genomsequenzen der bei Fledermäusen auftretenden bat-SL-CoVZC45 und bat-SL-CoVZXC21 überein. Die zehn Proben zeigen hingegen nur rund 79 Prozent Übereinstimmung in der Genomsequenz zu SARS-CoV und rund 50 Prozent zu MERS-CoV. Die Ergebnisse der phylogenetischen Untersuchungen werden auch als phylogenetischer Baum veranschaulicht.[29][32] Eine darauf basierende Darstellung ist im Artikel Betacoronavirus zu finden.
Der Aufbau des Genoms sowohl der SARS-CoV-2-Isolate wie auch der genannten Fledermaus-Coronaviren ist typisch für Viren der Lineage B (Untergattung Sarbecovirus, englisch SARS-like Betacoronavirus) der Gattung Betacoronavirus. Aufgrund der genetischen Distanzen zu SARS-CoV und zu MERS-CoV wurde SARS-CoV-2 zunächst als eine in Bezug auf den Menschen neue, ihn infizierende Betacoronavirus-Spezies angesehen.[29][32] Aufgrund der großen genetischen Übereinstimmung mit dem ursprünglichen SARS-Coronavirus hatte am 11. Februar 2020 die Coronavirus Study Group des ICTV jedoch vorgeschlagen, das neue Virus derselben Spezies Severe acute respiratory syndrome-related coronavirus zuzuordnen wie das bisherige.[1]
Das S-Protein (S für englisch spikes) ist für die Bindung an die Wirtszelle verantwortlich, funktionell wird es in die S1-Domäne und die S2-Domäne unterschieden. Die S1-Domäne vermittelt die Bindung an den Oberflächenrezeptor der Wirtszelle, die S2-Domäne vermittelt die Fusion der Zellmembran, durch Endozytose erfolgt dann der Eintritt des Virus in die Zelle. Das S-Gen von SARS-CoV-2 zeigt mit 75 % eine eher geringe Übereinstimmung in der Nukleotidsequenz mit den beiden Fledermausisolaten bat-SL-CoVZC45 und bat-SL-CoVZXC21 im Vergleich zur Genomanalyse. Insbesondere die Nukleotidsequenz, die für die S1-Domäne codiert, unterscheidet sich von diesen deutlich (68 % Übereinstimmung) und weist eine größere Ähnlichkeit mit der entsprechenden Nukleotidsequenz von SARS-CoV auf. Daraus lässt sich schließen, dass SARS-CoV und das neuartige Coronavirus den gleichen Zellrezeptor nutzen, das Angiotensin-konvertierende Enzym 2 (ACE2).[32] Dies konnte experimentell bewiesen werden, verleiche Abschnitt Pathogenese.
Morphologie

Die in einer Zellkultur über mehrere Tage vermehrten Viren können nach Abtrennung durch Ultrazentrifugation für die Untersuchung im Transmissionselektronenmikroskop (TEM) vorbereitet werden, dabei wird eine Negativkontrastierung verwendet. Das TEM-Bild zeigt Virionen von kugelförmiger bis pleomorpher Gestalt mit einem Durchmesser von 60 bis 140 Nanometer (nm). Auf der Oberfläche sind 9 bis 12 nm lange Spikes zu erkennen. Die Morphologie entspricht der anderer bekannter Vertreter der Familie der Coronaviridae. Die Wirtszellen, die im lichtmikroskopischen Bild einen cytopathischen Effekt aufweisen, können nach Fixierung und anschließendem Ultradünnschnitt (Dicke von 80 nm) ebenfalls mit dem TEM untersucht werden. Hier zeigen sich neben Virionen auch Einschlusskörperchen, die mit Viren gefüllte membrangebundene Vesikel im Cytoplasma enthalten.[9]
Herkunft
Das Virus wurde bislang nur bei Menschen nachgewiesen. Fachleute vermuteten bereits zu Beginn der Epidemie, dass als Hauptwirt ein anderes Säugetier oder Geflügel infrage komme. Der Übergang vom Tier auf den Menschen könne jedoch über einen noch nicht identifizierten Zwischenwirt erfolgt sein.[35] Chinesische Forscher verwiesen in diesem Zusammenhang im Journal of Medical Virology auf Schlangen wie den Vielgebänderten Krait (Bungarus multicinctus) und die Chinesische Kobra (Naja atra)[36][37] die auf dem Großmarkt neben anderen lebenden Wildtieren (sogenannten „Ye Wei“) wie Fledermäusen oder Kaninchen angeboten werden.[38] Diese Hypothese wurde von anderen Virologen für unwahrscheinlich erklärt, da es bisher keine Evidenz dafür gäbe, dass Coronaviren auch Reptilien infizieren könnten. Bisher seien Coronaviren ausschließlich bei Säugetieren und Vögeln gefunden worden.[35] Nachdem in Malaiischen Schuppentieren (englisch Sunda pangolins) ein Coronavirus mit 99 % genetischer Übereinstimmung zum SARS-CoV-2 gefunden wurde, geraten diese zunehmend in Verdacht, der Ursprung der neuen Epidemie zu sein, zumal diese trotz Verbots in China gehandelt werden.[23][39]
Virulenz und Pathogenese
Risikogruppe
Für Beschäftigte, die durch ihre berufliche Tätigkeit mit Infektionserregern in Kontakt kommen können, gilt in Deutschland die Biostoffverordnung (BioStoffV). Der bei der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA) eingerichtete Ausschuss für Biologische Arbeitsstoffe (ABAS) hat SARS-CoV-2 am 19. Februar 2020 vorläufig in die Risikogruppe 3 nach der BioStoffV eingeordnet.[40] Grundsätzlich erfolgt die Einstufung in Risikogruppen in den Technischen Regeln für Biostoffe (TRBA), die von der BAuA veröffentlicht werden, für Viren ist dies die TRBA 462: Einstufung von Viren in Risikogruppen. Beim Auftreten neuartiger, noch nicht zugeordneter Krankheitserreger erfolgt zunächst eine vorläufige Einstufung durch den ABAS. In der Begründung wird auf die Ähnlichkeit von SARS-CoV-2 mit dem SARS-CoV-1 hingewiesen, der die SARS-Epidemie 2002/2003 ausgelöst hat und auch die Ähnlichkeit in geringerem Umfang mit dem MERS-CoV wird erwähnt. Diese beiden Viren wurden ebenfalls der Risikogruppe 3 zugeordnet. Der ABAS nennt die „derzeit fehlenden Möglichkeiten zu Impfprävention und Therapie sowie die großen Verbreitungsmöglichkeit in der Bevölkerung“ als Begründung für die vorläufige Zuordnung zur Risikogruppe 3.[41]
Außerdem werden Empfehlungen zur Arbeit mit dem Virus bei der Diagnostik im Labor gegeben: Nicht gezielte Tätigkeiten (vergleiche § 5 BioStoffV) – ausgehend vom Untersuchungsmaterial, also beispielsweise die Probenvorbereitung, Probenaufbereitung und die Inaktivierung, um den Nachweis mittels RT-PCR (siehe Abschnitt Nachweismethoden) durchzuführen – können unter den Bedingungen der Schutzstufe 2 durchgeführt werden. Dabei sind alle Tätigkeiten, bei denen mit Aerosolbildung zu rechnen ist, in einer mikrobiologischen Sicherheitswerkbank der Klasse II durchzuführen. Außerdem ist die entsprechende persönliche Schutzausrüstung zu tragen. Gezielte Tätigkeiten nach § 5 BioStoffV dürfen nur in Laboratorien der Schutzstufe 3 durchgeführt werden, dies betrifft z. B. die Vermehrung des Virus in einer Zellkultur.[41] Die US-amerikanische Seuchenkontrollbehörde Centers for Disease Control and Prevention (CDC) hatte zuvor ähnliche Empfehlungen herausgegeben.[42]
Übertragungsweg
Am 20. Januar 2020 gab die chinesische Gesundheitskommission bekannt, dass eine Mensch-zu-Mensch-Übertragung möglich sei,[5][43] insbesondere wenn zwei Personen engen Kontakt zueinander haben (weniger als 1,8 m Abstand).[44] Es wird angenommen, dass sich das Virus wie andere Erreger von Atemwegserkrankungen durch Tröpfcheninfektion verbreitet.[41][44][13] Das Virus wurde bisher im Sekret des Nasen- und Rachenraumes, im Sputum, dem Stuhl und dem Blut nachgewiesen.[29][45] Chinesische Forscher schließen aufgrund von quantitativen Analysen von RT-PCR-Untersuchungen des Nasenrachenraums, dass das Virus wie Influenza auch durch Aerosole übertragbar ist.[46][40] Bei 17 untersuchten Patienten mit Krankheitssymptomen war die Viruslast in der Nase höher als im Rachen, das Auftreten gerade in den oberen Atemwegen unterscheidet SARS-CoV-2 damit von dem SARS-verursachenden Virus.[46]
Ob eine Übertragung auch durch das Berühren kontaminierter Oberflächen und Gegenstände stattfindet, ist unklar.[44] Eine Auswertung von 22 Studien, die sich mit der Persistenz von medizinisch relevanten Coronaviren (wie SARS-CoV und MERS-CoV) auf Oberflächen beschäftigen, zeigt, dass diese Viren bei Raumtemperatur bis zu neun Tage lang auf Oberflächen aus Metall, Glas oder Plastik überdauern können. Durchschnittlich bleiben sie vier bis fünf Tage infektiös. Allerdings werden sie durch geeignete Desinfektionsmittel inaktiviert (vergleiche Abschnitt Hygienemaßnahmen). Nach Aussage der beteiligten Wissenschaftler sollten diese Erkenntnisse auf SARS-CoV-2 übertragbar sein.[47][48]
In einer Studie mit neun Patientinnen, die im letzten Schwangerschaftsdrittel eine SARS-Cov2-Infektion erlitten hatten, zeigten sich nach der Geburt per Kaiserschnitt alle neun Kinder virusfrei. Daraus schlossen die Studienautoren, dass eine Übertragung des Virus im Mutterleib nicht stattfinde.[49] Die chinesischen Gesundheitsbehörden erfassten bis zum 6. Februar 2020 nur neun Säuglinge, bei denen ein positiver Virusnachweis erbracht wurde. Die Studienautoren zogen als mögliche Ursache dieser geringen Zahl eine mögliche hohe Zahl von Verläufen mit geringen Symptomen unter Kindern wie auch ein Defizit des Meldesystems in Betracht.[50]
Basisreproduktionszahl
Die Auswertung der Daten der ersten 425 Fälle in Wuhan ergab eine Basisreproduktionszahl von 2,2.[51] Eine Modellrechnung mit chinesischen und ausländischen Patientendaten vom 31. Dezember 2019 bis zum 28. Januar 2020 ergab einen Wert von 2,68.[52] Im Vergleich hierzu wurde für SARS eine Basisreproduktionsrate von 2,3 bis 2,6 berechnet.[53] Eine vergleichende Auswertung von 12 Studien, die bis zum 7. Februar 2020 veröffentlicht wurden, kommt zu dem Ergebnis, dass die Basisreproduktionszahl höher liegt, als bisher von der WHO angenommen, deren Schätzung bei 1,4 bis 2,5 liegt.[43] Die Wissenschaftler aus Schweden, China und Deutschland ermitteln, dass die Basisreproduktionszahl im Mittel bei 3,28, im Median bei 2,79 liegt und somit höher als der Wert bei SARS ist, den sie mit 2 bis 5 angeben.[54]
Anteil bestimmter Bevölkerungsgruppen
Eine Mitte Februar 2020 veröffentliche Auswertung der englischsprachigen und chinesischen Fachartikel kommt zu dem Ergebnis, dass alle Bevölkerungsgruppen infiziert werden können. Von den Infizierten waren 72 % 40 Jahre alt oder älter, 64 % waren männlich. 40 % der Patienten hatten chronische Erkrankungen wie Diabetes mellitus und Bluthochdruck.[43]
Anteil schwerer Verläufe und Sterblichkeit
Die WHO gab mit ihrem Situation Report – 18 vom 7. Februar 2020 beispielsweise für China bei 31.211 bestätigt infizierten Personen 4.821 Patienten (15,4 %) mit schweren Krankheitsverläufen bekannt.[55] Genaue Angaben der Sterblichkeit sind im Moment jedoch nicht möglich. Aufgrund bisher noch unbekannter weniger symptomatischer Fälle kann die Letalität einerseits geringer ausfallen. Auf der anderen Seite können die Patienten, die noch nicht genesen sind, noch versterben und damit kann die Letalität höher ausfallen. Auch für den Anteil der schweren Verläufe gilt dies. Bei Diagnosestellung muss noch nicht bekannt sein, ob der Patient schwer erkrankt oder sogar stirbt. Im Folgenden werden einige Studien zitiert, die Hinweise auf die Letalität geben.
Bei einer epidemiologischen Betrachtung von 99 hospitalisierten Fällen waren bis zum 25. Januar 2020 11 % gestorben, 31 % entlassen und 58 % noch im Krankenhaus.[56] Diese Studie ist ein erster Hinweis darauf, dass die Letalität hospitalisierter Patienten bei ca. 11 % liegt.
In einer am 2. Februar 2020 vorab veröffentlichten Studie wurde die Letalität der bestätigten Fälle geschätzt. Hierbei wurde sowohl die Zeit zwischen dem Einsetzen der ersten Krankheitszeichen und der Diagnosestellung (5,1 Tage, 95 % KI: 3,5-7,5) als auch die Zeit zwischen dem Einsetzen der ersten Krankheitszeichen und dem Tod (15,2 Tage, 95 % KI: 13,1-17,7) berücksichtigt. Im 1. Szenario wurde die Epidemie auf Grundlage des Indexpatienten vom 8. Dezember 2019 kalkuliert und eine Letalität von 4,6 % (95 % KI: 3.1-6.6) berechnet. Im 2. Szenario wurde anhand der in andere Länder exportierten Fälle eine Epidemie simuliert und eine Letalität von 7,7 % (95 % KI: 4,9-11,3 %) ermittelt. Die Autoren weisen darauf hin, dass die tatsächliche Letalität durch nicht diagnostizierte Fälle entsprechend niedriger ausfallen könne.[57]
Eine Fallstudie aus einem Krankenhaus in Wuhan beschreibt 138 Patienten mit radiologisch und virologisch gesicherter Pneumonie durch SARS-CoV-2 vom 1. Januar bis zum 2. Februar 2020. Rund ein Viertel dieser Patienten musste intensivmedizinisch behandelt werden. Der häufigste Grund für die Intensivtherapie war ein akutes Atemnotsyndrom, welches in rund der Hälfte der Fälle eine invasive Beatmung notwendig machte. Die Intensivpatienten waren mit im Median 66 Jahren signifikant älter als der Rest der Patienten mit im Median 51 Jahren. Zum Studienendpunkt waren rund 65 % der Patienten noch im Krankenhaus. Unter den Patienten befanden sich 40 Krankenhausmitarbeiter, die sich bei der Arbeit angesteckt hatten, sowie 17 Patienten des Krankenhauses, die in der Einrichtung angesteckt wurden. Die Mehrheit der Patienten erhielt Oseltamivir und Antibiotika. 4,3 % der Patienten verstarben bis zum Studienendpunkt. Rund die Hälfte erhielt Kortikosteroide. Die Studienautoren beschrieben diese antivirale Therapie aus ihrer Beobachtung als nicht effektiv.[58]
Inkubationszeit
Die Inkubationszeit kann laut Informationen des Robert Koch-Instituts (RKI) bis zu 14 Tage betragen.[13] Darüber hinaus gibt es Berichte chinesischer Forscher, welche die mögliche Inkubationszeit auf bis zu 24 Tage ausdehnen.[59] Eine Analyse der ersten 425 in Wuhan gemeldeten Fälle ergibt eine Inkubationszeit von im Mittel 5,2 Tagen und ein Durchschnittsalter von 59 Jahren. Die Autoren gehen davon aus, dass bereits Mitte Dezember 2019 im Umfeld des Fischmarktes Übertragungen von Mensch zu Mensch stattfanden.[51] Eine statistische Auswertung mehrerer Berichte von Infektionen in einem Haushalt oder in anderer enger räumlicher Begrenzung (sogenannte Cluster) ergibt eine Inkubationszeit von im Median 5 – 6 Tagen.[60]
Eine Ansteckung anderer Menschen während der Inkubationszeit ist trotz beschwerdefreien Gesundheitszustands möglich. Tests auf die Viruslast im Blut bei einzelnen Patienten legen den Verdacht nahe, dass manche Patienten auch während der Ausheilung bei klinischer Besserung weiterhin vorübergehend infektiös sein können.[61] Der Bericht dieser Publikation, welcher auf der Annahme einer asymptomatischen chinesischen Indexpatientin beruhen, wurde durch die Recherche der Fachzeitschrift Science widerlegt und vom RKI in Zweifel gezogen.[62] In einer Gruppe von 126 aus Wuhan nach Deutschland Evakuierten zeigten sich zwei Patienten in der RT-PCR des Rachenabstrichs positiv, welche keine oder nur sehr unspezifische Beschwerden aufwiesen.[63] Ebenso ist ein Fall eines subjektiv asymptomatischen zehnjährigen Jungen in Shenzhen beschrieben, dessen Blutbild und Entzündungszeichen im Labor unauffällig waren. In der weiteren Untersuchung zeigten sich jedoch radiologische Befunde vereinbar mit einer Pneumonie, und im Rachenabstrich ließ sich Virus-RNA nachweisen.[29]
Darüber hinaus existiert ein weiterer Fallbericht aus Guangzhou von zwei asymptomatisch Infizierten mit Virusnachweis im Nasenrachenraum. Die Autoren wiesen explizit auf die Verbreitungsgefahr des Virus durch beschwerdefreie Patienten in frühen Infektionsstadien hin.[64] Messungen der Viruslast im Sekret des Nasenrachenraums ergeben eine ähnlich hohe Viruslast zwischen beschwerdefreien und symptomatisch kranken Patienten.[46] Aufgrund von quantitativen Virusuntersuchungen im Sekret des Nasenrachenraums bei Patienten mit sehr leichten Symptomen schlossen die Forscher der Virologie der Charité und des Instituts für Mikrobiologie der Bundeswehr, dass auch bereits bei sehr milden Erkrankungssymptomen eine hohe Infektionsfähigkeit besteht.[65][23] Auch das Robert Koch-Institut berichtet über einzelne Fälle, bei denen sich Betroffene möglicherweise bei infizierten Personen angesteckt haben, die noch keine oder keine spezifischen Symptome gezeigt hatten.[13] Zum gleichen Ergebnis kommt eine chinesische Fallstudie, die sechs Patienten einer Familie betrachtet. Patientin 1 hat ihre fünf Verwandten mit COVID-19 angesteckt, ohne selber Symptome zu zeigen, wegen der Krankheitsfälle wurde auch sie isoliert und ärztlich überwacht. Der Virusnachweis durch RT-PCR bei ihr war nach 17 Tagen negativ, nach 19 Tagen positiv und nach 25 bzw. 30 Tagen wieder negativ.[59]
Pathogenese
Das Virus dringt wie bei SARS über den ACE2-Rezeptor in die menschliche Zelle ein.[30] Im Versuch mit HeLa-Zellen, die ACE2 des Menschen, der Chinesischen Hufeisennase (Rhinolophus sinicus), einer Schleichkatzenart (engl. civet), des Hausschweins und der Maus exprimieren, konnte SARS-CoV-2 das jeweilige ACE2-Protein als Rezeptor nutzen, um in die Zelle einzudringen, nur bei dem Maus-ACE2 gelang dies nicht, ebensowenig bei HeLa-Zellen, die kein ACE2 bildeten. An Rezeptoren, die von anderen Coronaviren genutzt werden, findet keine Bindung von SARS-CoV-2 statt.[30]
In einem Informationsblatt des deutschen Außenministeriums wird erwähnt, dass aus China Berichte „von Infektionsketten über die 4. Generationen hinaus“ vorliegen.[66]
Klinische und laborchemische Krankheitszeichen

Da diese Virusart erst im Januar 2020 nachgewiesen wurde, gibt es bisher nur erste Fallbeschreibungen. Eine Abgrenzung zu anderen Viruserkrankungen wie Influenza ist schwierig. Nach einer Inkubationszeit von bis zu 14 Tagen[13] können Fieber, Myalgien und trockener Husten auftreten. Häufig manifestiert sich die Krankheit auch mit allgemeinem, schwerem Krankheitsgefühl und auch Rückenschmerzen.[29] Symptome der oberen Atemwege sind, anders als bei Infektionen mit harmlosen Coronaviren, eher selten, auch Symptome des Verdauungstrakts, wie sie bei SARS häufiger beobachtet wurden, treten nur in wenigen Fällen auf. Im weiteren Verlauf kann sich eine schwere Atemnot aufgrund einer Infektion der unteren Atemwege bis zur Lungenentzündung entwickeln.[67] Diese kann mit Brustschmerzen im Sinne einer Pleuritis einhergehen. Die Mehrheit der Patienten zeigte die für schwere Virusinfekte typische Kombination aus einer Verminderung der gesamten weißen Blutzellen, einer Verminderung der Lymphozyten und einer Erhöhung laborchemischer Entzündungsparameter. Bildgebend zeigten sich in der Computertomographie der Lunge beidseitige, milchglasartige Verschattungen als Zeichen einer Bronchopneumonie.[67]
Die Mehrheit der Krankenhauseinweisungen der ersten Patienten erfolgte nach rund einwöchiger symptomatischer Krankheit aufgrund einer Verschlechterung des Zustandes. In den Fällen, in denen eine intensivmedizinische Behandlung notwendig wurde, ergab sich deren Notwendigkeit nach rund zehn Tagen nach Symptombeginn.[67]
In einer epidemiologischen Studie von 99 hospitalisierten Fällen fanden bei 13 Patienten eine nicht-invasive Beatmung, bei vier Patienten eine invasive Beatmung, bei neun Patienten eine Dialyse aufgrund eines Nierenversagens und bei drei Patienten eine extrakorporale Lungenunterstützung (ECMO) Anwendung.[56]
Ein wesentlicher Unterschied zum SARS-Coronavirus ist der, dass Patienten schon einige Tage vor Einsetzen der Krankheitssymptome infektiös sein können (beim SARS-Coronavirus waren die Patienten hingegen erst nach Auftreten der Symptome infektiös). Die Infektion lässt sich daher schwerer erkennen und schwieriger eindämmen. Bei Quarantänemaßnahmen reicht es deswegen nicht aus, nur die klinisch auffälligen Personen zu isolieren.[34]
Nachweismethoden
Vorgehensweise bei der Diagnostik
Das Virus ist mittels RT-PCR (Reverse-Transkriptase-Polymerase-Kettenreaktion) im Sputum, im Trachealsekret, in der bronchoalveolären Spülflüssigkeit und im Nasenrachen-Abstrich direkt nachzuweisen. Die Laboruntersuchung führt in Deutschland das Konsiliarlabor für Coronaviren an der Charité in Berlin durch. Diese Untersuchung wird nach zwei Kriterien empfohlen:[68]
- für Personen mit Verdacht auf Pneumonie und Aufenthalt im Risikogebiet 14 Tage vor Erkrankungsbeginn;
- für symptomatische Personen, die Kontakt zu einem bestätigten Fall hatten.
Bis zur Entwicklung eines indirekten Nachweises (Antikörpernachweis)[34][69] soll das Blutserum betroffener Personen aufbewahrt werden.[68] (Stand: 23. Januar 2020)
Bei positivem Befund sollte man mit der Therapie beginnen. Ist der Befund zwar negativ, besteht aber ein anhaltend hoher Verdacht auf eine Infektion mit dem neuartigen Coronavirus, empfiehlt das Robert Koch-Institut, die Diagnostik zu wiederholen. Bei erneut negativem Befund gilt die nCoV-Infektion als ausgeschlossen. Lässt sich die Diagnostik von vornherein nicht durchführen, muss erneut mit dem Konsiliarlaboratorium Rücksprache gehalten werden.[70]
Radiologen aus Changsha berichteten aus einer Fallserie von 167 Patienten über fünf Patienten, bei denen zum Zeitpunkt einer durch Computertomographie gesicherten Lungenentzündung die RT-PCR für das Virus negativ ausfiel und der Virusnachweis erst nach mehrmaligen Tests im Verlauf der Erkrankung gelang.[71]
RT-PCR
Die Nachweismethode der Charité ist die real-time quantitative Reverse-Transkriptase-Polymerase-Kettenreaktion (abgekürzt als qRT-PCR oder RT-qPCR). Sie basiert auf der Detektion von zwei Nukleotidsequenzen, bezeichnet als E Gen und RdRp Gen. Das erste Gen codiert für die Virushülle (E für engl. envelope ‚Hülle‘), das zweite Gen für die RNA-abhängige RNA-Polymerase (RdRP für engl. RNA-dependent RNA polymerase). Für das Primerdesign wurden das SARS-assoziierte Coronavirus und weitere, bei Fledermausarten vorkommende SARS-assoziierte Coronaviren (SARS-CoV) verwendet. Die erste Version des real-time RT-PCR-Assays ist erstellt worden, bevor die Genomsequenz von SARS-CoV-2 veröffentlicht wurde. Nach deren Veröffentlichung wurden die Primer ausgewählt, die auch für den Nachweis von SARS-CoV-2 geeignet sind. Das erste Assay (E Gen) dient als Screening, da es mehrere Virusspezies der Untergattung Sarbecovirus (aus der Gattung Betacoronavirus) nachweist. Verläuft diese Untersuchung positiv, so ist ein Bestätigungs-Assay (RdRp Gen) durchzuführen. Falls auch hier ein positives Ergebnis erhalten wird, schließt sich als drittes ein Charakterisierungs-Assay an (ebenfalls RdRp Gen). Die letzten beiden Assays verwenden eine Gensonde, die für SARS-CoV-2 spezifisch ist, beim zweiten Assay ist zusätzlich eine Sonde enthalten, die zur Nukleotidsequenz sowohl des RNA-Teilstücks des SARS-CoV als auch des SARS-CoV-2 passt.[17][72]
Die molekularbiologische Nachweismethode, die im Konsiliarlabor an der Charité verwendet wird, wurde schnell und effektiv entwickelt, eine erste Version war bereits am 13. Januar 2020 verfügbar. Dazu haben chinesische Wissenschaftler bereitwillig unveröffentlichte Befunde beigetragen. Von internationalen Forschungsnetzen kamen grundlegende Daten, und die globale Sektion des Europäischen Virus-Archivs (European Virus Archive – Global, EVAg) lieferte notwendige Produkte (SARS-CoV RNA und RNA-Transkripte) für die Assays.[72] Auch weitere Gruppen von Wissenschaftlern haben ihre entwickelten Methoden veröffentlicht. Dabei handelt es sich um PCR-Protokolle oder Auflistungen geeigneter Primer und deren für die RT-PCR verwendete Stoffmengenkonzentration, beispielsweise von den Centers for Disease Control and Prevention (CDC) in den USA, den CDC in China oder der Universität Hongkong. Sie unterscheiden sich darin, welche Gene der Virus-RNA nachgewiesen werden.[73] Für die Durchführung der qRT-PCR werden 90 Minuten bis drei Stunden benötigt.[74] Anstelle der real-time quantitative-Variante können die Produkte der Reverse-Transkriptase-PCR auch mittels Agarose-Gelelektrophorese nachgewiesen werden.[29]
Weitere Methoden
Genomanalyse
Laboratorien mit Ausstattung für eine Genomanalyse (DNA-Sequenzierung des Genoms) können SARS-CoV-2 auch auf diese Weise identifizieren.[75] Vollständige Genomanalysen von SARS-CoV-2-Isolaten zum Vergleich sind beispielsweise in der Gendatenbank des National Center for Biotechnology Information (NCBI) oder über die GISAID Plattform[76] verfügbar (vergleiche Abschnitt Molekulargenetik).
Nukleinsäurenachweise
Nach Aussage der Weltgesundheitsorganisation (WHO) ist ein vereinfachtes molekularbiologisches Verfahren, die Nucleic Acid Amplification Technology (NAAT) in der Entwicklung, die Assays dafür werden momentan validiert (Stand: 17. Januar 2020). Die NAAT-Methode beruht ebenfalls auf der RT-PCR, das fertig zusammengestellte Assay bietet jedoch den Vorteil, einfacher in der Handhabung zu sein und ließe sich von entsprechend ausgestatteten Routine-Laboratorien verwenden.[75]
Am 5. Februar 2020 gab die US-amerikanische Behörde CDC bekannt, ein derartiges Assay (test kit) für die Anwendung in akkreditierten Diagnoselaboratorien zur Verfügung zu stellen. Das Assay wird als Centers for Disease Control and Prevention (CDC) 2019-Novel Coronavirus (2019-nCoV) Real-Time Reverse Transcriptase (RT)-PCR Diagnostic Panel bezeichnet und ist für den Nachweis sowohl des neuartigen Coronavirus wie auch SARS-ähnlicher Coronaviren in Proben der oberen und unteren Atemwege von Patienten vorgesehen. Zuvor erfolgte eine beschleunigte Zulassung durch die Gesundheitsbehörde Food and Drug Administration (FDA), somit darf das Assay auch außerhalb von Forschungseinrichtungen verwendet werden.[77] Ein Testkit ermöglicht die Untersuchung von 700 – 800 Proben, 100 dieser Packungen gehen an US-amerikanische Labore, weitere 100 an internationale Laboratorien, die beispielsweise im Auftrag der WHO Untersuchungen durchführen.[78] Die Untersuchung dauert von der Probevorbereitung bis zum Vorliegen der Ergebnisse etwa vier Stunden.[74]
Schnelltests
Ende Januar 2020 hatte Xinhua, die Nachrichtenagentur der Regierung der Volksrepublik China gemeldet, dass die chinesische Behörde National Medical Products Administration (NMPA) am 26. Januar vier Testkits eines neuen Testverfahren zugelassen habe. Das Assay ist von dem Biotechnologieunternehmen Sansure Biotech aus Changsha entwickelt worden. Mit Hilfe der dafür geeigneten Laborautomatisierung sollen Testergebnisse bereits nach 30 Minuten vorliegen.[74][79] Die staatliche Nachrichtenagentur teilte weiterhin mit, dass eine in Wuxi in der östlichen Provinz Jiangsu ansässige Firma in Zusammenarbeit mit dem National Institute for Viral Disease Control and Prevention eine Schnellmethode entwickelt habe. Mit dem Testkit soll das Virus innerhalb von 8 – 15 Minuten nachgewiesen werden, die Firma könne täglich so viele Testkits produzieren, dass damit die Untersuchung von 4.000 Proben möglich sei und das Verfahren soll bereits in der Provinz Hubei eingesetzt worden sein.[80] Die Xinhua-Meldungen enthalten keinen Hinweis auf die dabei verwendeten molekularbiologischen Methoden. Weiterhin wird an der chinesischen Tianjin-Universität in Zusammenarbeit mit einem Pekinger Biotechnologiefirma ein Schnellverfahren entwickelt, bei dem nach 15 Minuten Resultate vorliegen sollen, es befindet sich im Probeeinsatz (Stand Februar 2020).[74]
Forscher der Hong Kong University of Science and Technology meldeten Anfang Februar 2020 die Entwicklung eines tragbaren Gerätes, mit dem das neuartige Coronavirus innerhalb von 40 Minuten nachweisbar sein soll. Für das im Vergleich zur herkömmlichen qRT-PCR schnellere Verfahren werden modifizierte Chip-Thermocycler verwendet.[74] Auch Forscher des Institute for Health Innovation & Technology (iHealthtech) an der National University of Singapore berichteten im Februar 2020 darüber, eine Schnellmethode zu entwickeln. Sie basiert auf der seit 2018 verwendeten enVision-Technologie, mit der Nukleinsäuren innerhalb von 30 bis 60 Minuten nachgewiesen werden. Es wird geschätzt, dass bis zur Marktreife des neuen Testverfahrens noch mehrere Monaten benötigt werden.[81]
Antikörpernachweis
Auch der Antikörpernachweis als serologische Untersuchung wird nach Angabe der WHO derzeit entwickelt (Stand: 17. Januar 2020). Dadurch wird es ein Assay (beispielsweise ein Immunassay wie ELISA) geben, mit dem Antikörper aus Patienproben (Blutserum) durch Antigen-Antikörper-Reaktion nachweisbar sind. Die WHO und das Robert Koch-Institut (RKI) in Deutschland rufen dazu auf, Serumproben von bestätigten oder Verdachtsfällen in der Akutphase zu sammeln und zu asservieren.[68] Die WHO empfiehlt, die erste Probe in der ersten Krankheitswoche und die zweite Probe drei bis vier Wochen später zu nehmen. Damit lässt sich eine Serokonversion überprüfen.[75][82]
In einem chinesischen Forschungslabor wurden im Januar 2020 erste ELISA-Tests durchgeführt, als Antigen wurde das Nukleokapsidprotein (N) eines Fledermaus-Coronavirus mit Ähnlichkeit zu SARS-CoV-2 verwendet. Damit ließen sich in Serumproben eines Patienten die Antikörper Immunglobulin G (IgG) und Immunglobulin M (IgM) nachweisen und deren Titer über mehrere Tage während des Krankheitsverlaufes bestimmen. In einem zweiten Test wurden Serumproben, die 20 Tage nach den ersten Symptomen entnommen wurden, untersucht. Alle Patientenseren, aber nicht die Seren von Gesunden zeigten eine stark positive IgG-Reaktion, einige Patientenseren zeigten zusätzlich eine IgM-Reaktion, was auf eine aktuelle Immunantwort, also eine momentane Infektion hindeutet.[30]
Zellkultur
Die Vermehrung des Virus zu Forschungszwecken in einer Zellkultur ist unter anderem in China, Australien, Frankreich, Deutschland und den USA gelungen.[9][69][83][84][85] Die chinesischen Wissenschaftler verwenden hierbei Epithelzellen des menschlichen Atemtrakts, die das mehrschichtige mukoziliäres Epithelgewebe (Flimmerepithel) simulieren, ebenso werden die Zelllinien Vero E6 und Huh-7 eingesetzt.[9][30]
Behandlung
Derzeit steht keine spezifische Behandlung zur Verfügung, allenfalls können Symptome gelindert werden; möglicherweise sind aber einige bereits existierende Virostatika, die zum Beispiel gegen MERS-CoV und HIV eingesetzt werden, auch bei einer Infektion mit SARS-CoV-2 wirksam.[86][87][88] Dazu gehören Proteasehemmer wie Indinavir, Saquinavir, Remdesivir, Lopinavir/Ritonavir und Interferon-beta.[89][90] Die Nationale Gesundheitskommission der VR China empfiehlt eine Kombination der HIV-Proteaseinhibitoren Lopinavir und Ritonavir mit inhalativ verabreichtem Interferon Alpha. In den Vereinigten Staaten wurde das Nukleotidanalogon Remdesivir, das eigentlich gegen Ebolafieber entwickelt wurde, eingesetzt. Ein Wirkungsnachweis steht noch aus.[91]
Neben Remdesivir sind in China zur Zeit noch Favipiravir und Chloroquin, ein Wirkstoff gegen Malaria in der Erprobung am Menschen.[92] Die klinischen Studien für Remdesivir finden in China und weiteren Staaten statt, Favipiravir – ein Wirkstoff, der gegen das Influenzavirus verwendet wird – wird an 70 Patienten in Shenzhen erprobt und hat am 16. Februar 2020 kurzfristig als wirksames Virustatikum für fünf Jahre unter dem Namen Favilavir die Zulassung erhalten und wird ab sofort produziert.[93] Chloroquin wurde in Krankenhäusern in Peking und der Provinz Guangdong an mehr als 100 Patienten getestet und steht mit Umifenovir (Handelsname Arbidol) – ein in Russland und China zugelassener Influenza-Wirkstoff – auf der empfohlenen Medikationsliste (Stand 20. Februar 2020).[94]
Aktuell rufen die Kliniken bereits genesene Patienten zu Blutspenden auf, um mit daraus gewonnenem antikörperreichem Plasma Akutfälle zu therapieren und wertvolle Erkenntnisse zur Entwicklung eines Impfstoffes zu erhalten.[95][96]
In einem Diagnose- und Behandlungsschema für Triagezentren aus Wuhan kommt bei Verdachtspatienten das Virostatikum Umifenovir und eine antibiotische Behandlung mit Linezolid, Nemonoxacin oder Fluorchinolonen zum Einsatz. Die antibiotische Behandlung wird mit einer zu erwartenden bakteriellen Zweitinfektion des durch den Virusinfekt geschädigten Lungengewebes begründet. Die Autoren betonen den Stellenwert des Therapiebeginns vor Eintreffen des RT-PCR-Tests aufgrund klinischer, laborchemischer und apparativer Untersuchungsbefunde. Patienten ohne Nachweis einer Verminderung der Lymphozytenzahl im Blut, ohne virale Pneumonie, ohne Dyspnoe (Atemnot) und ohne Reduktion der Sauerstoffsättigung unter <93 % sollten ohne RT-PCR-Testung in die Heimversorgung entlassen werden. Für diese Patienten ist die orale Gabe von Azithromycin oder Amoxicillin. Patienten mit Pneumonien anderer Genese sollen regulär und nach Möglichkeit ambulant behandelt werden. Schwangere Frauen und Menschen über 65 Jahre sollen als besondere Risikogruppen eher stationär behandelt werden. Die Studienautoren begründen ihr Vorgehen mit der Überforderung des Gesundheitswesens in Wuhan. Die Behandlung möglichst vieler Patienten zu Hause berge Risiken, sei aber notwendig, um die vielen kritischen Fälle zu versorgen.[97]
Vorbeugung
Entwicklung von Impfstoffen
Bereits unmittelbar nach Veröffentlichung der RNA-Sequenz des Virus wurde in mehreren Laboren damit begonnen, einen Impfstoff gegen das Virus zu entwickeln.[98] Die internationale Impfstoffinitiative CEPI (Coalition for Epidemic Preparedness Innovations) plante, bis Mitte Juni 2020 erste Tests mit bis dahin entwickelten Impfstoffen durchzuführen. Dafür erhielten mehrere potentiell geeignete Unternehmen finanzielle Unterstützungen.[23] In Deutschland betraf dies u. a. die Tübinger Biotechnologiefirma CureVac, die an der schnellen Impfstoffentwicklung arbeitete.[99] Das Robert Koch-Institut verwies darauf, dass derzeit klinische Studien mit Impfstoffen gegen MERS-CoV laufen würden.[13] Allerdings sind klinische Studien lediglich der erste Schritt, bis dass ein Impfstoff für die Bevölkerung zur Verfügung stehen würde, können weitere Monate vergehen.[100]
Hygienemaßnahmen
Allgemeinbevölkerung
Das Robert Koch-Institut hat am 28. Januar 2020 Hinweise für die Allgemeinbevölkerung gegeben, wie man das Risiko für eine Ansteckung wesentlich verringern kann. Hierbei wird in erster Linie auf folgende Punkte hingewiesen:
- auf eine gute Händehygiene achten (u. a. Hände mit Seife waschen: vor dem Essen, nach Kontakt zu anderen Menschen, nach dem Toilettengang, nach Niesen/Husten)
- einen Mindestabstand von ca. 1 Meter zu krankheitsverdächtigen Personen halten
- korrekte Hustenetikette (möglichst in die Armbeuge husten oder niesen, nicht in die Hand)
- den Kontakt und damit eine potentielle Infektion zu (Hoch-)Risiko-Gruppen, vornehmlich älteren Personen (>60 Jahre), die meist multimorbid sind, zu deren Schutz zu verringern oder ganz zu vermeiden.[101]
Atemschutzmasken werden hingegen in erster Linie für Kranke selbst oder für Pflegende empfohlen, die engen Kontakt mit Erkrankten haben. Ob das Tragen eines Mund-Nasen-Schutzes in der Öffentlichkeit das Risiko, sich selbst anzustecken, wesentlich reduziert, ist nicht wissenschaftlich belegt.[13]
Medizinisches Personal
Das Robert Koch-Institut hat am 24. Januar 2020 erste Hinweise gegeben, welche Hygienemaßnahmen zur Vermeidung einer Übertragung des Erregers durch Tröpfchen auf medizinisches Personal notwendig sind. Die empfohlenen Schutzmaßnahmen bestehen insbesondere aus dem Tragen eines langärmeligen Schutzkittels, einer Schutzbrille, einer Atemschutzmaske mindestens vom Standard FFP2 und von Schutzhandschuhen. Zur chemischen Desinfektion der Hände und Flächen sind Desinfektionsmittel geeignet, die die Wirkungsbereiche „begrenzt viruzid“, „begrenzt viruzid PLUS“ oder „viruzid“ abdecken.[102][103] Eine Auswertung von 22 Studien, die sich mit der Persistenz und Inaktivierung von medizinisch relevanten Coronaviren (wie SARS-CoV und MERS-CoV) unter anderem in Gesundheitseinrichtungen beschäftigen, zeigt, dass für die Oberflächendesinfektion Mittel auf der Basis von Ethanol, Wasserstoffperoxid oder Natriumhypochlorit in entsprechender Konzentration wirksam sind.[47] Neben der konsequenten Einhaltung der Basishygiene zählen außerdem die Unterbringung in einem Isolierzimmer möglichst mit Schleuse bzw. in einem Einzelzimmer mit eigener Nasszelle und das Abstellen eventuell vorhandener raumlufttechnischer Anlagen, über die ein Luftaustausch mit anderen Räumen möglich ist, zu den empfohlenen Hygienemaßnahmen.[70]
Epidemiologie
Nachdem der WHO bis zum 3. Januar 2020 insgesamt 44 Erkrankte gemeldet worden waren, verstarb am 9. Januar 2020 (Ortszeit) erstmals einer der Erkrankten, ein 61-jähriger Mann,[104] und am 15. Januar 2020 verstarb ein zweiter Erkrankter (im Alter von 69 Jahren) an den Folgen der Virusinfektion.[105]
Am 13. Januar 2020 wurde der erste Erkrankungsfall außerhalb Chinas bekannt. Es handelte sich um eine aus Wuhan am 8. Januar 2020 nach Bangkok (Thailand) eingereiste 61-jährige Frau.[106][107]
Am 20. Januar 2020 berichteten die chinesischen Behörden über einen starken Anstieg der Neuerkrankungen mit 139 neuen Fällen am 18./19. Januar 2020, davon einige auch außerhalb Wuhans in Shenzhen und Peking. Ein weiterer durch das Virus verursachter Todesfall und der erste Fall aus Südkorea wurden gemeldet.[108] Die chinesischen Behörden bestätigten zudem, dass sich Infektionen durch Mensch-zu-Mensch-Übertragung ereignet hatten.[13][29]
An den folgenden Tagen wurden der WHO täglich neue Erkrankungen aus China gemeldet, die Anzahl der Erkrankten erhöhte sich bis zum 26. Januar 2020 weltweit auf mehr als 2000 und die Anzahl der Todesfälle auf mehr als 50.[109] Auch an den folgenden Tagen erhöhten sich die gemeldeten Fallzahlen sprunghaft, so dass am 31. Januar 2020 bereits fast 10.000 Infizierte und mehr als 200 Todesfälle,[109] am 10. Februar mehr als 40.000 Infizierte und mehr als 900 Todesfälle[109] und am 22. Februar mehr als 77.000 Infizierte und mehr als 2.300 Todesfälle[109] der WHO gemeldet worden waren.
Am 27. Januar 2020 wurden erstmals auch in Deutschland Infektionen nachgewiesen.[110] Zunächst bei einem 33-jährigen Mann aus dem Landkreis Landsberg am Lech, der von einer Chinesin – der Indexpatientin – infiziert worden war, die sich vom 19. bis 22. Januar auf Dienstreise in dem Unternehmen Webasto SE in Bayern aufgehalten hatte. Zunächst hatte es geheißen, bei ihr seien Anzeichen für eine Infektion erst während des Rückflugs nach China aufgetreten;[61] tatsächlich hatte sie aber bereits zuvor aufgrund erster Symptome fiebersenkende Mittel eingenommen.[62] Bereits am Abend des 28. Januar wurde bekannt gegeben, dass sich auch drei Kollegen des 33-Jährigen mit SARS-CoV-2 infiziert hatten.[111] An den folgenden Tagen wurde das Virus bei mehreren Kontaktpersonen der Indexpatientin und bei Angehörigen der in Bayern infizierten Personen nachgewiesen, darunter ein fünfjähriges Kind.[99][112]
Am Abend des 30. Januar 2020 erklärte die Weltgesundheitsorganisation (WHO) die Epidemie zu einer Gesundheitlichen Notlage internationaler Tragweite.[113] Am 9. Februar 2020 überstieg die Zahl der registrierten Todesfälle mit über 800 die Gesamtzahl der Todesfälle bei der SARS-Pandemie 2002/2003.[43]
Meldepflicht
In Deutschland ist laut Infektionsschutzgesetz (§ 6) das Auftreten einer bedrohlichen übertragbaren Erkrankung an das zuständige Gesundheitsamt zu melden. Mit Wirkung vom 1. Februar 2020 wurde mittels Verordnung durch das Bundesministerium für Gesundheit (Verordnungsermächtigung nach § 15 IfSG) eine Ausdehnung der Meldepflicht beschlossen. Demnach sind sowohl der Verdacht einer Erkrankung, die Erkrankung und der Tod als auch der laborchemische Nachweis einer akuten Infektion mit dem neuartigen Coronavirus meldepflichtig. Die Meldung des Verdachts hat nur zu erfolgen, wenn der Verdacht sowohl durch das klinische Bild als auch durch einen wahrscheinlichen epidemiologischen Zusammenhang begründet ist. Diese Verordnung gilt bis zum 1. Februar 2021, sofern nicht mit Zustimmung des Bundesrates etwas anderes verordnet wird.[114]
Weblinks
- Johns Hopkins CSSE: Coronavirus COVID-19 Global Cases. Aktuelle Zahlen für bestätigte Infektionen, Todesfälle und Genesungen nach Ländern und Regionen – laufend aktualisiert. In: Website des Johns Hopkins University Center for Systems Science and Engineering. 13. Februar 2020, abgerufen am 13. Februar 2020 (englisch, zum Daten-Update Seite jeweils neu laden).
- COVID-19 (Coronavirus SARS-CoV-2). Übersichtsseite des Robert Koch-Instituts (RKI). In: Website des Robert Koch-Instituts. 13. Februar 2020, abgerufen am 13. Februar 2020.
- Antworten auf häufig gestellte Fragen zum Coronavirus SARS-CoV-2. In: Website des Robert Koch-Instituts. 15. Februar 2020, abgerufen am 15. Februar 2020.
- Atemwegserkrankungen durch neuartiges Coronavirus (2019-nCoV): Virusübertragung durch den Verzehr von Lebensmitteln oder den Kontakt mit Bedarfsgegenständen ist unwahrscheinlich. (PDF; 56 kB) In: Mitteilung des Bundesinstituts für Risikobewertung (BfR). 29. Januar 2020, abgerufen am 8. Februar 2020.
- Kann das neuartige Coronavirus über Lebensmittel und Spielzeug übertragen werden? Fragen und Antworten des BfR. In: Website des Bundesinstituts für Risikobewertung (BfR). 3. Februar 2020, abgerufen am 8. Februar 2020.
- Aktuelle Informationen zum Coronavirus. In: Website des Bundesministerium für Gesundheit. 13. Februar 2020, abgerufen am 13. Februar 2020.
- Q & A on novel coronavirus. In: Website des Europäischen Zentrums für die Prävention und die Kontrolle von Krankheiten (ECDC). 9. Februar 2020, abgerufen am 13. Februar 2020 (englisch).
- Weltgesundheitsorganisation: Coronavirus disease (COVID-19) outbreak. In: Website der Weltgesundheitsorganisation (WHO). (englisch).
- Wie gefährlich ist das neue Coronavirus aus China? Wissenschaftlich geprüft von Jasmina Neudecker. In: YouTube-Kanal von Terra X Lesch & Co. 30. Januar 2020, abgerufen am 8. Februar 2020.
Einzelnachweise
- ↑ a b c Alexander E. Gorbalenya, Susan C. Baker, Ralph S. Baric, Raoul J. de Groot, Christian Drosten, Anastasia A. Gulyaeva, Bart L. Haagmans, Chris Lauber, Andrey M Leontovich, Benjamin W. Neuman, Dmitry Penzar, Stanley Perlman, Leo L.M. Poon, Dmitry Samborskiy, Igor A. Sidorov, Isabel Sola, John Ziebuhr: Severe acute respiratory syndrome-related coronavirus: The species and its viruses – a statement of the Coronavirus Study Group. In: bioRxiv. 11. Februar 2020, S. 1–20, doi:10.1101/2020.02.07.937862 (englisch, biorxiv.org).
- ↑ Phylogeny of SARS-like betacoronaviruses including novel coronavirus (nCoV). In: Website Nextstrain. Abgerufen am 9. Februar 2020.
- ↑ David S. Hui, Esam I. Azhar, Tariq A. Madani, Francine Ntoumi, Richard Kock, Osman Dar, Giuseppe Ippolito, Timothy D. Mchugh, Ziad A. Memish, Christian Drosten, Alimuddin Zumla, Eskild Petersen: The continuing 2019-nCoV epidemic threat of novel coronaviruses to global health – The latest 2019 novel coronavirus outbreak in Wuhan, China. In: International Journal of Infectious Diseases. Band 91, 14. Januar 2020, S. 264–266, doi:10.1016/j.ijid.2020.01.009, PMID 31953166 (englisch, open access).
- ↑ Antonio C. P. Wong, Xin Li, Susanna K. P. Lau, Patrick C. Y. Woo: Global Epidemiology of Bat Coronaviruses. In: Viruses. Band 11, Nr. 2, Februar 2019, S. 174, doi:10.3390/v11020174, PMID 30791586, PMC 6409556 (freier Volltext) – (englisch).
- ↑ a b Nicky Phillips, Smriti Mallapaty, David Cyranoski: How quickly does the Wuhan virus spread? In: Nature. 21. Januar 2020, doi:10.1038/d41586-020-00146-w (englisch).
- ↑ a b Novel Coronavirus (2019-nCoV). (PDF; 1,0 MB) Situation Report – 22. WHO, 11. Februar 2020, abgerufen am 13. Februar 2020.
- ↑ Fast 100 weitere Tote durch Covid-19 in China. Süddeutsche Zeitung, 12. Februar 2020, abgerufen am 13. Februar 2020 (Direkt aus dem dpa-Newskanal).
- ↑ Stephanie Hegarty: The Chinese doctor who tried to warn others about coronavirus. BBC News, 6. Februar 2020, abgerufen am 6. Februar 2020 (englisch).
- ↑ a b c d Na Zhu, Dingyu Zhang, Wenling Wang, Xingwang Li, Bo Yang, Jingdong Song, Xiang Zhao, Baoying Huang, Weifeng Shi, Roujian Lu, Peihua Niu, Faxian Zhan, Xuejun Ma, Dayan Wang, Wenbo Xu, Guizhen Wu, George F. Gao, Wenjie Tan for the China Novel Coronavirus Investigating and Research Team: A Novel Coronavirus from Patients with Pneumonia in China, 2019. In: The New England Journal of Medicine. 24. Januar 2020, doi:10.1056/NEJMoa2001017 (englisch).
- ↑ Pneumonia of unknown cause – China. Website der WHO, 5. Januar 2020, abgerufen am 14. Januar 2020 (englisch).
- ↑ New virus surging in Asia rattles scientists. nature.com, 20. Januar 2020, abgerufen am 28. Januar 2020 (englisch).
- ↑ WHO Statement Regarding Cluster of Pneumonia Cases in Wuhan, China. WHO, 9. Januar 2020, abgerufen am 14. Januar 2020 (englisch).
- ↑ a b c d e f g h Antworten auf häufig gestellte Fragen zum neuartigen Coronavirus (2019-nCoV). In: Website des Robert Koch-Instituts. 21. Februar 2020, abgerufen am 23. Februar 2020.
- ↑ a b c F. Wu, S. Zhao, B. Yu, Y.-M. Chen, W. Wang, Y. Hu, Z.-G. Song, Z.-W. Tao, J.-H. Tian, Y.-Y. Pei, M. L. Yuan, Y.-L. Zhang, F.-H. Dai, Y. Liu, Q.-M. Wang, J.-J. Zheng, L. Xu, E. C. Holmes, Y.-Z. Zhang: Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome. In: Website Nucleotide des National Center for Biotechnology Information (NCBI). Abgerufen am 4. Februar 2020.
- ↑ WHO: Laboratory testing for 2019 novel coronavirus (2019-nCoV) in suspected human cases. Auf: who.int vom 14. Januar 2020.
- ↑ Karola Neubert: Erster Test für das neuartige Coronavirus in China ist entwickelt. In: Website Informationsdienst Wissenschaft (idw). 16. Januar 2020, abgerufen am 9. Februar 2020.
- ↑ a b Victor M. Corman, Tobias Bleicker, Sebastian Brünink, Christian Drosten, Olfert Landt, Marion P. G. Koopmans, Maria Zambon, Malik Peiris: Diagnostic detection of 2019-nCoV by real-time RT-PCR (protocol-v2-1). Hrsg.: Charité Virologie, Berlin. 17. Januar 2019, S. 1–13 (PDF, 1, MB [abgerufen am 1. Februar 2020]).
- ↑ Novel Coronavirus (2019-nCoV). ( vom 28. Januar 2020 im Internet Archive)
- ↑ Taxonomy ID: 2697049 Wuhan seafood market pneumonia virus. ( vom 3. Februar 2020 im Internet Archive)
- ↑ Ching-Tse Cheng: WHO declines to name new pneumonia after 'China' or 'Wuhan'. Taiwan News, 14. Januar 2020, abgerufen am 14. Januar 2020 (englisch).
- ↑ WHO issues best practices for naming new human infectious diseases. Weltgesundheitsorganisation, 8. Mai 2015, abgerufen am 6. Februar 2020 (englisch).
- ↑ Taxonomy Browser Severe acute respiratory syndrome coronavirus 2, Taxonomy ID: 2697049. In: Website National Center for Biotechnology Information (NCBI). Abgerufen am 16. Februar 2020 (englisch).
- ↑ a b c d Lars Fischer, Alina Schadwinkel: Verursacht das Coronavirus Engpässe bei Medikamenten? Stammt das Virus aus dem Pangolin? Website Spektrum.de, 10. Februar 2020, abgerufen am 15. Februar 2020.
- ↑ Shibo Jiang, Zhengli Shi, Yuelong Shu, Jingdong Song, George F. Gao, Wenjie Tan, Deyin Guo: A distinct name is needed for the new coronavirus. In: The Lancet. 19. Februar 2020, doi:10.1016/S0140-6736(20)30419-0 (englisch).
- ↑ Kristian G. Andersen, Andrew Rambaut, W. Ian Lipkin, Edward C. Holmes, Robert F. Garry: The Proximal Origin of SARS-CoV-2, auf: virologica.org, Quelle: ARTIC Network, 17. Februar 2020
- ↑ a b c d e NCBI Database Nucleotide, txid2697049 (Severe acute respiratory syndrome coronavirus 2). In: Website Nucleotide des National Center for Biotechnology Information (NCBI). Abgerufen am 16. Februar 2020.
- ↑ Matthew Frieman, Ralph Baric: Mechanisms of Severe Acute Respiratory Syndrome Pathogenesis and Innate Immunomodulation. In: Microbiology and Molecular Microbiology Reviews. Band 72, Dezember 2008, S. 672–685, doi:10.1128/MMBR.00015-08, PMID 19052324 (englisch, open access).
- ↑ Wuhan seafood market pneumonia virus. In: Website Genome des National Center for Biotechnology Information (NCBI). Abgerufen am 16. Februar 2020.
- ↑ a b c d e f g h i Jasper Fuk-Woo Chan, Shuofeng Yuan, Kin-Hang Kok, Kelvin Kai-Wang To, Hin Chu, Jin Yang, Fanfan Xing, Jieling Liu, Cyril Chik-Yan Yip, Rosana Wing-Shan Poon, Hoi-Wah Tsoi, Simon Kam-Fai Lo, Kwok-Hung Chan, Vincent Kwok-Man Poon, Wan-Mui Chan, Jonathan Daniel Ip, Jian-Piao Cai, Vincent Chi-Chung Cheng, Honglin Chen, Christopher Kim-Ming Hui, Kwok-Yung Yuen: A familial cluster of pneumonia associated with the 2019 novel coronavirus indicating person-to-person transmission: a study of a family cluster. In: The Lancet. 24. Januar 2020, doi:10.1016/S0140-6736(20)30154-9 (englisch).
- ↑ a b c d e Peng Zhou, Xing-Lou Yang, Xian-Guang Wang, Ben Hu, Lei Zhang, Wei Zhang, Hao-Rui Si, Yan Zhu, Bei Li, Chao-Lin Huang, Hui-Dong Chen, Jing Chen, Yun Luo, Hua Guo, Ren-Di Jiang, Mei-Qin Liu, Ying Chen, Xu-Rui Shen, Xi Wang, Xiao-Shuang Zheng, Kai Zhao, Quan-Jiao Chen, Fei Deng, Lin-Lin Liu, Bing Yan, Fa-Xian Zhan, Yan-Yi Wang, Geng-Fu Xiao, Zheng-Li Shi: A pneumonia outbreak associated with a new coronavirus of probable bat origin. In: Nature. 3. Februar 2020, doi:10.1038/s41586-020-2012-7 (englisch, dieser Artikel wurde am 23. Januar 2020 vorab ohne Peer-Review auf bioRxiv veröffentlicht).
- ↑ D. Paraskevis, E. G. Kostaki, G. Magiorkinis, G. Panayiotakopoulos, G. Sourvinos, S. Tsiodras: Full-genome evolutionary analysis of the novel corona virus (2019-nCoV) rejects the hypothesis of emergence as a result of a recent recombination event. In: bioRxiv. 27. Januar 2020, doi:10.1101/2020.01.26.920249 (englisch).
- ↑ a b c d Roujian Lu, Xiang Zhao, Juan Li, Peihua Niu, Bo Yang, Honglong Wu, Wenling Wang, Hao Song, Baoying Huang, Na Zhu, Yuhai Bi, Xuejun Ma, Faxian Zhan, Liang Wang, Tao Hu, Hong Zhou, Zhenhong Hu, Weimin Zhou, Li Zhao, Jing Chen, Yao Meng, Ji Wang, Yang Lin, Jianying Yuan, Zhihao Xie, Jinmin Ma, William J Liu, Dayan Wang, Wenbo Xu, Edward C Holmes, George F Gao, Guizhen Wu, Weijun Chen, Weifeng Shi, Wenjie Tan: Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding. In: The Lancet. 29. Januar 2020, doi:10.1016/S0140-6736(20)30251-8 (englisch).
- ↑ Genom-Analysen klären Herkunft von 2019-nCoV. In: Website Deutsches Ärzteblatt. 30. Januar 2020, abgerufen am 9. Februar 2020.
- ↑ a b c 2019-nCoV: Erste Bilder vom Virus und Erkenntnisse zum klinischen Verlauf. In: Website Deutsches Ärzteblatt. 27. Januar 2020, abgerufen am 11. Februar 2020.
- ↑ a b Ewen Callaway, David Cyranoski: Why snakes probably aren’t spreading the new China virus – One genetic analysis suggests reptilian reservoir — but researchers doubt that the coronavirus could have originated in animals other than birds or mammals. In: Nature. 23. Januar 2020, doi:10.1038/d41586-020-00180-8 (englisch).
- ↑ Wei Ji, Wei Wang, Xiaofang Zhao, Junjie Zai, Xingguang Li: Homologous recombination within the spike glycoprotein of the newly identified coronavirus may boost cross‐species transmission from snake to human. In: Journal of Medical Virology. 22. Januar 2020, doi:10.1002/jmv.25682 (englisch).
- ↑ Researchers trace coronavirus outbreak in China to snakes. In: Website EurekAlert! 22. Januar 2020, abgerufen am 26. Januar 2020 (englisch).
- ↑ Coronavirus: How worried should we be? BBC News, 27. Januar 2020, abgerufen am 27. Januar 2020 (englisch).
- ↑ David Cyranoski: Did pangolins spread the China coronavirus to people? In: Nature. 7. Februar 2020, doi:10.1038/d41586-020-00364-2 (englisch).
- ↑ a b Informationen der BAuA: Neuartiges Virus SARS-CoV-2 (bislang 2019-nCoV) durch den ABAS in Risikogruppe 3 eingestuft und Empfehlungen zur Labordiagnostik gegeben. In: Website der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA). 19. Februar 2020, abgerufen am 23. Februar 2020.
- ↑ a b c Beschluss 1/2020 des ABAS vom 19.2.2020 und Begründung zur vorläufigen Einstufung des Virus SARS-CoV-2 in Risikogruppe 3 und Empfehlungen zu nicht gezielten Tätigkeiten (Labordiagnostik) und gezielten Tätigkeiten mit SARS-CoV-2. (PDF; 140 kB) In: Website der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin. 19. Februar 2020, abgerufen am 23. Februar 2020.
- ↑ Interim Laboratory Biosafety Guidelines for Handling and Processing Specimens Associated with Coronavirus Disease 2019 (COVID-19). In: Website der US-amerikanischen Centers for Disease Control and Prevention (CDC). 16. Februar 2020, abgerufen am 23. Februar 2020 (englisch).
- ↑ a b c d Zhangkai J. Cheng, Jing Shan: 2019 Novel coronavirus: where we are and what we know. In: Infection. 18. Februar 2020, doi:10.1007/s15010-020-01401-y (englisch).
- ↑ a b c How COVID-19 Spreads. In: Website der US-amerikanischen Centers for Disease Control and Prevention (CDC). 17. Februar 2020, abgerufen am 23. Februar 2020 (englisch).
- ↑ Michelle L. Holshue, Chas DeBolt et al. for the Washington State 2019-nCoV Case Investigation Team: First Case of 2019 Novel Coronavirus in the United States. In: The New England Journal of Medicine. 31. Januar 2020, doi:10.1056/NEJMoa2001191 (englisch).
- ↑ a b c Lirong Zou, Feng Ruan, Mingxing Huang, Lijun Liang, Huitao Huang, Zhongsi Hong, Jianxiang Yu, Min Kang, Yingchao Song, Jinyu Xia, Qianfang Guo, Tie Song, Jianfeng He, Hui-Ling Yen, Malik Peiris, Jie Wu: SARS-CoV-2 Viral Load in Upper Respiratory Specimens of Infected Patients. In: The New England Journal of Medicine. 19. Februar 2020, doi:10.1056/NEJMc2001737 (englisch).
- ↑ a b Günter Kampf, Daniel Todt, Stephanie Pfaender, Eike Steinmann: Persistence of coronaviruses on inanimate surfaces and its inactivation with biocidal agents. In: The Journal of Hospital Infection. 6. Februar 2020, doi:10.1016/j.jhin.2020.01.022 (englisch).
- ↑ Meike Drießen: Wie lang Coronaviren auf Flächen überleben und wie man sie inaktiviert. In: Website Informationsdienst Wissenschaft (idw). 7. Februar 2020, abgerufen am 8. Februar 2020.
- ↑ Huijun Chen, Juanjuan Guo, Chen Wang, Fan Luo, Xuechen Yu, Wei Zhang, Jiafu Li, Dongchi Zhao, Dan Xu, Qing Gong, Jing Liao, Huixia Yang, Wei Hou, Yuanzhen Zhang: Clinical characteristics and intrauterine vertical transmission potential of COVID-19 infection in nine pregnant women: a retrospective review of medical records. In: The Lancet. 12. Februar 2020, doi:10.1016/S0140-6736(20)30360-3 (englisch).
- ↑ Min Wei, Jingping Yuan, Yu Liu, Tao Fu, Xue Yu, Zhi-Jiang Zhang: Novel Coronavirus Infection in Hospitalized Infants Under 1 Year of Age in China. In: Journal of the American Medical Association. 14. Februar 2020, doi:10.1001/jama.2020.2131 (englisch).
- ↑ a b Li Q, Guan X, Wu P, Wang X, Zhou L, Tong Y, Ren R, Leung KSM, Lau EHY, Wong JY, Xing X, Xiang N, Wu Y, Li C, Chen Q, Li D, Liu T, Zhao J, Li M, Tu W, Chen C, Jin L, Yang R, Wang Q, Zhou S, Wang R, Liu H, Luo Y, Liu Y, Shao G, Li H, Tao Z, Yang Y, Deng Z, Liu B, Ma Z, Zhang Y, Shi G, Lam TTY, Wu JTK, Gao GF, Cowling BJ, Yang B, Leung GM, Feng Z.: Early Transmission Dynamics in Wuhan, China, of Novel Coronavirus–Infected Pneumonia. In: The New England Journal of Medicine. 29. Januar 2020, doi:10.1056/NEJMoa2001316 (englisch).
- ↑ Joseph T. Wu, Kathy Leung, Gabriel M. Leung: Nowcasting and forecasting the potential domestic and international spread of the 2019-nCoV outbreak originating in Wuhan, China: a modelling study. In: The Lancet. 31. Januar 2020, doi:10.1016/S0140-6736(20)30260-9 (englisch).
- ↑ Tao Liu, Jianxiong Hu, Jianpeng Xiao, Guanhao He, Min Kang, Zuhua Rong, Lifeng Lin, Haojie Zhong, Qiong Huang, Aiping Deng, Weilin Zeng, Xiaohua Tan, Siqing Zeng, Zhihua Zhu, Jiansen Li, Dexin Gong, Donghua Wan, Shaowei Chen, Lingchuan Guo, Yan Li, Limei Sun, Wenjia Liang, Tie Song, Jianfeng He, Wenjun Ma: Time-varying transmission dynamics of Novel Coronavirus Pneumonia in China. In: bioRxiv. 13. Februar 2020, doi:10.1101/2020.01.25.919787 (englisch).
- ↑ Ying Liu, Albert A. Gayle, Annelies Wilder-Smith, Joacim Rocklöv: The reproductive number of COVID-19 is higher compared to SARS coronavirus. In: Journal of Travel Medicine. 13. Februar 2020, S. taaa021, doi:10.1093/jtm/taaa021 (englisch).
- ↑ Novel Coronavirus (2019-nCoV). (PDF; 0,9 MB) Situation Report – 18. WHO, 7. Februar 2020, abgerufen am 8. Februar 2020.
- ↑ a b Nanshan Chen, Min Zhou, Xuan Dong, Jieming Qu, Fengyun Gong, Yang Han, Yang Qiu, Jingli Wang, Ying Liu, Yuan Wei, Jia'an Xia, Ting Yu, Xinxin Zhang, L. i. Zhang: Epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in Wuhan, China: a descriptive study. In: The Lancet. 30. Januar 2020, doi:10.1016/S0140-6736(20)30211-7.
- ↑ Sung-mok Jung, Andrei R. Akhmetzhanov, Katsuma Hayashi, Natalie M. Linton, Yichi Yang, Baoyin Yuan, Tetsuro Kobayashi, Ryo Kinoshita, Hiroshi Nishiura: Real time estimation of the risk of death from novel coronavirus (2019-nCoV) infection: Inference using exported cases. In: medRxiv. 2. Februar 2020, doi:10.1101/2020.01.29.20019547 (englisch).
- ↑ D. Wang, B. Hu, C. Hu et al.: Clinical Characteristics of 138 Hospitalized Patients With 2019 Novel Coronavirus–Infected Pneumonia in Wuhan, China. In: Journal of the American Medical Association. 7. Februar 2020, doi:10.1001/jama.2020.1585 (englisch).
- ↑ a b Yan Bai, Lingsheng Yao, Tao Wei, Fei Tian, Dong-Yan Jin, Lijuan Chen, Meiyun Wang: Presumed Asymptomatic Carrier Transmission of COVID-19. In: Journal of the American Medical Association. 21. Februar 2020, doi:10.1001/jama.2020.2565 (englisch).
- ↑ Coronavirus disease 2019 (COVID-19). (PDF; 1,1 MB) Situation Report – 30. WHO, 19. Februar 2020, abgerufen am 23. Februar 2020.
- ↑ a b Camilla Rothe, Mirjam Schunk, Peter Sothmann, Gisela Bretzel, Guenter Froeschl, Claudia Wallrauch, Thorbjörn Zimmer, Verena Thiel, Christian Janke, Wolfgang Guggemos, Michael Seilmaier, Christian Drosten, Patrick Vollmar, Katrin Zwirglmaier, Sabine Zange, Roman Wölfel, Michael Hoelscher: Transmission of 2019-nCoV Infection from an Asymptomatic Contact in Germany. In: The New England Journal of Medicine. 30. Januar 2020, doi:10.1056/NEJMc2001468 (englisch).
- ↑ a b Kai Kupferschmidt: Study claiming new coronavirus can be transmitted by people without symptoms was flawed. In: Science. 3. Februar 2020 (englisch, sciencemag.org).
- ↑ Sandra Ciesek et al.: Evidence of SARS-CoV-2 Infection in Returning Travelers from Wuhan, China. NEJM, 18. Februar 2018 doi:10.1056/NEJMc2001899
- ↑ Jing Liu : Asymptomatic cases in a family cluster with SARS-CoV-2 infection. Lancet, 19. Februar 2020, doi:10.1016/S1473-3099(20)30114-6
- ↑ 2019-nCoV offenbar schon bei sehr leichten Symptomen übertragbar. In: Website Deutsches Ärzteblatt. 4. Februar 2020, abgerufen am 5. Februar 2020.
- ↑ Gesundheitsdienst, Stand 27. Januar 2020. (PDF) Auswärtiges Amt, abgerufen am 27. Januar 2020.
- ↑ a b c Huang C, Wang Y, Li X, Ren L, Zhao J, Hu Y, Zhang L, Fan G, Xu J, Gu X, Cheng Z, Yu T, Xia J, Wei Y, Wu W, Xie X, Yin W, Li H, Liu M, Xiao Y, Gao H, Guo L, Xie J, Wang G, Jiang R, Gao Z, Jin Q, Wang J, Cao B: Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China. In: The Lancet. 24. Januar 2020, doi:10.1016/S0140-6736(20)30183-5 (englisch).
- ↑ a b c 2019-nCoV: Verdachtsabklärung und Maßnahmen – Orientierungshilfe für Ärztinnen und Ärzte. (PDF; 59 kB) In: Website des Robert Koch-Instituts. 23. Januar 2020, abgerufen am 26. Januar 2020.
- ↑ a b Jef Akst: Australian Lab Cultures New Coronavirus as Infections Climb. In: Website The-Scientist. 29. Januar 2020, abgerufen am 4. Februar 2020 (englisch).
- ↑ a b Kathrin Strobel: Was tun bei Verdacht auf Coronavirus 2019-nCoV? In: medical-tribune.de. Medical Tribune Deutschland, 28. Januar 2020, abgerufen am 28. Januar 2020.
- ↑ Xingzhi Xie, Zheng Zhong, Wei Zhao, Chao Zheng, Fei Wang, Jun Liu: Chest CT for Typical 2019-nCoV Pneumonia: Relationship to Negative RT-PCR Testing. In: Radiology. 12. Februar 2020, S. 1–11, doi:10.1148/radiol.2020200343 (englisch).
- ↑ a b Victor M. Corman, Olfert Landt, Marco Kaiser, Richard Molenkamp, Adam Meijer, Daniel K. W. Chu, Tobias Bleicker, Sebastian Brünink, Julia Schneider, Marie Luisa Schmidt, Daphne G. J. C. Mulders, Bart L. Haagmans, Bas van der Veer, Sharon van den Brink, Lisa Wijsman, Gabriel Goderski, Jean-Louis Romette, Joanna Ellis, Maria Zambon, Malik Peiris, Herman Goossens, Chantal Reusken, Marion P. G. Koopmans, Christian Drosten: Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. In: Eurosurveillance. Band 25, Nr. 3, 23. Januar 2020, ISSN 1560-7917, S. 1–8, doi:10.2807/1560-7917.ES.2020.25.3.2000045.
- ↑ Novel Coronavirus (2019-nCoV) technical guidance: Laboratory testing for 2019-nCoV in humans. In: Website WHO. Weltgesundheitsorganisation (WHO), 2020, abgerufen am 5. Februar 2020 (englisch).
- ↑ a b c d e Coronavirus outbreak: New test kit can detect the virus in just 15 minutes. In: Website MSN. 10. Februar 2020, abgerufen am 16. Februar 2020 (englisch).
- ↑ a b c Laboratory testing for 2019 novel coronavirus (2019-nCoV) in suspected human cases. In: Website WHO. Weltgesundheitsorganisation (WHO), 17. Januar 2020, abgerufen am 5. Februar 2020 (englisch).
- ↑ Global Initiative on Sharing All Influenza Data (GISAID). In: Website der GISAID (Bundesrepublik Deutschland als Host (Datenbankanbieter)). Abgerufen am 5. Februar 2020 (englisch).
- ↑ CDC Tests for 2019-nCoV. In: Website der US-amerikanischen Centers for Disease Control and Prevention (CDC). 5. Februar 2020, abgerufen am 10. Februar 2020 (englisch).
- ↑ Stephanie Soucheray: CDC sends out nCoV test kits as Wisconsin confirms case. In: Website der University of Minnesota. CIDRAP – Center for Infectious Disease Research and Policy, 5. Februar 2020, abgerufen am 10. Februar 2020 (englisch).
- ↑ New test kits for coronavirus approved in China. In: Website Xinhua. 30. Januar 2020, abgerufen am 10. Februar 2020 (englisch).
- ↑ China's Jiangsu develops rapid test kit for coronavirus. In: Website Xinhua. 31. Januar 2020, abgerufen am 16. Februar 2020 (englisch).
- ↑ Dean Koh: iHealthtech researchers working on Wuhan novel coronavirus (2019-nCoV) detection kit. In: Website mobi health news der National University of Singapore. 6. Februar 2020, abgerufen am 16. Februar 2020 (englisch).
- ↑ Hinweise zur Testung von Patienten auf Infektion mit dem in Wuhan entdeckten neuartigen Coronavirus (2019-nCoV). In: Website des Robert Koch-Instituts (RKI). 3. Februar 2020, abgerufen am 5. Februar 2020.
- ↑ Institut Pasteur isolates strains of coronavirus 2019-nCoV detected in France. In: Website EurekAlert! 31. Januar 2020, abgerufen am 4. Februar 2020 (englisch).
- ↑ Erstmals nCoV in Deutschland in Zellkultur isoliert. In: Website Institut für Mikrobiologie der Bundeswehr. 31. Januar 2020, abgerufen am 3. Februar 2020.
- ↑ CDC Grows 2019-nCoV Virus in Cell Culture. In: Website der US-amerikanischen Centers for Disease Control and Prevention (CDC). 6. Februar 2020, abgerufen am 10. Februar 2020 (englisch).
- ↑ Zhijian Xu, Cheng Peng, Yulong Shi, Zhengdan Zhu, Kaijie Mu, Xiaoyu Wang, Weiliang Zhu: Nelfinavir was predicted to be a potential inhibitor of 2019 nCov main protease by an integrative approach combining homology modelling, molecular docking and binding free energy calculation. In: bioRxiv. 28. Januar 2020, S. 2020.01.27.921627, doi:10.1101/2020.01.27.921627 (englisch).
- ↑ Jon Cohen: Can an anti-HIV combination or other existing drugs outwit the new coronavirus? In: Science. 27. Januar 2020, doi:10.1126/science.abb0659 (englisch).
- ↑ Xin Liu, Xiu-Jie Wang: Potential inhibitors for 2019-nCoV coronavirus M protease from clinically approved medicines. In: bioRxiv. 29. Januar 2020, doi:10.1101/2020.01.29.924100 (englisch).
- ↑ Catharine I. Paules, Hilary D. Marston, Anthony S. Fauci: Coronavirus Infections – More Than Just the Common Cold. In: Journal of the American Medical Association. 23. Januar 2020, doi:10.1001/jama.2020.0757, PMID 31971553 (englisch).
- ↑ 上海药物所和上海科技大学联合发现一批可能对新型肺炎有治疗作用的老药和中药. Chinese Academy of Sciences, 25. Januar 2020 (chinesisch).
- ↑ Fu-Sheng Wang, Chao Zhang: What to do next to control the 2019-nCoV epidemic? In: The Lancet. Band 395, Nr. 10222, 8. Februar 2020, S. 391–393, doi:10.1016/S0140-6736(20)30300-7 (englisch).
- ↑ Wang Xiaoyu: Drugs with potential against coronavirus in human trials. In: Website China Daily. 15. Februar 2020, abgerufen am 16. Februar 2020 (englisch).
- ↑ Zhang Yangfei: First antiviral drug approved to fight coronavirus. In: Website China Daily. 17. Februar 2020, abgerufen am 17. Februar 2020 (englisch).
- ↑ Wang Xiaoyu: Progress seen in epidemic fight. In: Website China Daily. 20. Februar 2020, abgerufen am 19. Februar 2020 (englisch).
- ↑ Wang Xiaodong: Plasma therapy could lead to development of vaccine for novel coronavirus pneumonia. In: Website China Daily. 17. Februar 2020, abgerufen am 18. Februar 2020 (englisch).
- ↑ Zhang Zhihao: More plasma needed from recovered coronavirus patients, experts say. In: Website China Daily. 17. Februar 2020, abgerufen am 18. Februar 2020 (englisch).
- ↑ Jinnong Zhang, Luqian Zhou, Yuqiong Yang, Wei Peng, Wenjing Wang, Xuelin Chen: Therapeutic and triage strategies for 2019 novel coronavirus disease in fever clinics. In: The Lancet Respiratory Medicine. 13. Februar 2020, doi:10.1016/S2213-2600(20)30071-0 (englisch).
- ↑ Jon Cohen: Scientists are moving at record speed to create new coronavirus vaccines—but they may come too late. In: Science. 27. Januar 2020, doi:10.1126/science.abb0612 (englisch).
- ↑ a b Sechste Infektion bestätigt: Kind in Bayern an Coronavirus erkrankt. In: Website tagesschau.de. 31. Januar 2020, abgerufen am 9. Februar 2020.
- ↑ Florian Schumann: Deutscher Coronavirus-Experte: „Wir müssen uns auf eine Pandemie einstellen“. In: Website Der Tagesspiegel. 13. Februar 2020, abgerufen am 15. Februar 2020.
- ↑ Prof. Dr. Christian Drosten Direktor des Instituts für Virologie, Charité – Universitätsmedizin Berlin: „Wie gefährlich wird das neue Coronavirus?“
- ↑ Liste der vom Robert Koch-Institut geprüften und anerkannten Desinfektionsmittel und -verfahren. In: Website des Robert Koch-Instituts. 6. September 2018, abgerufen am 8. Februar 2020.
- ↑ Empfehlungen des Robert Koch-Institutes für die Hygienemaßnahmen und Infektionskontrolle bei Patienten mit bestätigter Infektion durch 2019-nCoV. In: Website des Robert Koch-Instituts. 7. Februar 2020, abgerufen am 8. Februar 2020.
- ↑ Amy Qin, Javier C. Hernández: China Reports First Death From New Virus. The New York Times, 10. Januar 2020, abgerufen am 14. Januar 2020 (englisch).
- ↑ Novel Coronavirus – China. WHO, 12. Januar 2020, abgerufen am 19. Januar 2020 (englisch).
- ↑ Wuhan pneumonia outbreak: First case reported outside China. BBC News, 14. Januar 2020, abgerufen am 14. Januar 2020 (englisch).
- ↑ Neue Lungenkrankheit auch in Thailand. Deutsche Welle, 13. Januar 2020, abgerufen am 14. Januar 2020.
- ↑ China coronavirus: Number of cases jumps as virus spreads to new cities. 20. Januar 2020, abgerufen am 20. Januar 2020 (englisch).
- ↑ a b c d Novel Coronavirus (2019-nCoV) situation reports; Coronavirus disease (COVID-2019) situation reports. WHO (englisch, abgerufen im Januar bis Februar 2020).
- ↑ Pressemitteilung: Bestätigter Coronavirus-Fall in Bayern – Infektionsschutzmaßnahmen laufen. In: Website Bayerisches Staatsministerium für Gesundheit und Pflege. 27. Januar 2020, abgerufen am 9. Februar 2020.
- ↑ Coronavirus: Drei weitere Fälle in Bayern. In: Website tagesschau.de. 29. Januar 2020, abgerufen am 9. Februar 2020.
- ↑ Pressemitteilung: 7. Fall in München Klinik Schwabing in stabilem Zustand. In: Website Bayerisches Staatsministerium für Gesundheit und Pflege. 27. Januar 2020, abgerufen am 9. Februar 2020.
- ↑ WHO ruft wegen Coronavirus weltweite Notlage aus. Süddeutsche Zeitung, 30. Januar 2020, abgerufen am 30. Januar 2020.
- ↑ Verordnung über die Ausdehnung der Meldepflicht nach § 6 Absatz 1 Satz 1 Nummer 1 und § 7 Absatz 1 Satz 1 des Infektionsschutzgesetzes auf Infektionen mit dem erstmals im Dezember 2019 in Wuhan/Volksrepublik China aufgetretenen neuartigen Coronavirus („2019-nCoV“). Bundesministerium der Justiz und für Verbraucherschutz, Bundesamt für Justiz (Deutschland), 30. Januar 2020, abgerufen am 1. Februar 2020.