„SARS-CoV-2“ – Versionsunterschied
| [gesichtete Version] | [gesichtete Version] |
Keine Bearbeitungszusammenfassung |
Leyo (Diskussion | Beiträge) K format |
||
| Zeile 30: | Zeile 30: | ||
}} |
}} |
||
Das [[Viren|Virus]] '''SARS-CoV-2''' (Abk. für {{enS|severe acute respiratory syndrome coronavirus type 2}})<ref name="rki_11122020">{{Internetquelle |autor= |url=https://www.rki.de/DE/Content/InfAZ/N/Neuartiges_Coronavirus/Steckbrief.html?nn=13490888#doc13776792bodyText1 |titel=Epidemiologischer Steckbrief zu SARS-CoV-2 und COVID-19 → Erreger |titelerg= (Stand: 11. Dezember 2020) |werk=Website des Robert Koch-Instituts: [https://www.rki.de/DE/Home/homepage_node.html COVID-19 in Deutschland] |hrsg=[[Robert Koch-Institut]] |datum=2020-12-11|abruf=2021-01-06 |kommentar=}}</ref>, auch als ''Schweres akutes Atemwegssyndrom-Coronavirus-Typ 2'' bezeichnet, umgangssprachlich (neuartiges) '''Coronavirus''' genannt, ist ein dem [[SARS-Coronavirus|SARS-Erreger]] ähnliches neues [[Betacoronavirus|Beta-Coronavirus]] mit wahrscheinlich [[Zoonose|zoonotischem Ursprung]].<ref name="Dorp2020">{{ |
Das [[Viren|Virus]] '''SARS-CoV-2''' (Abk. für {{enS|severe acute respiratory syndrome coronavirus type 2}})<ref name="rki_11122020">{{Internetquelle |autor= |url=https://www.rki.de/DE/Content/InfAZ/N/Neuartiges_Coronavirus/Steckbrief.html?nn=13490888#doc13776792bodyText1 |titel=Epidemiologischer Steckbrief zu SARS-CoV-2 und COVID-19 → Erreger |titelerg= (Stand: 11. Dezember 2020) |werk=Website des Robert Koch-Instituts: [https://www.rki.de/DE/Home/homepage_node.html COVID-19 in Deutschland] |hrsg=[[Robert Koch-Institut]] |datum=2020-12-11|abruf=2021-01-06 |kommentar=}}</ref>, auch als ''Schweres akutes Atemwegssyndrom-Coronavirus-Typ 2'' bezeichnet, umgangssprachlich (neuartiges) '''Coronavirus''' genannt, ist ein dem [[SARS-Coronavirus|SARS-Erreger]] ähnliches neues [[Betacoronavirus|Beta-Coronavirus]] mit wahrscheinlich [[Zoonose|zoonotischem Ursprung]].<ref name="Dorp2020">{{Literatur |Autor=Lucy van Dorp, Mislav Acman, Damien Richard, Liam P. Shaw, Charlotte E. Ford |Titel=Emergence of genomic diversity and recurrent mutations in SARS-CoV-2 |Sammelwerk=[[Infection, Genetics and Evolution]] |Band=83 |Datum=2020-09 |DOI=10.1016/j.meegid.2020.104351 |PMC=7199730 |PMID=32387564 |Seiten=104351}}</ref><ref name="rki_11122020"/> Das Virus wurde Anfang 2020 als Auslöser von [[COVID-19]] identifiziert.<ref name="rki_11122020"/> |
||
COVID-19 trat laut Chinas Regierung erstmals Ende 2019 in der chinesischen Stadt [[Wuhan]] als „Lungenkrankheit unbekannter [[Pathogenese|Genese]]“ auf.<ref name="entdeckung_in_Wuhan" /> Die [[Weltgesundheitsorganisation]] (WHO) nannte COVID-19 am 30. Januar 2020 eine „[[Gesundheitliche Notlage internationaler Tragweite|gesundheitliche Notlage von internationaler Tragweite]]“. Da die Erkrankung sich weltweit ausbreitete, wurde COVID-19 am 11. März 2020 als [[Pandemie]] eingestuft.<ref>Florian Rötzer: [https://www.heise.de/tp/features/WHO-ruft-international-Notlage-aus-4650512.html ''WHO ruft international Notlage aus''] 30. Januar 2020.</ref><ref>{{Internetquelle |url=https://www.rki.de/DE/Content/InfAZ/N/Neuartiges_Coronavirus/Risikobewertung.html |titel=Coronavirus SARS-CoV-2: Risikobewertung zu COVID-19 |titelerg=„Die weltweite Ausbreitung von [[COVID-19]] wurde am 11.03.2020 von der WHO zu einer Pandemie erklärt.“ |werk=Website des RKI |hrsg=[[Robert Koch-Institut]] (RKI) |datum=2020-05-26 |abruf=2020-05-26}}</ref> |
COVID-19 trat laut Chinas Regierung erstmals Ende 2019 in der chinesischen Stadt [[Wuhan]] als „Lungenkrankheit unbekannter [[Pathogenese|Genese]]“ auf.<ref name="entdeckung_in_Wuhan" /> Die [[Weltgesundheitsorganisation]] (WHO) nannte COVID-19 am 30. Januar 2020 eine „[[Gesundheitliche Notlage internationaler Tragweite|gesundheitliche Notlage von internationaler Tragweite]]“. Da die Erkrankung sich weltweit ausbreitete, wurde COVID-19 am 11. März 2020 als [[Pandemie]] eingestuft.<ref>Florian Rötzer: [https://www.heise.de/tp/features/WHO-ruft-international-Notlage-aus-4650512.html ''WHO ruft international Notlage aus''] 30. Januar 2020.</ref><ref>{{Internetquelle |url=https://www.rki.de/DE/Content/InfAZ/N/Neuartiges_Coronavirus/Risikobewertung.html |titel=Coronavirus SARS-CoV-2: Risikobewertung zu COVID-19 |titelerg=„Die weltweite Ausbreitung von [[COVID-19]] wurde am 11.03.2020 von der WHO zu einer Pandemie erklärt.“ |werk=Website des RKI |hrsg=[[Robert Koch-Institut]] (RKI) |datum=2020-05-26 |abruf=2020-05-26}}</ref> |
||
| Zeile 55: | Zeile 55: | ||
Aus Modellierungen aufgrund der Untersuchung der Veränderungen des Erbmaterials RNA des Virus wird als erstes Auftreten des Virus als wahrscheinlich zwischen Oktober und Anfang Dezember eingegrenzt. Die Verbreitungsmuster der verschiedenen unterscheidbaren Virusmutationen spricht für eine massenhafte weltweite Ausbreitung des Virus durch eine Vielzahl von verschiedenen Ausbreitungsereignissen.<ref>François Balloux et al.: ''Emergence of genomic diversity and recurrent mutations in SARS-CoV-2.'' 5. Mai 2020 [[doi:10.1016/j.meegid.2020.104351]], frei zugänglicher Volltext unter: [https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7199730/ gleicher Artikel in NCBI-NIH-Artikel-Datenbank]</ref> |
Aus Modellierungen aufgrund der Untersuchung der Veränderungen des Erbmaterials RNA des Virus wird als erstes Auftreten des Virus als wahrscheinlich zwischen Oktober und Anfang Dezember eingegrenzt. Die Verbreitungsmuster der verschiedenen unterscheidbaren Virusmutationen spricht für eine massenhafte weltweite Ausbreitung des Virus durch eine Vielzahl von verschiedenen Ausbreitungsereignissen.<ref>François Balloux et al.: ''Emergence of genomic diversity and recurrent mutations in SARS-CoV-2.'' 5. Mai 2020 [[doi:10.1016/j.meegid.2020.104351]], frei zugänglicher Volltext unter: [https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7199730/ gleicher Artikel in NCBI-NIH-Artikel-Datenbank]</ref> |
||
Als [[Indexpatient|Patient null]] wird ein 55-jähriger Mann aus der Provinz [[Hubei]] vermutet, der sich bereits am 17. November 2019 infiziert haben könnte.<ref>Aylin Woodward: [https://www.businessinsider.com/coronavirus-did-not-jump-wuhan-market-chinese-cdc-says-2020-5 ''The Chinese CDC now says the coronavirus didn't jump to people at the Wuhan wet market — instead, it was the site of a superspreader event''], auf [[Business Insider]] vom 29. Mai 2020</ref><!-- Probeweise Löschung siehe Disk „Entdeckungsgeschichte“ zitiert zweifelhafte Studie In Frankreich wurde durch retrospektive Analyse ein Fall im Dezember 2019 nahe Paris nachgewiesen.<ref> |
Als [[Indexpatient|Patient null]] wird ein 55-jähriger Mann aus der Provinz [[Hubei]] vermutet, der sich bereits am 17. November 2019 infiziert haben könnte.<ref>Aylin Woodward: [https://www.businessinsider.com/coronavirus-did-not-jump-wuhan-market-chinese-cdc-says-2020-5 ''The Chinese CDC now says the coronavirus didn't jump to people at the Wuhan wet market — instead, it was the site of a superspreader event''], auf [[Business Insider]] vom 29. Mai 2020</ref><!-- Probeweise Löschung siehe Disk „Entdeckungsgeschichte“ zitiert zweifelhafte Studie In Frankreich wurde durch retrospektive Analyse ein Fall im Dezember 2019 nahe Paris nachgewiesen.<ref>{{Literatur |Autor=A. Deslandes, V. Berti, Y. Tandjaoui-Lambotte, Chakib Alloui, E. Carbonnelle |Titel=SARS-CoV-2 was already spreading in France in late December 2019 |Sammelwerk=International Journal of Antimicrobial Agents |Band=55 |Nummer=6 |Datum=2020-06 |DOI=10.1016/j.ijantimicag.2020.106006 |Seiten=106006}}</ref> Der Patient hatte keinerlei Verbindungen zu China/Wuhan, seine Frau arbeitete aber nahe dem Flughafen in einem Supermarkt. Einen noch früheren Verdacht auf Patientin null gibt es im [[Elsass]] (Frankreich) vom 16. November 2019.<ref>{{Internetquelle |url=https://www.fondation-diaconat.fr/images/Presse/2020/CP-HAS-Imagerie-mdicale-7-mai-2020.pdf |titel=Pressemitteilung |hrsg=Albert Schweitzer Klinik Colmar |datum=2020-05-07 |abruf=2020-06-07 |format=PDF}}</ref> Ende Probeweise Löschung siehe Disk „Entdeckungsgeschichte“ zitiert zweifelhafte Studie--> Nicht alle frühen COVID-19-Fälle können mit dem [[Wet market|Markt]] in Verbindung gebracht werden; die Historie des [[Ausbruch (Epidemiologie)|Ausbruchs]] ist wohl komplizierter als zunächst angenommen.<ref name="SciTechDaily:UoS" /> In einer Vorab-Publikation aus dem Herbst konnte aufgrund umfangreichen Genomvergleichs eine vermutliche RNA-Sequenz der Ausgangsform („[[Stammvater]]“, en. progenitor: proCoV2) ermittelt werden, die (wie zu erwarten) vom Genom der real existierenden Referenzform etwas abweicht. Aus den Daten lässt sich vermuten, dass dieses Virus bereits einige Wochen vor den Fällen im Dezember 2019 Menschen infiziert hat. Die Forscher lokalisieren dieses Virus und seine unmittelbaren Nachfolger in China.<ref>Sudhir Kumar, Qiqing Tao, Steven Weaver, Maxwell Sanderford, Marcos A. Caraballo-Ortiz, Sudip Sharma, Sergei L. K. Pond, Sayaka Miura: [https://www.biorxiv.org/content/10.1101/2020.09.24.311845v2.article-info An evolutionary portrait of the progenitor SARS-CoV-2 and its dominant offshoots in COVID-19 pandemic], auf: bioRxiv vom 29. September 2020, [[doi:10.1101/2020.09.24.311845]] (Preprint), dazu:<br />Joseph Caspermeyer: [https://scitechdaily.com/covid-19-patient-zero-data-analysis-identifies-the-mother-of-all-sars-cov-2-genomes/ ''COVID-19 Patient Zero: Data Analysis Identifies the “Mother” of All SARS-CoV-2 Genomes''], auf: SciTechDaily vom 7. November 2020</ref> |
||
Am 10. November 2019 begab sich eine damals 25-jährige Frau wegen eines Hautausschlags in [[Mailand]] in ärztliche Behandlung. Anfang 2021 konnte durch [[Biopsie|Untersuchung]] einer seinerzeit entnommenen Hautprobe der Nachweis erbracht werden, dass die Frau schon damals mit SARS-CoV-2 infiziert war. Sie ist damit aktuell der erste nachgewiesene Fall auf der Welt. Ob sie nicht nur in Italien, sondern womöglich weltweit Patient null ist, muss noch geklärt werden.<ref>{{Internetquelle |url=https://www.spiegel.de/wissenschaft/corona-erster-fall-koennte-aus-italien-stammen-aus-dem-november-2019-a-8757a6d9-48be-4f20-836e-de4ba14db2ba |titel=Bislang ältester Nachweis einer Corona-Infektion könnte aus Italien stammen |werk=[[Der Spiegel]] |datum=2021-01-11| zugriff=2021-01-12 }}</ref> |
Am 10. November 2019 begab sich eine damals 25-jährige Frau wegen eines Hautausschlags in [[Mailand]] in ärztliche Behandlung. Anfang 2021 konnte durch [[Biopsie|Untersuchung]] einer seinerzeit entnommenen Hautprobe der Nachweis erbracht werden, dass die Frau schon damals mit SARS-CoV-2 infiziert war. Sie ist damit aktuell der erste nachgewiesene Fall auf der Welt. Ob sie nicht nur in Italien, sondern womöglich weltweit Patient null ist, muss noch geklärt werden.<ref>{{Internetquelle |url=https://www.spiegel.de/wissenschaft/corona-erster-fall-koennte-aus-italien-stammen-aus-dem-november-2019-a-8757a6d9-48be-4f20-836e-de4ba14db2ba |titel=Bislang ältester Nachweis einer Corona-Infektion könnte aus Italien stammen |werk=[[Der Spiegel]] |datum=2021-01-11| zugriff=2021-01-12 }}</ref> |
||
| Zeile 243: | Zeile 243: | ||
Laut einer Studie des Friedrich-Loeffler-Instituts (FLI) zeigen [[Rinder]] eine geringe Empfänglichkeit gegenüber SARS-CoV-2.<ref>https://www.agrarheute.com/tier/rind/corona-studie-empfaenglich-rinder-572258 Corona-Studie: Wie empfänglich sind Rinder?</ref> |
Laut einer Studie des Friedrich-Loeffler-Instituts (FLI) zeigen [[Rinder]] eine geringe Empfänglichkeit gegenüber SARS-CoV-2.<ref>https://www.agrarheute.com/tier/rind/corona-studie-empfaenglich-rinder-572258 Corona-Studie: Wie empfänglich sind Rinder?</ref> |
||
Während im Labor infizierte Mäuse offenbar keine Krankheitssymptome entwickeln, war es Y.-C. Wang und Kollegen in China möglich, bei [[Farbmaus#Einsatz in der biomedizinischen Forschung|C57BL/6-Labormäusen]] mit [[CRISPR/Cas9]] das ACE2 der Mäuse (mACE2, murines ACE2) durch das des Menschen (hACE2, humanes ACE2) zu ersetzen. Die hACE2-Mäuse zeigten Virusreplikation von SARS-CoV-2 in ihren Lungen, der Luftröhre und im Gehirn. Auch der Verdauungstrakt war betroffen, so wie es bei manchen menschlichen Patienten beobachtet wird. Sie scheinen damit geeignet, um etwa einen Impfstoff zu testen, bevor er Menschen verabreicht wird;<ref name="Sun2020">Shi-Hui Sun, You-Chun Wang ''et al.'': [https://www.cell.com/cell-host-microbe/fulltext/S1931-3128(20)30302-4 A Mouse Model of SARS-CoV-2 Infection and Pathogenesis], in: Cell Host and Microbe vom 26. Mai 2020, [[doi:10.1016/j.chom.2020.05.020]]</ref><ref>Jacinta Bowler: [https://www.sciencealert.com/scientists-have-found-a-way-to-infect-mice-with-the-coronavirus-here-s-why-that-s-super-important Scientists Find a Way to Infect Mice With Coronavirus. Here's Why That's So Important], auf: science<sup>alert</sup> vom 1. Juni 2020</ref> eine Alternative zur Methode die Wirkung eines Mittels auf künstlich mutierte Sarbecoviren zu testen, wie jüngst bei [[Remdesivir]] und SARS-CoV/SARS-CoV-2 RdRp (altes SARS-Virus mit [[RNA-abhängige RNA-Polymerase|RdRP]]-Gen von SARS-CoV-2) geschehen.<ref name="Pruijssers2020">Andrea J. Pruijssers |
Während im Labor infizierte Mäuse offenbar keine Krankheitssymptome entwickeln, war es Y.-C. Wang und Kollegen in China möglich, bei [[Farbmaus#Einsatz in der biomedizinischen Forschung|C57BL/6-Labormäusen]] mit [[CRISPR/Cas9]] das ACE2 der Mäuse (mACE2, murines ACE2) durch das des Menschen (hACE2, humanes ACE2) zu ersetzen. Die hACE2-Mäuse zeigten Virusreplikation von SARS-CoV-2 in ihren Lungen, der Luftröhre und im Gehirn. Auch der Verdauungstrakt war betroffen, so wie es bei manchen menschlichen Patienten beobachtet wird. Sie scheinen damit geeignet, um etwa einen Impfstoff zu testen, bevor er Menschen verabreicht wird;<ref name="Sun2020">Shi-Hui Sun, You-Chun Wang ''et al.'': [https://www.cell.com/cell-host-microbe/fulltext/S1931-3128(20)30302-4 A Mouse Model of SARS-CoV-2 Infection and Pathogenesis], in: Cell Host and Microbe vom 26. Mai 2020, [[doi:10.1016/j.chom.2020.05.020]]</ref><ref>Jacinta Bowler: [https://www.sciencealert.com/scientists-have-found-a-way-to-infect-mice-with-the-coronavirus-here-s-why-that-s-super-important Scientists Find a Way to Infect Mice With Coronavirus. Here's Why That's So Important], auf: science<sup>alert</sup> vom 1. Juni 2020</ref> eine Alternative zur Methode die Wirkung eines Mittels auf künstlich mutierte Sarbecoviren zu testen, wie jüngst bei [[Remdesivir]] und SARS-CoV/SARS-CoV-2 RdRp (altes SARS-Virus mit [[RNA-abhängige RNA-Polymerase|RdRP]]-Gen von SARS-CoV-2) geschehen.<ref name="Pruijssers2020">{{Literatur |Autor=Andrea J. Pruijssers, Amelia S. George, Alexandra Schäfer, Sarah R. Leist, Lisa E. Gralinksi |Titel=Remdesivir Inhibits SARS-CoV-2 in Human Lung Cells and Chimeric SARS-CoV Expressing the SARS-CoV-2 RNA Polymerase in Mice |Sammelwerk=Cell Reports |Band=32 |Nummer=3 |Datum=2020-07 |DOI=10.1016/j.celrep.2020.107940 |PMC=7340027 |PMID=32668216 |Seiten=107940}}</ref><ref>Tina Hesman Saey: [https://www.sciencenews.org/article/remdesivir-covid-19-coronavirus-pandemic-gilead Remdesivir may work even better against COVID-19 than we thought], auf: ScienceNews vom 13. Juli 2020</ref> |
||
Eine mögliche Gefährdung von [[Meeressäuger|Meeressäugetieren]] (Waale, Robben, Seeotter) ist schwer zu beurteilen.<ref>Jan Osterkamp: [https://www.spektrum.de/news/bedroht-corona-auch-wale-und-robben/1804925 Bedroht Corona auch Wale und Robben?], auf: spektrum.de vom 7. Dezember 2020</ref> |
Eine mögliche Gefährdung von [[Meeressäuger|Meeressäugetieren]] (Waale, Robben, Seeotter) ist schwer zu beurteilen.<ref>Jan Osterkamp: [https://www.spektrum.de/news/bedroht-corona-auch-wale-und-robben/1804925 Bedroht Corona auch Wale und Robben?], auf: spektrum.de vom 7. Dezember 2020</ref> |
||
| Zeile 339: | Zeile 339: | ||
Forscher der ''[[Hong Kong University of Science and Technology]]'' meldeten Anfang Februar 2020 die Entwicklung eines tragbaren Gerätes, mit dem das neuartige Coronavirus innerhalb von 40 Minuten nachweisbar sein soll. Für das im Vergleich zur herkömmlichen qRT-PCR schnellere Verfahren werden modifizierte [[Chip-Thermocycler]] verwendet.<ref name="msn_20200210" /> Auch Forscher des ''Institute for Health Innovation & Technology'' (iHealthtech) an der ''[[National University of Singapore]]'' berichteten im Februar 2020 darüber, eine Schnellmethode zu entwickeln. Sie basiert auf der seit 2018 verwendeten [[enVision]]-Technologie, mit der Nukleinsäuren innerhalb von 30 bis 60 Minuten nachgewiesen werden. Es wird geschätzt, dass bis zur [[Marktreife]] des neuen Testverfahrens noch mehrere Monate benötigt werden.<ref name="NUS_20200131">{{Internetquelle |autor=Dean Koh |url=https://www.mobihealthnews.com/news/asia-pacific/ihealthtech-researchers-working-wuhan-novel-coronavirus-2019-ncov-detection-kit |titel=iHealthtech researchers working on Wuhan novel coronavirus (2019-nCoV) detection kit |werk=Website mobi health news der [[National University of Singapore]] |datum=2020-02-06 |abruf=2020-02-16 |sprache=en}}</ref> |
Forscher der ''[[Hong Kong University of Science and Technology]]'' meldeten Anfang Februar 2020 die Entwicklung eines tragbaren Gerätes, mit dem das neuartige Coronavirus innerhalb von 40 Minuten nachweisbar sein soll. Für das im Vergleich zur herkömmlichen qRT-PCR schnellere Verfahren werden modifizierte [[Chip-Thermocycler]] verwendet.<ref name="msn_20200210" /> Auch Forscher des ''Institute for Health Innovation & Technology'' (iHealthtech) an der ''[[National University of Singapore]]'' berichteten im Februar 2020 darüber, eine Schnellmethode zu entwickeln. Sie basiert auf der seit 2018 verwendeten [[enVision]]-Technologie, mit der Nukleinsäuren innerhalb von 30 bis 60 Minuten nachgewiesen werden. Es wird geschätzt, dass bis zur [[Marktreife]] des neuen Testverfahrens noch mehrere Monate benötigt werden.<ref name="NUS_20200131">{{Internetquelle |autor=Dean Koh |url=https://www.mobihealthnews.com/news/asia-pacific/ihealthtech-researchers-working-wuhan-novel-coronavirus-2019-ncov-detection-kit |titel=iHealthtech researchers working on Wuhan novel coronavirus (2019-nCoV) detection kit |werk=Website mobi health news der [[National University of Singapore]] |datum=2020-02-06 |abruf=2020-02-16 |sprache=en}}</ref> |
||
Eine deutsche Forschergruppe der [[Universität Bonn]] um [[Hendrik Streeck|Hendrick Streeck]] hat im April 2020 die Ergebnisse einer Schnelltestvalidierung vorgestellt<ref>{{Literatur |Autor=M. Döhla, C. Boesecke, B. Schulte, C. Diegmann, E. Sib |Titel=Rapid point-of-care testing for SARS-CoV-2 in a community screening setting shows low sensitivity |Sammelwerk=Public Health |Band=182 | |
Eine deutsche Forschergruppe der [[Universität Bonn]] um [[Hendrik Streeck|Hendrick Streeck]] hat im April 2020 die Ergebnisse einer Schnelltestvalidierung vorgestellt.<ref>{{Literatur |Autor=M. Döhla, C. Boesecke, B. Schulte, C. Diegmann, E. Sib |Titel=Rapid point-of-care testing for SARS-CoV-2 in a community screening setting shows low sensitivity |Sammelwerk=Public Health |Band=182 |Nummer= |Datum=2020-05 |DOI=10.1016/j.puhe.2020.04.009 |PMC=7165286 |PMID=32334183 |Seiten=170–172}}</ref> Hierbei wurde der ''CoV-2 Rapid Test'' im Rahmen eines Screenings der Bevölkerung getestet und mit parallel gewonnenen Proben zur PCR-Diagnostik verglichen. Von insgesamt 49 Personen waren 22 positiv in der PCR; der Schnelltest erkannte jedoch nur acht davon richtig als positiv (Sensitivität 36,4 %). Von den 27 PCR-negativen Personen wurden durch den Schnelltest 24 richtig als negativ diagnostiziert (Spezifität 88,9 %). |
||
==== Antigenschnelltest ==== |
==== Antigenschnelltest ==== |
||
Version vom 14. Januar 2021, 09:49 Uhr
Das Virus SARS-CoV-2 (Abk. für englisch severe acute respiratory syndrome coronavirus type 2)[4], auch als Schweres akutes Atemwegssyndrom-Coronavirus-Typ 2 bezeichnet, umgangssprachlich (neuartiges) Coronavirus genannt, ist ein dem SARS-Erreger ähnliches neues Beta-Coronavirus mit wahrscheinlich zoonotischem Ursprung.[5][4] Das Virus wurde Anfang 2020 als Auslöser von COVID-19 identifiziert.[4]
COVID-19 trat laut Chinas Regierung erstmals Ende 2019 in der chinesischen Stadt Wuhan als „Lungenkrankheit unbekannter Genese“ auf.[6] Die Weltgesundheitsorganisation (WHO) nannte COVID-19 am 30. Januar 2020 eine „gesundheitliche Notlage von internationaler Tragweite“. Da die Erkrankung sich weltweit ausbreitete, wurde COVID-19 am 11. März 2020 als Pandemie eingestuft.[7][8]
Das Virus ist laut Beobachtungen unter dem Rasterelektronenmikroskop zwischen 60 und 140 Nanometern groß und wird in der Regel durch Tröpfchen und Aerosole übertragen.[9] Die Infektion erfolgt in der Regel über enge Kontakte und über Aerosole im gesellschaftlichen Umgang. Bei der weltweiten COVID-19-Pandemie spielten auch plötzliche „explosive“ Übertragungsereignisse, auch Superspreading-Events bezeichnet, vielfach eine große Rolle.[10][11] Ein weiterer Beschleunigungsfaktor für die Ausbreitung des Virus könnte in der Außentemperatur liegen, da sich das Virus nach einer chinesischen Studie bei 4 Grad Celsius als besonders persistent (langfristig aktiv) erwiesen hat.[12] Übertragung durch Schmierinfektion wurde hingegen nicht beobachtet, ist aber nicht ganz auszuschließen.[13][14][15]
Entdeckungsgeschichte
Im Dezember 2019 wurden in der Großstadt Wuhan gehäuft schwere Lungenentzündungen unbekannter Ursache festgestellt.[6]
Am 30. Dezember 2019 informierte der chinesische Arzt Li Wenliang in einer WeChat-Gruppe seine Arztkollegen über sieben Patienten, die wegen Verdachts auf Infektion mit dem SARS-Virus im Zentralkrankenhaus Wuhan behandelt wurden;[16] dafür wurde er von der chinesischen Polizei ermahnt. Li selbst erkrankte später an COVID-19 und starb.[17]
Das Chinesische Zentrum für Krankheitskontrolle und -prävention entsandte am 31. Dezember 2019 ein Team in die Stadt.[18] Am selben Tag wurde das China-Büro der WHO durch die chinesischen Behörden offiziell informiert, dass im Dezember 2019 in Wuhan mehrere Personen an schwerer Lungenentzündung erkrankt waren und dass als deren Ursache ein uncharakterisierter Erreger vermutet werde. Bis zum 3. Januar 2020 wurden der WHO insgesamt 44 Erkrankte gemeldet, darunter Schwerkranke. Da mehrere Erkrankte auf dem örtlichen wet market „Südchinesischer Großhandelsmarkt für Fische und Meeresfrüchte Wuhan“ (chinesisch 武汉华南海鲜批发市场, Pinyin Wǔhàn huánán hǎixiān pīfā shìchǎng) gearbeitet hatten, wurde dort der primäre Infektionsort vermutet.[19][20] Kurz nach Auftreten der Krankheit im Dezember 2019 hatten 27 (66 %) der ersten 41 Krankenhauspatienten den Markt im Zentrum Wuhans besucht. Die Infektionen von 13 der übrigen Betroffenen hingen allerdings nicht mit diesem Ort zusammen.[21][22]
Am 7. Januar 2020 gab der die Virusidentifizierung leitende chinesische Virologe Xu Jianguo (徐建国) bekannt, der Krankheitserreger sei ein bisher unbekanntes Coronavirus. Dies hätten Untersuchungen von Blutproben und Rachenabstrichen von 15 Erkrankten ergeben. Die WHO bestätigte diese Erkenntnis am 9. Januar 2020.[23][24] Am 13. Januar 2020 wurde die komplette RNA-Genomsequenz eines Isolats des neuen Coronavirus in der NCBI-GenBank hinterlegt (GenBank-Nummer MN908947).[25] Nahezu gleichzeitig wurde ein erstes Nachweisverfahren publiziert.[26][27][28]
Eine phylogenetische Analyse der Genomsequenzen aus Umweltproben des Marktes (etwa von Oberflächen) zeigte, dass sie mit den Viren der ersten Patienten aus Wuhan sehr nahe verwandt sind.[29] Nach einer Studie des Wuhan Hospitals hatte der erste identifizierte Patient den Markt nicht besucht.[30] Keines der untersuchten Tiere vom Markt wurde positiv auf SARS-CoV-2 getestet, was die Annahme stützt, das Virus sei nicht dort auf den Menschen übergesprungen. Offenbar hatte sich das Virus bereits zuvor unbemerkt unter Menschen etabliert. Der Markt könnte daher Schauplatz eines frühen Superspreader-Ereignisses gewesen sein.
Aus Modellierungen aufgrund der Untersuchung der Veränderungen des Erbmaterials RNA des Virus wird als erstes Auftreten des Virus als wahrscheinlich zwischen Oktober und Anfang Dezember eingegrenzt. Die Verbreitungsmuster der verschiedenen unterscheidbaren Virusmutationen spricht für eine massenhafte weltweite Ausbreitung des Virus durch eine Vielzahl von verschiedenen Ausbreitungsereignissen.[31]
Als Patient null wird ein 55-jähriger Mann aus der Provinz Hubei vermutet, der sich bereits am 17. November 2019 infiziert haben könnte.[32] Nicht alle frühen COVID-19-Fälle können mit dem Markt in Verbindung gebracht werden; die Historie des Ausbruchs ist wohl komplizierter als zunächst angenommen.[29] In einer Vorab-Publikation aus dem Herbst konnte aufgrund umfangreichen Genomvergleichs eine vermutliche RNA-Sequenz der Ausgangsform („Stammvater“, en. progenitor: proCoV2) ermittelt werden, die (wie zu erwarten) vom Genom der real existierenden Referenzform etwas abweicht. Aus den Daten lässt sich vermuten, dass dieses Virus bereits einige Wochen vor den Fällen im Dezember 2019 Menschen infiziert hat. Die Forscher lokalisieren dieses Virus und seine unmittelbaren Nachfolger in China.[33]
Am 10. November 2019 begab sich eine damals 25-jährige Frau wegen eines Hautausschlags in Mailand in ärztliche Behandlung. Anfang 2021 konnte durch Untersuchung einer seinerzeit entnommenen Hautprobe der Nachweis erbracht werden, dass die Frau schon damals mit SARS-CoV-2 infiziert war. Sie ist damit aktuell der erste nachgewiesene Fall auf der Welt. Ob sie nicht nur in Italien, sondern womöglich weltweit Patient null ist, muss noch geklärt werden.[34]
Benennung
Das Virus „SARS-CoV-2“ wird umgangssprachlich (nach der Virusfamilie) als „neuartiges Coronavirus“,[35] „neues Coronavirus“,[36] „Coronavirus“ oder nur als „Corona“ bezeichnet.[37] Die von der WHO vom 13. Januar bis zum 11. Februar 2020 verwendete Bezeichnung „2019-nCoV“ galt nach deren Aussage nur vorläufig.[38] Das National Center for Biotechnology Information (NCBI) nahm es als Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1 in die Taxonomie-Datenbank auf. Das NCBI ist jedoch für Virusnamen und -klassifikationen nicht maßgebend. Das Virus wurde dort – ebenfalls vorläufig – als Wuhan seafood market pneumonia virus geführt; als Synonyme galten 2019-nCoV und Wuhan coronavirus.[39]
Die WHO griff diverse Namensvorschläge nicht auf, die gemeinsam hatten, das Virus nach dem Ort seiner Erstidentifikation als Wuhan respiratory syndrome coronavirus (WRS-CoV) zu benennen. In der Vergangenheit hatte es Beschwerden gegeben, als Viren ihren Namen nach Ländern oder Regionen erhielten.[40] (Beispiele: das Marburg-Virus, das MERS-CoV). Daher hatte die WHO 2015 Benennungen nach dem Entdeckungsort für unerwünscht erklärt.[41] In der NCBI-Taxonomie-Datenbank aufgeführte Synonyme sind 2019-nCoV, COVID-19, COVID-19 virus, Wuhan coronavirus und Wuhan seafood market pneumonia virus (Stand 16. Februar 2020).[42][43]
Am 11. Februar 2020 gab die WHO bekannt, die durch das Virus verursachte Erkrankung als „COVID-19“ (oder „Covid-19“ für corona virus disease 2019) benannt zu haben.[44][45] Am selben Tag schlug die Coronavirus Study Group (CSG) des International Committee on Taxonomy of Viruses (ICTV) auf dem Preprint-Server bioRxiv für das Virus die nun offizielle Bezeichnung SARS-CoV-2 vor (für severe acute respiratory syndrome coronavirus 2).[1] Dem widersprach eine Woche später eine Gruppe chinesischer Virologen, die stattdessen „HCoV-19“ („Humanes Coronavirus 2019“) einführen wollten. Damit würde der Virusname an den von der WHO bestimmten Namen der Krankheit COVID-19 angeglichen. Außerdem bestünde Gefahr, SARS-CoV-2 mit SARS-CoV zu verwechseln. Sie betonten, dass sich „2019-nCoV“ von dem SARS-Virus in biologischer und epidemiologischer Hinsicht unterscheide, ebenso wie die klinischen Symptome von COVID-19 und SARS verschieden seien.[46] Zur Unterscheidung wird der Erreger von SARS auch als SARS-CoV-1 bezeichnet.[47]
Merkmale
Systematik
SARS-CoV-2 ist einer der Vertreter der Spezies Severe acute respiratory syndrome-related coronavirus (SARS-assoziiertes Coronavirus, Akronym SARSr-CoV). Zu dieser Spezies gehören aktuell (März 2020) nur SARS-CoV-2 und SARS-CoV-1. Letzteres war bisher einfach als SARS-CoV bekannt und ist der Erreger von SARS, während SARS-CoV-2 COVID-19 auslöst.
Die Spezies Severe acute respiratory syndrome-related coronavirus ist aktuell die einzige Spezies in der Untergattung Sarbecovirus. Der Name „Sarbecovirus“ bezieht sich auf die englische Bezeichnung SARS-like betacoronavirus (bzw. SARS-related betacoronavirus).[48][49]
Die Untergattung Sarbecovirus gehört der heutigen Gattung Betacoronavirus an. Die frühere Gattung Coronavirus wurde abgeschafft und deren Mitglieder auf die neuen Gattungen Alpha-, Beta-, Gamma- und Deltacoronavirus aufgeteilt. Zu den Beta-Coronaviren gehören u. a. auch SARS-CoV und MERS-CoV.[4]
Die Gattung Betacoronavirus gehört zur Unterfamilie der Orthocoronavirinae (früher schlicht Coronavirinae), und diese zur Familie der Coronaviridae, diese zur Unterordnung Cornidovirineae, diese zur Ordnung Nidovirales. Letztere wird noch in den virologischen Bereich der RNA-Viren bzw. Riboviren (Riboviria) klassifiziert, da ihr Erbmaterial aus RNA besteht. Dadurch werden aber keine weiteren Verwandtschaftsbeziehungen im phylogenetischen Sinne ausgedrückt.
Einen Stammbaum der SARS-CoV-2-Isolate, der ihre Verwandtschaft untereinander zeigt, findet man bei Li et al. (Ende Februar 2020).[50] Die Isolate gliedern sich in zwei Hauptgruppen (L-Typ nach der Aminosäure Leucin und S-Typ nach Serin), was Anlass zur Vermutung gab, das Virus könnte sich in zwei (unterschiedlich infektiöse) Zweige aufgeteilt haben.[51][52] Allerdings war es nach Meinung anderer Experten zum Zeitpunkt Anfang März 2020 noch zu früh, darüber eindeutige Aussagen machen zu können.[53][54][55][56] Die in beiden Hauptzweigen des Stammbaums basal liegenden Isolate stammen aus Wuhan, was zeigt, dass nach aktueller Datenlage das Virus dort seinen Ausgang genommen hat. Gleichwohl ist nicht ausgeschlossen, dass es einen unbekannten Vorläufer von anderswo, etwa aus der chinesischen Provinz Yunnan, in Tieren oder Menschen, gegeben haben könnte; auch das Einschleppen nach China durch den Import von Wirtstieren ist nicht ausschließbar (siehe unten →Herkunft und Wirtsspektrum).
Eine weitere Studie Anfang April machte drei Stämme A, B und C aus. Stamm A ist dem Fledermausvirus BatCoV/RaTG13 am ähnlichsten und scheint sich von Wuhan aus weltweit verbreitet zu haben; in Festlandchina selbst ist aber der Stamm B vorherrschend, der außer in China auch in Ostasien verbreitet ist. Stamm C ist der hauptsächliche Typ in Europa.[57][58]
Mutationen
Ein weltweiter Überblick zeigt den jeweiligen Stand der gemeldeten Mutationen (11. Januar 2021 rund 4020). Hiernach gibt es die elf Unterstämme 19A (04.12.2019 Erstnachweis), 19B (11.01.2020), 20A (29.01.2020), 20B (18.02.2020), 20C (17.02.2020), 20D (27.02.2020), 20E (= EU1, 20.05.2020), 20F (06.03.2020), 20G (30.06.2020), 20H (= 501Y.V2, 28.02.2020) und 20I (= 501Y.V1, 13.09.2020).[59]
Das Virus mutiert offenbar relativ langsam (ein bis zwei Mutationen pro Monat), es lässt sich also im Vergleich mit Influenzaviren zwei- bis viermal soviel Zeit.[60][61] Das bedeutet zum einen, dass es per Genomanalyse keine sehr hohe Auflösung bezüglich der Ausbreitungswege des Virus gibt, zum anderen lässt es darauf hoffen, dass eine nach überstandener Krankheit erworbene Immunität lange (monatelang) anhält. Allerdings hatten isländische Virologen von deCODE Genetics (isländisch Íslensk erfðagreining) bis zum 24. März 2020 vierzig verschiedene Mutationen allein bei Infizierten aus diesem Land identifiziert.[62][63][64] Eine der Betroffenen war mit zwei verschiedenen Ausprägungen von SARS-CoV-2 coinfiziert.[65][66]
Bei der Überwachung der genetischen Vielfalt und der Entwicklung des Virus müssen unterschieden werden:
- Stille Mutationen, die sich wegen der Degeneration des genetischen Codes nicht auf die kodierten Proteine auswirken und eine molekulargenetische Uhr definieren,
- Mutationen mit Auswirkungen auf den Phänotyp (das Erscheinungsbild des Virus in all seinen Ausprägungen). Diese weisen offenbar bei SARS-CoV-2 auf fortlaufende Anpassung an seinen neuen menschlichen Wirt hin. Wichtig für die Entwicklung von Antikörpern und Impfstoffen ist es, herauszufinden, welche Teile der kodierten Proteine stabil bleiben und konserviert werden, damit die Mittel durch Anpassung der Viren nicht schnell wirkungslos werden.[5]
Eine genaue Analyse dieser Sachverhalte findet sich bei Lucy van Dorp et al. (2020),[5] die Infektiosität des Virus könnte sich mit der Zeit erhöhen,[67][68] eine Befürchtung, die grundsätzlich auch beim bisher nur schwer von Mensch zu Mensch übertragbaren MERS-CoV besteht. In einer italienischen Studie vom Juli 2020 wurden (zu diesem Zeitpunkt) sechs SARS-CoV-Varianten unterschieden. Stamm G ist in Europa am häufigsten, dieser ist seit Ende Februar weiter mutiert in die Stämme GR und GH. Der ursprüngliche Stamm L aus Wuhan wird immer weniger gefunden, wie auch der Stamm V. Ein Stamm S wurde in einigen Gebieten der USA und Spaniens gefunden.[60]
Die im Westen dominierende Form des Virus, die sich ab Februar 2020 in Europa stark ausbreitete und von dort auch in andere Länder, hat eine Mutation D614G im Spike-Protein[69][70][71][72] und weicht damit von der Wuhan-Variante ab. Insbesondere hat diese Mutation vier- bis fünfmal mehr Spikes auf der Oberfläche des Virus.[73]
In der britischen Grafschaft Kent wurde im Dezember 2020 die Variante VOC-202012/01 (auch mit B.1.1.7, B117,[74] VUI-202012/01 und N501Y.V1 bezeichnet[75]) des Coronavirus SARS-CoV-2 mit den Mutationen 69-70del, P681H und N501Y (letztere am Spike-Protein) festgestellt.[76][77] Diese hat nach Mitteilung der britischen Regierung vom 19. Dezember gegenüber den anderen Varianten bereits die Oberhand gewonnen.[78][79] Die New and Emerging Respiratory Virus Threats Advisory Group (NERVTAG) ist der Ansicht, dass sich der neue Virusstamm, zu dem auch Varianten mit der Mutation P681H oder der Deletion von H69 und V70 im Spike-Protein gehören, schneller verbreiten kann;[80][81][82] die molekulare Ursache könnte im Fall der Mutation N501Y die bessere Bindung an den menschlichen Zellrezeptor ACE2 des viralen Spike-Proteins sein,[83][84] während die Deletion von H69 und V70 die Bindung mancher menschlicher Antikörper an das Spike-Protein verschlechtern könnte.[83] Am 20. Dezember 2020 war die Variante mit der Mutation N501Y außer in Südengland und London auch in den Niederlanden, Dänemark, Italien und Australien nachgewiesen worden.[85] In Australien war diese in New South Wales nördlich von Sydney aufgetaucht. Am 21. Dezember waren dort bereits 83 Fälle von Infektionen damit bekannt.[86] In Frankreich und in Spanien wurden die ersten Fälle am 26. Dezember 2020 bestätigt.[87] Auch in Deutschland wurde die Variante nachgewiesen (hier seit November, nachträglich festgestellt am 28. Dezember 2020), womit sich frühere Befürchtungen bestätigten;[88][89][90] genauso wie in Südkorea,[91] Indien,[74] Kanada und den USA. [92]In der Schweiz sind bereits 86 Fälle bekannt.[93]
Am 18. Dezember 2020 meldete das südafrikanische Gesundheitsministerium die Entdeckung einer weiteren variante 501.V2 alias N501Y.V2. Sie hat ebenfalls die Mutation N501Y, welche unabhängig von der in Kent ein weiteres Mal aufgetreten zu sein scheint. Diese Variante soll möglicherweise noch ansteckender sein und auch bei jungen Leuten einen schweren Krankheitsverlauf verursachen können.[77][94][95][96] Diese Variante wurde offenbar bereits in Großbritannien nachgewiesen,[97] sowie in Finnland,[92]in der Schweiz,[98] Frankreich[99] und Deutschland.[100] – in allen Ländern waren dies Rückkehrer von Reisen aus Südafrika.
Am 24. Dezember 2020 wurde bekannt, dass auch in Nigeria eine neue Variante von SARS-CoV-2 aufgetaucht ist, die sich offenbar von der britischen und südafrikanischen unterscheidet.[101]
Am 10. Januar 2021 wurde eine weitere Variante aus Japan gemeldet, die dort bei vier Einreisenden aus Brasilien gefunden wurde. Die Variante ähnelt denen aus England und Südafrika.[102][100]
Molekulargenetik und Phylogenetik
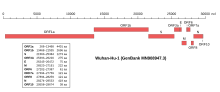
Das Virusgenom besteht, wie in Coronaviren üblich, aus einzelsträngiger RNA (ssRNA) mit positiver Polarität. Das Isolat Wuhan-Hu-1 (NCBI-GenBank-Nummer MN908947[103]) umfasst 29.903 nt (Nukleotide) mit zwei 265 nt bzw. 229 nt langen untranslatierten Bereichen am 5′-Ende bzw. am 3′-Ende.[25] Die putativen (vermuteten) Gene könnten für zehn Proteine codieren: ein 7096 Aminosäuren (AS) langes ORF1ab-Polyprotein (Replikase-Komplex), ein 1273 AS langes Oberflächen-Glykoprotein (S für englisch spikes, vergleiche Peplomer), ein 75 AS langes Hüllprotein (E für engl. envelope, vergleiche Virushülle), ein 222 AS langes Membran-Glykoprotein (M), ein 419 AS langes Nukleokapsid-Phosphoprotein (N) und weitere fünf Proteine (ORF3a, ORF6, ORF7a, ORF8 und ORF10).[25] Die Abfolge der Gene entspricht jener des SARS-Virus und der aller anderen Coronaviren.[104] Im November 2020 wurde nachträglich die Identifizierung eines „versteckten“ (überlappenden) Gens ORF3d bekannt gegeben.[105]
Mit Stand 16. Februar 2020 gab es mehr als 40 vollständige Genomanalysen von SARS-CoV-2-Isolaten. Die Genomgröße liegt zwischen 29.825 und 29.903 nt.[103] Der GC-Gehalt (der Anteil der Nukleinbasen Guanin und Cytosin) liegt bei 38,0 Mol-Prozent.[106][107] Die beiden Virusisolate HKU-SZ-002a (NCBI-GenBank-Nummer MN938384[103]) und HKU-SZ-005b (NCBI-GenBank-Nummer MN975262[103]) stammen von Patienten einer Familie aus Shenzhen und unterscheiden sich lediglich durch zwei Nukleotide. Die Genomanalyse dieser beiden Isolate ergab, dass sie nahe verwandt mit den bei Fledermäusen (englisch bat) auftretenden SARS-ähnlichen Coronaviren bat-SL-CoVZXC21 (NCBI-GenBank-Nummer MG772934) und bat-SL-CoVZC45 (NCBI-GenBank-Nummer MG772933) sind, zu letzterem besteht eine Übereinstimmung in der Nukleotidabfolge von 89 %. Das Genom der beiden Fledermaus-Coronaviren wurde 2018 sequenziert, bat-SL-CoVZC45 wurde bei der Chinesischen Hufeisennase (Rhinolophus sinicus)[108] aus der Familie der Hufeisennasen (Rhinolophidae) gefunden, die Wirtstiere wurden in Zhoushan in der ostchinesischen Provinz Zhejiang in den Jahren 2015 und 2017 untersucht.[107]

Ein weiteres Virusisolat (WIV04, NCBI-GenBank-Nummer MN996528[103]) von SARS-CoV-2 aus der bronchoalveolären Spülflüssigkeit eines der ersten Patienten zeigt ebenfalls phylogenetisch größte Ähnlichkeit mit einem bei einer anderen Fledermausart (Java-Hufeisennase, wissenschaftlich Rhinolophus affinis, englisch intermediate horseshoe bat, verbreitet in Indonesien (Java), Indien, Vietnam, China)[108] in der chinesischen Provinz Yunnan isolierten Coronavirus BatCoV RaTG13; die Genomsequenzen stimmen zu 96,2 % überein.[109][47] Auch eine am 27. Januar 2020 publizierte genetische Analyse verwies auf Fledermäuse als mutmaßlicher Ursprungswirt des Virus.[110] Am 29. Januar 2020 wurde in der Fachzeitschrift The Lancet eine genetische Analyse von zehn Virusproben publiziert, die bei neun Erkrankten gewonnen worden waren. Demnach war die Genomsequenz aller zehn Proben zu 99,98 Prozent identisch, was darauf hinweist, dass die neu entdeckte Coronavirusvariante erst vor Kurzem auf den Menschen übergegangen ist.[84][111][112] Die Genomsequenz stimmt zu 88 bzw. 87 % Prozent mit den Genomsequenzen der bei Fledermäusen auftretenden bat-SL-CoVZC45 und bat-SL-CoVZXC21 überein. Die zehn Proben zeigen hingegen nur rund 79 Prozent Übereinstimmung in der Genomsequenz zu SARS-CoV und rund 50 Prozent zu MERS-CoV. Die Ergebnisse der phylogenetischen Untersuchungen werden auch als phylogenetischer Baum, der die Verwandtschaftsverhältnisse von SARS-CoV-2 innerhalb der Coronaviridae zeigt, veranschaulicht.[107][84] Eine darauf basierende Darstellung ist im Artikel Betacoronavirus zu finden.
Der Aufbau des Genoms sowohl der SARS-CoV-2-Isolate wie auch der genannten Fledermaus-Coronaviren ist typisch für Viren der Lineage B (Untergattung Sarbecovirus, englisch SARS-like Betacoronavirus) der Gattung Betacoronavirus. Aufgrund der genetischen Distanzen zu SARS-CoV und zu MERS-CoV wurde SARS-CoV-2 zunächst als eine in Bezug auf den Menschen neue, ihn infizierende Betacoronavirus-Spezies angesehen.[107][84] Aufgrund der großen genetischen Übereinstimmung mit dem ursprünglichen SARS-Coronavirus hatte am 11. Februar 2020 die Coronavirus Study Group des ICTV jedoch vorgeschlagen, das neue Virus derselben Spezies Severe acute respiratory syndrome-related coronavirus zuzuordnen wie das bisherige.[1]
Das S-Protein (S für englisch spikes) ist für die Bindung an die Wirtszelle verantwortlich, funktionell wird es in die S1-Domäne und die S2-Domäne unterschieden. Die S1-Domäne vermittelt die Bindung an den Oberflächenrezeptor der Wirtszelle, die S2-Domäne vermittelt die Fusion der Zellmembran, durch Endozytose erfolgt dann der Eintritt des Virus in die Zelle. Das S-Gen von SARS-CoV-2 zeigt mit 75 % eine eher geringe Übereinstimmung in der Nukleotidsequenz mit den beiden Fledermausisolaten bat-SL-CoVZC45 und bat-SL-CoVZXC21 im Vergleich zur Genomanalyse. Insbesondere die Nukleotidsequenz, die für die S1-Domäne codiert, unterscheidet sich von diesen deutlich (68 % Übereinstimmung) und weist aber eine größere Ähnlichkeit mit der entsprechenden Nukleotidsequenz von BatCoV RaTG13 auf. Es wurde aufgezeigt, dass SARS-CoV-2 und SARS-CoV den gleichen Zellrezeptor nutzen, das Angiotensin-konvertierende Enzym 2 (ACE2).[84] Dies konnte experimentell sicher nachgewiesen werden, vergleiche COVID-19#Krankheitsentstehung bei COVID-19.
Beim Vergleich des Genoms von SARS-CoV-2 mit dem verwandter Fledermaus-Coronaviren zeigten sich neben der bekannten Änderung am Spike-protein zwei weitere „stille Mutationen“ (in den Nichtstrukturproteinen NSP4 und NSP16, siehe Coronaviridae §Genom), die zwar nichts an den kodierten Proteinen ändern, aber einen Einfluss auf die 3D-Faltung der RNA haben. Es wird vermutet, dass dies dazu beitragen könnte, dass infizierte Personen (anfangs) zwar ansteckend, aber (noch) symptomfrei sind.[113]
Morphologie

Die in einer Zellkultur über mehrere Tage vermehrten Viren können nach Abtrennung durch Ultrazentrifugation für die Untersuchung im Transmissionselektronenmikroskop (TEM) vorbereitet werden, dabei wird eine Negativkontrastierung verwendet. Das TEM-Bild zeigt Virionen von kugelförmiger bis pleomorpher Gestalt mit einem Durchmesser von 60 bis 140 Nanometer (nm). Auf der Oberfläche sind 9 bis 12 nm lange Spikes zu erkennen. Die Morphologie entspricht der anderer bekannter Vertreter der Familie der Coronaviridae. Die Wirtszellen, die im lichtmikroskopischen Bild einen cytopathischen Effekt aufweisen, können nach Fixierung und anschließendem Ultradünnschnitt (Dicke von 80 nm) ebenfalls mit dem TEM untersucht werden. Hier zeigen sich neben Virionen auch Einschlusskörperchen, die mit Viren gefüllte membrangebundene Vesikel im Cytoplasma enthalten.[18]
Replikationszyklus

Der Replikationszyklus der Viren verläuft über mehrere Stufen.[114]
(1) Zunächst bleiben Virionen an der Oberfläche einer Wirtszellen haften. Dies geschieht spezifisch über bestimmte Oberflächenmerkmale (Rezeptoren) der Wirtszelle, im Fall von SARS-COV-2 über die Bindung des viralen Glykoprotein S an den ACE2-Rezeptor. Der ACE2-Rezeptor der Wirtszellen könnte deshalb ein möglicher Ansatzpunkt für eine Therapie sein.[115] Ob weitere Moleküle der Zelloberfläche das Glykoprotein S binden, ist noch nicht geklärt. Im Vergleich zu SARS-CoV hat das Glykoprotein S von SARS-CoV-2 eine RGD-Peptidsequenz entwickelt, womit Rezeptoren der Integrinfamilie ebenfalls als Bindungspartner in Frage kommen[116].
(2) Nach Bindung an den ACE2-Rezeptor spaltet die membranständige Serinprotease TMPRSS2 das virale Glykoprotein S, wodurch S als fusogenes Protein aktiviert wird und der Eintritt in die Wirtszelle erfolgt. Auch TMPRSS2 ist ein potentieller Ansatzpunkt für ein wirksames Medikament.[117][118]
(3) Die Erreger dringen in die Wirtszelle ein (vereinfachte Darstellung).[119]
(4) Vor Beginn der Virusvermehrung wird die Erbsubstanz (RNA) des Virus aus dem Kapsid freigesetzt (nur ein möglicher Weg dargestellt).
(5) Nun folgt der eigentliche Vermehrungsvorgang, die Replikation. Da SARS-COV-2 über RNA positiver Polarität verfügt, kann die RNA direkt als „Bauanleitung“ (mRNA) für virusspezifische Proteine genutzt werden (Translation). Für die Wirtszelle ist die Virus-RNA praktisch nicht von eigener mRNA zu unterscheiden und der Syntheseapparat (Ribosomen) der Wirtszelle produziert virusspezifische Proteine (S, M, E, N, RNA-Polymerase).[120]
(6) Die RNA als genetische Information (Erbsubstanz) des Virus wird in der Wirtszelle durch Kopieren vervielfältigt (RNA-Replikation). Dazu sind die Enzyme der Wirtszelle nicht in der Lage, diese Aufgabe wird von der viralen RNA-Polymerase übernommen, die viele Kopien der gesamten Virus-RNA herstellt.
(7) Hat die Wirtszelle virale RNA-Kopien und Virusproteine in ausreichender Menge hergestellt, werden sie ins endoplasmatische Retikulum (ER) aufgenommen und lagern sich zu neuen Viren zusammen (Selfassembly).[121]
(8) Die fertigen Viruspartikel werden als Golgi-Vesikel aus dem ER abgeschnürt (Knospung).
(9) Durch Exozytose gelangen die Viren aus der Wirtszelle.
Herkunft und Wirtsspektrum
Seit man die Krankheit als Viruskrankheit erkannt hat, werden verschiedene Tiergruppen als Ursprung oder wenigstens Überträger des Erregers diskutiert. Eine molekulare Datierungsschätzung mittels Genom-Vergleich der verschiedenen SARS-CoV-2-Isolate legt einen Ursprung der Virusvariante im November 2019 nahe.[30][50] Van Dorp und ihre Kollegen ermittelten aufgrund phylogenetischer Analysen der verschiedenen Virusvarianten Anfang Mai 2020, dass das Virus zwischen dem 6. Oktober und dem 11. Dezember 2019 auf den Menschen übergesprungen sein soll.[5] Eine ausführliche Abhandlung der verschiedenen Abstammungstheorien – eingeschlossen kontrovers diskutierter – vom Juli 2020 findet man bei Sallard et al.[122]
Eine vergleichende Studie zum Infektionsrisiko von SARS-CoV-2 / COVID-19 haben im August 2020 Joana Damas et al. vorgelegt. Danach ist das Bindungspotential des Spike-Proteins an den jeweiligen ACE2-Rezeptor bei Primaten (Mensch, Bonobo, Gemeiner Schimpanse, Westlicher Flachlandgorilla) am größten, bei folgenden Spezies dagegen sehr gering: Kalifornischer Seelöwe, Hausmaus, Amerikanerkrähe und Mississippi-Alligator.[123] Insgesamt können mehr als 60 Säugetier-Arten von SARS-CoV-2 infiziert werden, darunter auch Füchse, Yaks, Riesenpandas und Koalas. (Stand: 6. November 2020).[124]
Schlangen und Vögel
Fachleute vermuteten zu Beginn der Epidemie in China, dass als Hauptwirt ein anderes Säugetier oder Geflügel infrage komme. Der Übergang vom Tier auf den Menschen könne jedoch über einen noch nicht identifizierten Zwischenwirt erfolgt sein.[125] Chinesische Forscher verwiesen im Journal of Medical Virology auf Schlangen wie den Vielgebänderten Krait (Bungarus multicinctus) und die Chinesische Kobra (Naja atra),[126][127] die auf dem Großmarkt, der als Infektionsort der ersten Infizierten vermutet wird, neben anderen lebenden Wildtieren (sogenannten Ye Wei) wie Fledermäusen oder Kaninchen angeboten werden.[128] Diese Hypothese wurde von anderen Virologen für unwahrscheinlich erklärt,[129] da es bisher keine Evidenz dafür gebe, dass Coronaviren auch Reptilien infizieren können. Bisher seien Coronaviren ausschließlich bei Säugetieren und Vögeln gefunden worden.[125] Weitere Untersuchungsergebnisse zu Hühnern und Enten (Galloanserae spp.) siehe unten.
Schuppentiere und Fledertiere

Hufeisennasen – möglicherweise mehrere höhlenbewohnende Arten – waren das Reservoir des Erregers SARS-CoV[-1], der die SARS-Epidemie einige Jahre zuvor ausgelöst hatte, mit dem Larvenroller (Paguma larvata, englisch masked palm civet) als möglichem Zwischenwirt zwischen Fledertier und Mensch. Seitdem wurden verschiedene weitere Betacoronaviren (insbesondere auch SARS-artige der Untergattung Sarbecovirus) vor allem bei Fledertieren, aber auch bei Menschen gefunden.[30]
Wie im November 2020 bekannt wurde, litten bereits in der zweiten Hälfte des Jahres 2012 vier Minenarbeiter an schweren Atemwegserkrankungen, die zuvor in einer Höhle beim Ort Tongguan (通关镇)[130] im Landkreis Mojiang (墨江县) in der chinesischen Provinz Yunnan gearbeitet hatten. Bei den Untersuchungen an den Patienten und in den Höhlen in diesem und den folgenden Jahren fand man SARS-ähnliche Viren (SARSr-CoVs, Untergattung Sarbecovirus) mit Ähnlichkeiten zu dem Fledermaus-Coronavirus SARSr-CoV Rp3. Eine der dabei gefundenen Varianten wurde später in BatCoV RaTG13 umbenannt, aber keine der damals gefundenen Varianten ist identisch mit SARS-CoV-2.[131][109]
Bis 2017 wurden in den Höhlen in Yunnan SARS-ähnliche Coronaviren in folgenden Fledermausspezies gefunden: in Hufeisennasenarten bei der Java-Hufeisennase (Rhinolophus affinis, en. intermediate horseshoe bat), der Chinesischen Hufeisennase (R. sinicus) und der Großen Hufeisennase (R. ferrumequinum); sowie in der Stoliczka-Dreizackblattnase (Aselliscus stoliczkanus, en. Stoliczka's trident bat).[48]
In Kot einer Horn-Hufeisennase[108] (Rhinolophus cornutus, en. little Japanese horseshoe bat) aus der Präfektur Iwate im Norden der japanischen Hauptinsel Honshu vom Jahr 2013 wurde im Herbst 2020 ein Rc-o319 genannter Sarbecovirus-Stamm gefunden, dessen Genom zu 81 % mit dem von SARS-CoV-2 übereinstimmt.[132][133]
Aufgrund der Ähnlichkeit der Bindungsstelle (en. receptor binding domain, RBD) des Spike-Proteins an den menschlichen Rezeptor ACE2 (hACE2) gilt inzwischen das Virus-Isolat BatCoV RaTG13[134] (gefunden in Java-Hufeisennasen Rhinolophus affinis, englisch intermediate horseshoe bat in Yunnan, in Bruchstücken auch bei erkrankten und verstorbenen Minenarbeitern aus Yunnan 2016)[135], als wichtiger Kandidat für den Ursprung von SARS-CoV-2, auch wenn nicht klar ist, ob die Übertragung direkt erfolgte. Die Übereinstimmungen der Genom-Sequenz zwischen RaTG13 und SARS-CoV-2 beträgt 96 %.[47][30]
Nachdem in Malaiischen Schuppentieren (Manis javanica, en. Sunda pangolin) Coronaviren mit hoher genetischer Übereinstimmung zum SARS-CoV-2 gefunden wurden (Manis-CoV, genauer Pan_SL-CoV_GD/P1L,[129] Isolate SRR10168377 und SRR10168378),[50] gerieten diese in Verdacht, der Ursprung der Pandemie zu sein, zumal diese trotz Verbots in China gehandelt werden.[45][47][136][137][138][139][129] Die Übereinstimmung betrug in diesem Fall 90 % über das gesamte Genom, aber 99 % in einer spezifischen Region des Spike-Proteins (S-Protein), die es dem Virus erlaubt, an die ACE-Rezeptoren der menschlichen Zellen zu binden.[30]
Interessanterweise ist das in den Java-Hufeisennasen (R. affinis) isolierte Virus RaTG13 gerade in diesem Genom-Abschnitt zu SARS-CoV-2 mit nur 77 % Übereinstimmung vergleichsweise unterschiedlich.[30]
Dies bedeutet, dass die aus den Malaiischen Schuppentieren isolierten Coronaviren in menschliche Zellen eindringen können, das aus Java-Hufeisennasen isolierte jedoch nicht.[30] Außerdem ist dieses Ergebnis verträglich mit der Annahme, dass das neuartige Coronavirus SARS-CoV-2 das Ergebnis einer Rekombination der RNA-Moleküle zweier verschiedener Viren sein könnte, eines dem RaTG13 aus Fledermäusen von Yunnan, das andere dem Pan_SL-CoV_GD aus den Schuppentieren von Guangdong nahestehend. Dann wäre SARS-CoV-2 entstanden als eine neue Chimäre aus zwei Viren, die diesen beiden Linien jeweils sehr nahestanden.[30][140] Diese Annahme wurde durch eine weitere Studie von Xiaojun Li und Kollegen (Duke University, Los Alamos National Laboratory, University of Texas, El Paso und New York University) Ende Mai 2020 unterstützt.[141][142][143]
Zwar besitzen Coronaviren anders als etwa Influenzaviren ein unsegmentiertes Genom (monopartit), d. h. nur ein einziges Nukleinsäuremolekül (hier RNA). Eine Rekombination von Segmenten als Ganzes (Reassortment) ist also im Gegensatz zu diesen nicht möglich. Insbesondere um den Ursprung des alten SARS-Virus SARS-CoV[-1] zu erklären wurde bereits früher bei dieser Virusfamilie einen Rekombinationsmechanismus, und zwar innerhalb des (einzigen) Genom-Segments, beschrieben (homologe Rekombination).[30][144] Eine solche Rekombination kann, egal ob segmentiertes oder unsegmentiertes Genom, zu einem neuen Virus führen, das eine neue Wirtsspezies befallen und krank machen kann.[30] Das Rekombinationsereignis kann daher zum Ausgangspunkt einer neuen Epidemie werden, wie es bei SARS vermutet (und bei Influenza stets befürchtet) wird. Voraussetzung ist die Doppelinfektion (Koinfektion) eines (einzelnen) Wirtsindividuums durch die beiden Ausgangsviren.[30] Allerdings bleibt bislang (Stand 2. Juni 2020) ungeklärt, in welcher Spezies die hypothetische Doppelinfektion stattgefunden haben könnte, und unter welchen Umständen dies geschehen sein könnte.[30]
Als alternatives Szenario, das ohne Rekombination auskommt, wird verschiedentlich etwa folgendes vorgeschlagen: Die gemeinsamen Vorfahren von RaTG13 und SARS-CoV-2 stammen danach ursprünglich von den Schuppentier-Coronaviren ab, von deren SARS-CoV-2-ähnlichstem Stamm sie sich vor mehr als 140 Jahren trennten. Diese Linie spaltete sich vor etwa 40–70 Jahren erneut auf: eine Linie verblieb in Fledermäusen und verlor dort die Bindungsfähigkeit ihres Spike-Proteins an das menschliche ACE2 (hACE2). Die andere behielt diese Fähigkeit und sprang zuletzt als SARS-CoV-2 auf den Menschen über.[145] Die verschiedenen Möglichkeiten werden auch von Halloy et al. in einem PrePrint vom Juli 2020 diskutiert.[135] Auch Boni et al. vertreten Ende Juli 2020 die Ansicht, dass SARS-CoV-2 nicht direkt aus einer Rekombination von Fledermaus- und Schuppentier-Coronaviren hervorgegangen ist, sondern dass sich seine Entwicklungslinie von der des Fledermausvirus RaTG13 vor ca. 50 Jahren getrennt hat.[146]
Weiteres zu Nilflughunden siehe unten (Abschnitt Weitere Wirbeltiere).
Anfang Dezember 2020 wurde erstmals über Funde SARS-CoV-2-ähnlicher Coronaviren bei Fledermäusen außerhalb Chinas berichtet. Neben dem oben erwähnten Fund von Rc-o319 bei der Horn-Hufeisennase aus Japan könnte man bei zwei bereits im Jahr 2010 eingefrorenen Exemplaren der Kochang-Hufeisennase[108] (Rhinolophus shameli, en. Shamel's horseshoe bat) aus dem Norden Kambodschas fündig geworden sein, die Genom-Analyse ist aber erst zu 70 % abgeschlossen (Stand 6. Dezember 2020).[132]
Marderhunde als mögliche Zwischenwirte

Laut Christian Drosten könnten Marderhunde (Nyctereutes procyonoides, eine Fuchsart) möglicherweise die gesuchten Zwischenwirte sein. Auch das ursprüngliche SARS-Virus (SARS-CoV-1) wurde in Marderhunden gefunden, die wegen ihres Fells in China gezüchtet werden und somit als Überträger auf den Menschen in Frage kommen.[147][148][149]
Haustiere als Wirte
Haushunde und Hauskatzen waren die ersten Tiere, bei denen es im Haushalt ihrer Besitzer zu Übertragungen von Mensch zu Tier kam. Testtiere beider Arten wurden daher auch in Laborexperimenten infiziert, um den Verlauf der Krankheit und mögliche Rückübertragungen auf den Menschen zu erforschen. Einer im September 2020 publizierten Studie zufolge gibt es keine Hinweise für eine Rückübertragung der Viren auf den Menschen, wohl aber Belege dafür, dass die Immunantwort von infizierten Tieren beider Arten diese vor einer zweiten Infektion schützt.[150] Laut WHO gab es bereits im März 2020 Hinweise, dass Haustiere SARS-CoV-2 nicht als Träger weiterverbreiten.[151] Jedoch können einige andere Viren aus der Corona-Virusfamilie Coronaviridae auch bei Haustieren Erkrankungen auslösen, z. B. die beiden Alphacoronaviren CCoV (Hunde) und FCoV (Katzen).[152]
Hunde
Am 28. Februar 2020 gab die Regierung Hongkongs bekannt, erstmals einen Hund positiv auf das Virus getestet zu haben, der im Haushalt seiner infizierten Halter lebte.[153] Die WHO bestätigte, die SARS-CoV-2-Proben seien „schwach positiv“[154] getestet worden. Obwohl bei dem Hund das Virus im Blut nachgewiesen werden konnte,[155] löste es bei ihm keine klinisch nachweisbaren Hinweise auf eine Erkrankung aus.[156] Das Tier wurde zuletzt am 12. und 13. März 2020 mit negativem Befund auf SARS-CoV-2 getestet, so dass seine Quarantäne beendet und es dem Besitzer zurückgegeben wurde. Zwei Tage nach Ende der Quarantäne verstarb der Hund, ohne dass ein direkter Zusammenhang mit dem Virusbefall nachweisbar war.[157]
Mitte März wurden in Hongkong zwei weitere Hunde positiv auf SARS-CoV-2 getestet, die ebenfalls ohne auffällige Symptome einer Infektion waren.[158] Aus Japan wurde im September 2020 bekannt, dass dort zwischen April und August vier Hunde von an SARS-CoV-2 erkrankten Haltern positiv getestet, ohne auffällige Symptome einer Infektion isoliert und nach wiederholtem, negativem Test an ihre gesundeten Halter zurückgegeben worden waren.[159]
Mitte April 2020 erschien ein Artikel über die Möglichkeit von streunenden Hunden als Zwischenwirt für die Übertragung von Sarbecoviren (RaTG13, Pangolin-CoV) von Wildtieren (Fledermäusen, Schuppentieren) auf den Menschen. Eine wichtige Rolle spielt dabei das Zinkfingerprotein ZAP.[160][161]
Bei einer experimentellen Studie wurden drei Hunde mit dem Virus infiziert. Keiner der Hunde zeigte klinische Zeichen einer Infektion und es konnte auch keine Ausscheidung von vermehrungsfähigem Virus nachgewiesen werden.[162]
Katzen
In Lüttich (Belgien) wurde Ende März 2020 die Hauskatze eines Infizierten positiv auf SARS-CoV-2 getestet. Das Tier litt vorübergehend an Durchfall, Erbrechen und erschwerter Atmung.[163][164] Eine Ende März 2020 in Hongkong bei einer Hauskatze nachgewiesene Infektion verlief hingegen symptomlos.[165] Antikörpernachweise hatten zuvor bereits in Wuhan ergeben, dass dort auch Katzen infiziert worden waren.[166] Zudem wurde mehrfach in Laborexperimenten belegt, dass infizierte Katzen die Viren an andere Katzen weitergeben können.[167][168][169] Es besteht der Verdacht, dass eine Katze das Virus zwischen Bewohnern eines Altenheims in Bayern übertragen haben könnte, obwohl sie voneinander isoliert waren.[170] Eine weitere infizierte Katze wurde in Barcelona untersucht. Das Tier war wegen einer Herzerkrankung eingeschläfert worden, jedoch ergab die Autopsie, dass es nicht an, sondern mit SARS-CoV-2 erkrankt war.[171] Auch in der Schweiz wurde das Virus bei einer Katze nachgewiesen.[172]
Eine experimentelle Studie bei der sieben Katzen infiziert worden waren zeigte dass diese rund fünf Tage übertragbares Virus ausschieden und auch andere Katzen infizieren konnten. Keine der untersuchten Katzen zeigte klinische Zeichen einer Infektion. Die durchgemachte Infektion schützte die Tiere vor einer Reexposition mit dem Virus.[173]
Marderverwandte
Im April und Mai 2020 wurden Infektionen und Erkrankungen von Amerikanischen Nerzen (Neovison vison, wie im Englischen auch Mink genannt) in mehreren niederländischen Nerz-Farmen festgestellt. Das Virus wurde vermutlich von infizierten Mitarbeitern eingeschleppt und danach von Tier zu Tier weitergegeben.[174] Die erkrankten Nerze zeigten ähnliche Symptome wie Menschen: Atemwegsbeschwerden, Probleme mit dem Verdauungstrakt, erhöhte Sterblichkeit.[175] In der vom Feinstaub belasteten Luft der Tierhaltungen wurde virale RNA nachgewiesen.[176] Detaillierte Analysen des genetisches Codes der in den Farmen und im Umland der Farmen umlaufenden SARS-CoV-2-Varianten erbrachten Anhaltspunkte dafür, dass sich zwei infizierte Mitarbeiter der Farmen bei den Nerzen angesteckt haben und dass zudem mehrere im Bereich der Farmen frei laufende Katzen ebenfalls „farm-typische“ SARS-CoV-2-Infektionen aufwiesen,[177] weswegen auch sie als mögliche Überträger von Viren auf die Nerze infrage kommen.[178] Die Studie wurde auf der ESCMID-Konferenz über Coronavirus-Erkrankungen (ECCVID, online vom 23. bis 25. September 2020) vorgestellt. Sie zeigt, dass das Virus offenbar zwischen Mensch und Amerikanischem Nerz hin- und zurück gesprungen ist, und liefert jedenfalls einen starken Beweis dafür, dass eine Übertragung von diesem Tier auf Mensch (zoonotisch) möglich ist. Untersucht wurden Ausbrüche auf 16 Nerzfarmen.[179] Im US-Bundesstaat Utah wurden zwischen Juli und September 2020 – nach auffälligen Häufungen von Todesfällen in mehreren Zuchtbetrieben – ebenfalls infizierte Mink entdeckt.[180] Mitte Oktober 2020 wurde bekannt, dass aufgrund dieser Gefahr in vielen Pelztierfarmen Massenkeulungen vorgenommen wurden, so wurden alleine im US-Bundesstaat Utah fast 10.000 Tiere getötet, in Spanien über 92.000 und (von denen 90 % mit SARS-CoV-2 infiziert gewesen sein sollen) in den Niederlanden über 1 Million. Nach Ansicht der WHO-Expertin Maria Van Kerkhove ist das Risiko einer Ansteckung des Menschen durch ein solches Tier jedoch nur „sehr begrenzt“.[181] Anfang November wurde von amtlichen Stellen in Dänemark angekündigt, sämtliche im Land gehaltenen – bis zu 17 Millionen Nerze – zu töten.[182] Vorausgegangen waren Erkenntnisse über Mutationen des Virus in Nerzen, gegen die einige der in Entwicklung befindlichen Impfstoffe gegen das Virus beim Menschen voraussichtlich nicht wirksam sind.[183] Die Nerze hatten sich wahrscheinlich bei Menschen angesteckt. Die Nerzzucht in Dänemark ist ein bedeutender Wirtschaftszweig mit jährlich rund 17 Millionen Fellen in rund 1100 Zuchtfarmen, auf denen die Tiere auf engem Raum in Käfigen gehalten werden.[183] Bei den Nerzen in Dänemark wurde eine neue Virusvariante („Cluster 5“) entdeckt,[184] von der bis zum 6. November 2020 auch bereits 200 Menschen in diesem Land infiziert hatten. Ob diese Variante gefährlicher ist als die bisherigen (oder weniger), war zu diesem Zeitpunkt noch nicht abschließend bekannt.[185][186][187][188][124] Insgesamt sind mit Stand 7. November 2020 sechs Länder von infizierten Tieren dieser Art betroffen.[189] Nach dem Friedrich-Loeffler-Institut (FLI) mit Stand 6. November 2020 sind für Deutschland keine besonderen Schutzmaßnahmen nötig, da es wegen des Verbots der Haltung von Nerzen als Pelztiere in diesem Land keine Nerzfarmen gibt.[190] Eine ausführliche Stellungnahme dazu mit entsprechenden Empfehlungen hat das Europäische Zentrum für die Prävention und die Kontrolle von Krankheiten (European Centre for Disease Prevention and Control, ECDC) am 12. November 2020 abgegeben.[191] Mitte November hatte die dänische Regierung mitgeteilt, dass diese Variante ausgemerzt sei.[192]
Durch Laborexperimente in Korea wurde belegt, dass Frettchen empfänglich für eine SARS-CoV-2-Infektion sind und diese auch an Artgenossen weitergeben können.[193] Das FLI bestätigte aufgrund eigener Tests den Befund aus Korea[170] und wies zugleich darauf hin, dass auch Nilflughunde empfänglich für eine SARS-CoV-2-Infektion sind, Schweine und Hühner hingegen nicht. Insbesondere die Empfänglichkeit von Frettchen sei ein wichtiger Befund, „da sie als Modelltiere für die Infektion des Menschen zur Erprobung von Impfstoffen oder Medikamenten eingesetzt werden könnten“.[194]
Ein experimentelles Vakzin gegen COVID-19 wird an den in ihrem Bestand stark gefährdeten (Endangered, IUCN 3.1) Schwarzfußiltissen erprobt.[195] Finnland entwickelt einen Impfstoff für Marderhunde und Amerikanische Nerze (Minks), um in den Pelztierfarmmen keine Massenkeulungen vornehmen zu müssen.[196] Auch Russland entwickelt einen Impfstoff für Nerze, Katzen und Nagetiere. Dieser soll nach Planung Ende Januar 2021 verfügbar sein.[197]
Weiteres zu Frettchen siehe unten (Abschnitt Weitere Wirbeltiere).
Primaten
Ein Übergang von SARS-CoV-2 vom Menschen auf Menschenaffen wurde erstmals Anfang 2021 nachgewiesen, und zwar bei den Gorillas im San Diego Zoo Safari Park. Laut einer Mitteilung der Zooverwaltung hatten zwei Gorillas am 6. Januar zu husten begonnen, weswegen Fäkalien der Tiere auf das Virus getestet wurden. Aufgrund der positiven Befunde wurden weitere Tests durch das veterinärmedizinische Labor des U.S. Department of Agriculture durchgeführt, die ebenfalls positive Befunde erbrachten.[198][199][200] Abgesehen vom Husten seien in der Gorilla-Gruppe keine gesundheitlichen Auffälligkeiten zu beobachten.
Weitere Befunde:
- Im Jahr 2016 wurde bei Schimpansen im Tai-Nationalpark (Elfenbeinküste) eine Infektion mit dem Humanen Coronavirus OC43 (HCoV-OC43, ein Betacoronavirus aus der Untergattung Embecovirus, Spezies Betacoronavirus 1)[201] beobachtet, das bei Menschen milde erkältungsartige Symptome hervorruft. Diese zeigten auch die Schimpansen. Es wurde (insbesondere für Wildhüter) empfohlen, zu den Wildtieren einen Mindestabstand von 7 bis 10 Meter zu halten und auch gegenüber den Tieren entsprechende Quarantänezeiten einzuhalten.[202]
- Eine chinesische Forschergruppe um Chuan Qin stellte im März 2020 vorläufige Ergebnisse ihrer Untersuchungen an Rhesusaffen als Preprint zur Verfügung. Hierbei ging es insbesondere um die Frage der Infektiosität nach überstandener Erkrankung.[203][204][205] Auch eine im Mai 2020 in Science publizierte Studie an Rhesusaffen berichtete von „schützender Immunität“ nach erstmaliger Erkrankung.[206]
- Niederländische Forscher berichteten im März 2020 in Science, dass SARS-CoV-2 bei Javaneraffen eine „COVID-19-ähnliche Krankheit“ verursache, weswegen diese Tiere als Modell für das Testen von vorbeugenden und therapeutischen Strategien geeignet seien.[207]
Weitere Wirbeltiere
Im New Yorker Bronx Zoo wurde Anfang April 2020 ein erwachsener Tiger positiv auf SARS-CoV-2 getestet,[208] nachdem bei ihm trockener Husten und keuchender Atem aufgefallen waren, jedoch keine Atemnot. Weiterhin wiesen auch zwei Löwen und fünf Tiger ähnliche Symptome auf, weswegen auch bei ihnen eine Infektion mit SARS-CoV-2 vermutet wurde. Infiziert wurden die Tiere vermutlich von einem asymptomatischen Bediensteten des Zoos. Wenige Tage nach dem Auftreten von Krankheitszeichen erholten sich die Tiere wieder.[209] Im Joburg Zoo in Johannesburg (Südafrika) infizierte sich im Juli 2020 ein Puma bei einem infizierten Tierbetreuer.[210] Chinesische Forscher berichteten im April 2020 in der Fachzeitschrift Science, dass sich das Virus in Hunden, Schweinen, Hühnern und Enten nur schlecht („poorly“) vermehre, und bestätigten, dass Frettchen und Katzen infiziert werden können.[211] Auch Goldhamster, die nach einer Infektion mit SARS-CoV[-1] nur sehr schwache Symptome entwickelt hatten und daher als Modelltiere ungeeignet waren, ließen sich im Labor mit SARS-CoV-2 infizieren, zeigten deutliche Symptome und wiesen hohe Viruskonzentrationen in Lunge und Darm auf.[212]
Wie bereits oben erwähnt, hatte das Friedrich-Loeffler-Institut (FLI) aufgrund eigener Tests solche Befunde bestätigt: Nilflughunde sind neben Frettchen (im Gegensatz zu Schweinen und Hühnern) empfänglich für eine SARS-CoV-2-Infektion.[170][194]
Diese Ergebnisse wurden durch eine Studie von Kore Schlottau (WHO) et al. (veröffentlicht im Juli 2020) ein weiteres Mal bestätigt und vertieft. Getestet wurden Nilflughunde (Rousettus aegyptiacus, englisch fruit bats), Frettchen (von den Autoren als Mustela putorius bezeichnet), Hausschweine (Sus scrofa domesticus) und Haushühner (Gallus gallus domesticus). Die Hausschweine und Haushühner erwiesen sich auch hier als nicht empfänglich für SARS-CoV-2. Sieben von neun Nilflughunden erkrankten zunächst an Rhinitis, das Virus wanderte mit weiterem Fortschreiten der Erkrankung über die Luftröhre teilweise bis in die Lunge. Bei den Frettchen wurde zwar eine noch effizientere Virusreplikation, aber bis auf eine mögliche leichte Rhinitis keine Krankheitssymptome beobachtet. Sie entwickelten wie auch die Nilflughunde Antikörper gegen SARS-CoV-2.[213]
Laut einer Studie des Friedrich-Loeffler-Instituts (FLI) zeigen Rinder eine geringe Empfänglichkeit gegenüber SARS-CoV-2.[214]
Während im Labor infizierte Mäuse offenbar keine Krankheitssymptome entwickeln, war es Y.-C. Wang und Kollegen in China möglich, bei C57BL/6-Labormäusen mit CRISPR/Cas9 das ACE2 der Mäuse (mACE2, murines ACE2) durch das des Menschen (hACE2, humanes ACE2) zu ersetzen. Die hACE2-Mäuse zeigten Virusreplikation von SARS-CoV-2 in ihren Lungen, der Luftröhre und im Gehirn. Auch der Verdauungstrakt war betroffen, so wie es bei manchen menschlichen Patienten beobachtet wird. Sie scheinen damit geeignet, um etwa einen Impfstoff zu testen, bevor er Menschen verabreicht wird;[215][216] eine Alternative zur Methode die Wirkung eines Mittels auf künstlich mutierte Sarbecoviren zu testen, wie jüngst bei Remdesivir und SARS-CoV/SARS-CoV-2 RdRp (altes SARS-Virus mit RdRP-Gen von SARS-CoV-2) geschehen.[217][218]
Eine mögliche Gefährdung von Meeressäugetieren (Waale, Robben, Seeotter) ist schwer zu beurteilen.[219]
Risikogruppe nach Biostoffverordnung
Für Beschäftigte, die durch ihre berufliche Tätigkeit mit Infektionserregern in Kontakt kommen können, gilt in Deutschland die Biostoffverordnung (BioStoffV). Der bei der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA) eingerichtete Ausschuss für Biologische Arbeitsstoffe (ABAS) hat SARS-CoV-2 am 19. Februar 2020 vorläufig in die Risikogruppe 3 nach der BioStoffV eingeordnet (zweithöchste Stufe).[220] Grundsätzlich erfolgt die Einstufung in Risikogruppen in den Technischen Regeln für biologische Arbeitsstoffe (TRBA), die von der BAuA veröffentlicht werden, für Viren ist dies die TRBA 462: Einstufung von Viren in Risikogruppen. Beim Auftreten neuartiger, noch nicht zugeordneter Krankheitserreger erfolgt zunächst eine vorläufige Einstufung durch den ABAS. In der Begründung wird auf die Ähnlichkeit von SARS-CoV-2 mit dem SARS-CoV-1 hingewiesen, der die SARS-Pandemie 2002/2003 ausgelöst hat, und auch die Ähnlichkeit in geringerem Umfang mit dem MERS-CoV wird erwähnt. Diese beiden Viren wurden ebenfalls der Risikogruppe 3 zugeordnet. Der ABAS nennt die „derzeit fehlenden Möglichkeiten zu Impfprävention und Therapie sowie die große Verbreitungsmöglichkeit in der Bevölkerung“ als Begründung für die vorläufige Zuordnung zur Risikogruppe 3.[221]
Außerdem werden Empfehlungen zur Arbeit mit dem Virus bei der Diagnostik im Labor gegeben: Nicht gezielte Tätigkeiten (vergleiche § 5 BioStoffV) – ausgehend vom Untersuchungsmaterial, also beispielsweise die Probenvorbereitung, Probenaufbereitung und die Inaktivierung, um den Nachweis mittels RT-PCR (siehe Abschnitt Nachweismethoden) durchzuführen – können unter den Bedingungen der Schutzstufe 2 durchgeführt werden. Dabei sind alle Tätigkeiten, bei denen mit Aerosolbildung zu rechnen ist, in einer mikrobiologischen Sicherheitswerkbank der Klasse II durchzuführen. Außerdem ist die entsprechende persönliche Schutzausrüstung zu tragen. Gezielte Tätigkeiten nach § 5 BioStoffV dürfen nur in Laboratorien der Schutzstufe 3 durchgeführt werden, dies betrifft z. B. die Vermehrung des Virus in einer Zellkultur.[221] Die amerikanische Gesundheitsbehörde CDC hatte zuvor ähnliche Empfehlungen herausgegeben.[222]
Klinische Erscheinungen
| Klassifikation nach ICD-10 | |
|---|---|
| U07.1 | COVID-19, Virus nachgewiesen |
| U07.2 | COVID-19, Virus nicht nachgewiesen |
| ICD-10 online (WHO-Version 2019) | |
Nachweismethoden
Vorgehensweise beim Nachweis
- → Siehe: COVID-19#Diagnostik
RT-PCR-Test
Die Nachweismethode der Charité ist die real-time quantitative Reverse-Transkriptase-Polymerase-Kettenreaktion (abgekürzt als qRT-PCR oder RT-qPCR), die als RT-PCR-Test oder auch nur als PCR-Test bezeichnet wird.
Dabei reagiert der Test auf das Vorhandensein zweier bestimmter kurzer Gensequenzen (Nukleotidsequenzen), die kennzeichnend für die genannten Viren sind, bezeichnet als E-Gen und RdRp-Gen. Das E-Gen codiert für die Virushülle (E für engl. envelope ‚Hülle‘), das RdRp-Gen für die RNA-abhängige RNA-Polymerase (RdRp für engl. RNA-dependent RNA polymerase). Der Test reagiert somit auf Bestandteile des Virus als auch auf Virenreste.[28] Neben dem an der Charité entwickelten Test haben weitere Institutionen eigene Tests entwickelt. Der vom CCDC entwickelte Test ist gegen die RNA-Region des Nucleokapsids (N-Gen) und des Nichtstrukturproteins nsp10 gerichtet. Der Test der Hong Kong University ebenso gegen die RNA-Region des Nucleokapsids und des Nichtstrukturproteins nsp14. Der vom CDC entwickelte Test richtet drei Primer gegen die RNA-Region des Nukleocapsids und beinhaltet auch einen Primer gegen die menschliche RNase P, welches eine Qualitätskontrolle der RNA-Extraktion und damit eine bessere Vergleichbarkeit der Tests ermöglicht.[223]
Der PCR-Test gilt als »Goldstandard« für den Nachweis des neuen Coronavirus SARS-CoV-2.[224] Ein positiver PCR-Test ist nicht gleichbedeutend mit Infektiosität: Der PCR-Test ist bei der empfohlenen Abstrich-Technik stets, in einigen Fällen mehrere Wochen, länger positiv als vermehrungsfähige Viren nachweisbar sind.[225][226][227] Die Infektiosität kann jedoch mittels des zum Testergebnis gehörenden Ct-Wertes abgeschätzt werden. Je höher dieser Wert, desto weniger Viruslast war vorhanden.[228] Aufgrund bisheriger Erfahrungen gibt das RKI an, dass bei einem hohen Ct-Wert (> 30) entsprechend einem niedrigen Gehalt von Virus-RNA in den meisten Fällen keine Anzüchtbarkeit in Zellkulturen beobachtet werden konnte. Eine Verkürzung der Quarantänezeit aufgrund eines Testergebnisses wird allerdings nicht empfohlen, allerdings sei eine Entisolierung bei diesem schwach-positiven Testergebnis im Rahmen einer ärztlichen Einzelfallentscheidung möglich.[229]
Ablauf
Der Test beinhaltet drei standardisierte Einzeltests (im Dt. „Assays“ genannt, aus dem Engl.) in einem definierten Verfahrensablauf:
- Screening: Das erste Assay (E-Gen) dient als Screening, da es mehrere Virusspezies der Untergattung Sarbecovirus (aus der Gattung Betacoronavirus) nachweist.
- Bestätigung: Verläuft das Screening positiv, so ist ein Bestätigungs-Assay (RdRp-Gen) durchzuführen.
- Charakterisierung: Falls auch dort ein positives Ergebnis erhalten wird, schließt sich als drittes ein Charakterisierungs-Assay an (ebenfalls RdRp-Gen).
Die letzten beiden Assays verwenden (neben anderen) eine Gensonde, die für SARS-CoV-2 spezifisch ist. Beim zweiten Assay ist zusätzlich eine Sonde enthalten, die zur Nukleotidsequenz sowohl des RNA-Teilstücks des SARS-CoV-1 als auch des SARS-CoV-2 passt.[28]
Für die Durchführung der qRT-PCR werden 90 Minuten bis drei Stunden benötigt.[230] Anstelle der real-time quantitative-Variante können die Produkte der Reverse-Transkriptase-PCR auch mittels Agarose-Gelelektrophorese nachgewiesen werden.[107]
Entwicklung
Die erste Version des real-time RT-PCR-Assays ist erstellt worden, bevor die Genomsequenz von SARS-CoV-2 veröffentlicht war. Für das Primerdesign wurden hier noch das erste SARS-assoziierte Coronavirus SARS-CoV-1 und weitere, bei Fledermausarten vorkommende SARS-assoziierte Coronaviren (Sarbecoviren[28]) verwendet. Nach der Veröffentlichung der Genomsequenz wurden die Primer ausgewählt, die für den Nachweis von SARS-CoV-2 geeignet sind.[28]

Die molekularbiologische Nachweismethode, die im Konsiliarlabor an der Charité verwendet wird, wurde in Zusammenarbeit mit der Berliner Biotechnologiefirma TIB Molbiol im Schnellverfahren entwickelt,[231] eine erste Version war bereits am 13. Januar 2020 verfügbar.[28] Dazu haben chinesische Wissenschaftler bereitwillig unveröffentlichte Befunde beigetragen. Von internationalen Forschungsnetzen kamen grundlegende Daten, und die globale Sektion des Europäischen Virus-Archivs (EVAg) lieferte notwendige Produkte (SARS-CoV-1-RNA und RNA-Transkripte) für die Assays.[28] Auch weitere Gruppen von Wissenschaftlern haben ihre entwickelten Methoden veröffentlicht. Dabei handelt es sich um PCR-Protokolle oder Auflistungen geeigneter Primer und deren für die RT-PCR verwendete Stoffmengenkonzentration, beispielsweise von den Centers for Disease Control and Prevention (CDC) in den USA, den CDC in China oder der Universität Hongkong. Sie unterscheiden sich darin, welche Gene der Virus-RNA nachgewiesen werden, beispielsweise das N-Gen (N für das Nukleokapsid-Phosphoprotein), das ORF1ab-Gen (codiert für das ORF1ab-Polyprotein) oder das Gen für das Spike-Protein.[232] Von der in Berlin entwickelten Nachweismethode wurden in den ersten zwei Monaten bereits 40.000 Testkits, zur Untersuchung von 4 Millionen Proben, in über 60 Länder verkauft.[233]
In Deutschland wurde im März 2020 eine Methode vorgestellt, bei der Proben mehrerer Testpersonen zusammengeführt und gemeinsam getestet werden. Ein negativer Befund bedeutet, dass die Ergebnisse aller Proben zuverlässig negativ sind, und nur bei einem positiven Befund müssen die Proben einzeln untersucht werden. Daher lassen sich, so die Forscher, mit vergleichbarem Aufwand deutlich mehr Personen testen als mit herkömmlichen Verfahren, sofern die untersuchten Personen eine geringe Infektionswahrscheinlichkeit aufweisen. Damit ließe sich, so die Forscher, in Deutschland die Zahl der täglichen Untersuchungen auf 200.000 bis 400.000 steigern.[234]
Aussagekraft
Die wichtigsten Gütekriterien für die Aussagekraft (Validität) diagnostischer Labortests sind Spezifität und Sensitivität.
Unter Spezifität wird die Wahrscheinlichkeit verstanden, dass eine eigentlich negative Probe auch als negativ erkannt wird (Ausschluss von Falsch-Positiven). Unter Sensitivität dagegen, die Wahrscheinlichkeit, dass eine eigentlich positive Probe auch als positiv erkannt wird (Ausschluss von Falsch-Negativen).
Bei niedriger Prävalenz und niederschwelliger Testindikation (z. B. Testung asymptomatischer Personen) wird insbesondere an die Spezifität des Tests eine hohe Anforderung gestellt.[235] Präventives Testen ohne begründeten Verdacht erhöht das Risiko falsch-positiver Ergebnisse. Das RKI empfiehlt jedoch angesichts der epidemiologischen Lage die Testung von asymptomatischen engen Kontaktpersonen, sowie Personen mit erhöhtem Risiko für Ansteckung und Verbreitung wie zum Beispiel Mitarbeitern des Gesundheitspersonals oder Bewohnern von Regionen mit erhöhtem Infektionsgeschehen.[236]
Zu Umfang und Bedeutung falsch-positiver Testergebnisse gibt das Robert Koch-Institut in seinen FAQ unter „Welche Rolle spielen falsch-positive Testergebnisse?“ an, dass aufgrund des Funktionsprinzips des PCR-Tests und der hohen Qualitätsanforderungen die analytische Spezifität bei nahezu 100 % liege. Daraus folgert das RKI, dass bei korrekter Durchführung der Teste und fachkundiger Beurteilung der Ergebnisse von einer sehr geringen Zahl falsch-positiver Befunde auszugehen sei und die Einschätzung der Lage nicht verfälscht werde.[237] Dem entspricht auch die Einschätzung der Nationalen Akademie der Wissenschaften: Sie betont, dass die Quote von falsch positiven PCR-Testergebnissen erheblich geringer ist als anhand der bloßen technischen Spezifitätsdaten einzelner Tests angegeben, da initial positive Ergebnisse in der Praxis stets einer Bestätigungstestung unterzogen werden.[238]
Spezifität
Der Test reagiert positiv auf SARS-CoV-2 und das SARS-CoV-1. Die Kreuzreaktivität auf SARS-CoV-1 ist gewollt, um SARS-CoV-1 (aus Laborbeständen) als positive Testkontrolle verfügbar zu machen.[28] Weitere Daten aus der Test-Entwicklung deuten darauf hin, dass der Test auf „wahrscheinlich alle asiatischen Viren“[28] (übersetzt aus dem Englischen) aus der Untergattung Sarbecovirus[28] ein positives Ergebnis liefert.
In der Entwicklung wurde sichergestellt, dass der Test nicht positiv auf die endemischen humanen Coronaviren (HCoV-229E, -NL63, -OC43, -HKU1), das MERS-CoV und viele andere übliche Erreger von respiratorischen Erkrankungen reagiert. Wie andere etablierte Teste für menschliche Coronaviren reagiert auch dieser Test positiv auf verschiedene, beim Menschen unbekannte Coronaviren (insbesondere solche von bestimmten Fledermausarten).[28]
Um festzustellen, dass der Test nicht auf ein anderes Virus ungewollt positiv reagiert, werden Proben getestet, die das Zielvirus nicht enthalten. Somit wird hier sichergestellt, dass der Test eine negative Probe in diesen Fällen auch tatsächlich als negativ anzeigt. Aufgrund dieser Untersuchungen wird die Spezifität dieses Tests als äußerst hoch eingeschätzt, sofern (aus anderen Erwägungen) sichergestellt werden kann, dass die getestete Probe frei von SARS-CoV-1 und anderen asiatischen Sarbecoviren ist.[28]
In einem Ringversuch der deutschen Instand e. V. (Gesellschaft zur Förderung der Qualitätssicherung in medizinischen Laboratorien) wurde im Mai und Juni 2020 die Qualität von 463 Laboren aus 36 Ländern unter anderem darauf untersucht, ob sie bei den Tests zuverlässig falsch positive Ergebnisse ausschließen können. Dabei erzielten die Labore für die SARS-CoV-2 negativen Proben überwiegend richtige negative Ergebnisse (97,8 % bis 98,6 %). Labore, die nicht ausschließlich 100 % richtige Ergebnisse liefern konnten, wurden nicht zertifiziert.[239]
Die Foundation for Innovative New Diagnostics in Genf hat im Juni 2020 die Sensitivität und die Spezifität von SARS-CoV-2-Testsystemen von 21 Herstellern durch Vergleich mit einem eigenen Inhouse-Test ermittelt. Die Spezifität wurde anhand von jeweils 100 negativen Proben bestimmt und hatte eine Spannweite von 96 % bis 100 %. Unklar ist bislang, ob die als falsch-positiv interpretierten Ergebnisse tatsächlich falsch-positiv sind, oder auf falsch-negative Referenzergebnisse des eigenen Inhouse-Tests zurückzuführen sind.[240]
In einer Publikation aus dem Friedrich-Loeffler-Institut Pitfalls in SARS-CoV-2 PCR Diagnostics berichten die Autoren über das Auftreten von herstellerseitigen Kontaminationen kommerzieller Primer/Sonden-Sets mit der SARS-CoV-2-Zielsequenz des RT-qPCR. Da auch Kontaminationen der verwendeten Reagenzien zu falschen Testergebnissen führen können, empfehlen die Autoren eine Chargen-Prüfung sowie systematischer Qualitätskontrollen im Testablauf.[241]
Sensitivität
Während die hohe Spezifität weitgehend akzeptiert wurde, wird die Sensitivität des Tests des Öfteren kritisiert und es wird von häufigen falsch negativen Ergebnissen berichtet. Im Ergebnis führte das z. B. bei mehrfach hintereinander getesteten Patienten dazu, dass der Status immer wieder zwischen positiv, negativ und unklarem Ergebnis wechselte. Das Problem wird hier aber nicht auf der „technischen“ Seite des Tests gesehen, sondern in der richtigen Ausführung und Handhabung.[242][243] In einer Untersuchung von Abstrichergebnissen von Patienten zeigte sich eine starke Abhängigkeit der Sensitivität vom Zeitpunkt seit der Exposition mit dem Virus und dem Krankheitsverlauf. Am Tag nach dem Kontakt mit einem positiv Getesteten konnte bei einem Drittel Virus nachgewiesen werden. Am Tag des Symptombeginns war der Test bei 62 % der Patienten positiv. Die höchste Sensitivität wurde drei Tage nach Symptombeginn mit 80 %. Drei Wochen nach Symptombeginn ließen sich noch bei rund einem Drittel der Patienten Virus-RNA nachweisen.[244]
Womöglich werden zu wenige Proben, Proben von den falschen Stellen oder Proben auf die falsche Art entnommen. Das kann dazu führen, dass das Virus in der Probe fehlt, aber im Menschen trotzdem vorhanden ist. Der Test fällt dann „technisch-korrekt“ negativ aus, obschon der Mensch Spuren des Virus in sich trägt.[245][242][243]
Aus der oben erwähnten systematischen Überprüfung der Tests verschiedener Hersteller durch die Foundation for Innovative New Diagnostics in Genf im Juni 2020, bei der die Sensitivität anhand von jeweils 50 positiven Proben bestimmt wurde, ergab sich, dass die Sensitivität eine Spannweite von 90,00 % bis 100,00 % hatte.[240]
Weitere Methoden
Genomanalyse
Laboratorien mit Ausstattung für eine Genomanalyse (DNA-Sequenzierung des Genoms), also einem Sequenzierautomaten, können SARS-CoV-2 auch auf diese Weise identifizieren.[246] Vollständige Genomanalysen von SARS-CoV-2-Isolaten zum Vergleich sind beispielsweise in der Gendatenbank des National Center for Biotechnology Information (NCBI) oder über die GISAID-Plattform[247] verfügbar (vergleiche Abschnitt Molekulargenetik und Phylogenetik).
Nukleinsäurenachweis


Die Weltgesundheitsorganisation (WHO) berichtete bereits Mitte Januar 2020 über die Entwicklung von vereinfachten molekularbiologischen Verfahren, die Nucleic Acid Amplification Technology (NAAT), deren Assays validiert wurden. Die NAAT-Methode beruht ebenfalls auf der RT-PCR, das fertig zusammengestellte Assay bietet jedoch den Vorteil, einfacher in der Handhabung zu sein und lässt sich von entsprechend ausgestatteten Routine-Laboratorien verwenden.[246]
Am 5. Februar 2020 gab die US-amerikanische Behörde CDC bekannt, ein derartiges Assay (Testkit) für die Anwendung in akkreditierten Diagnoselaboratorien zur Verfügung zu stellen. Das Assay wird als Centers for Disease Control and Prevention (CDC) 2019-Novel Coronavirus (2019-nCoV) Real-Time Reverse Transcriptase (RT)-PCR Diagnostic Panel bezeichnet und ist für den Nachweis sowohl des neuartigen Coronavirus wie auch SARS-ähnlicher Coronaviren in Proben der oberen und unteren Atemwege von Patienten vorgesehen. Zuvor erfolgte eine beschleunigte Zulassung durch die Gesundheitsbehörde Food and Drug Administration (FDA), somit darf das Assay seit 4. Februar 2020 auch außerhalb von Forschungseinrichtungen verwendet werden.[248] Ein Testkit ermöglicht die Untersuchung von 700 bis 800 Proben, 100 dieser Packungen gingen an US-amerikanische Labore, weitere 100 an internationale Laboratorien, die beispielsweise im Auftrag der WHO Untersuchungen durchführen.[249] Die Untersuchung dauert von der Probevorbereitung bis zum Vorliegen der Ergebnisse etwa vier Stunden.[230] Das Hamburger Unternehmen Altona Diagnostics sowie das Darmstädter Unternehmen R-Biopharm bieten seit Februar 2020 ein ähnliches Testkit für die RT-PCR an, das ebenfalls weltweit vertrieben wird. Ein Testkit ermöglicht die Analyse von etwa 100 Proben.[250] Allerdings sind diese bisher nur für die Verwendung in der Forschung zugelassen.[251]
Schnelltest
Der in den Medien verwendete Begriff Schnelltest ist nicht klar definiert, einerseits ist damit die Erwartung verbunden, die Zeitdauer bis zum Vorliegen eines Testergebnisses im Vergleich zur mittlerweile routinemäßig eingesetzten RT-qPCR-Methode zu verkürzen,[252] andererseits kann damit auch gemeint sein, dass es sich um ein Point-of-Care-Testing (deutsch patientennahe Labordiagnostik) handelt,[252] bei dem der Schritt „Probentransport zur Analyse im Zentrallabor“ entfällt, da entsprechende Geräte vor Ort, beispielsweise in einer Arztpraxis verwendet werden. Somit werden auch Kartuschensysteme, die auf der RT-PCR-Methode beruhen, in den Medien manchmal als Schnelltest bezeichnet, hier aber in einem eigenen Abschnitt dargestellt.
Ende Januar 2020 hatte Xinhua berichtet, dass die chinesische Behörde National Medical Products Administration (NMPA) am 26. Januar vier Testkits eines neuen Testverfahrens zugelassen habe. Das Assay ist von dem Biotechnologieunternehmen Sansure Biotech aus Changsha entwickelt worden. Mit Hilfe der dafür geeigneten Laborautomatisierung sollen Testergebnisse bereits nach 30 Minuten vorliegen.[230][253] Die staatliche Nachrichtenagentur teilte weiterhin mit, dass eine in Wuxi in der östlichen Provinz Jiangsu ansässige Firma in Zusammenarbeit mit dem National Institute for Viral Disease Control and Prevention eine Schnellmethode entwickelt habe. Mit dem Testkit soll das Virus innerhalb von 8–15 Minuten nachgewiesen werden, die Firma könne täglich so viele Testkits produzieren, dass damit die Untersuchung von 4000 Proben möglich sei und das Verfahren soll bereits in der Provinz Hubei eingesetzt worden sein.[254] Die Xinhua-Meldungen enthalten keinen Hinweis auf die dabei verwendeten molekularbiologischen Methoden. Weiterhin wird an der chinesischen Tianjin-Universität in Zusammenarbeit mit einer Pekinger Biotechnologiefirma ein Schnellverfahren entwickelt, bei dem nach 15 Minuten Resultate vorliegen sollen. Es befindet sich im Probeeinsatz (Stand Februar 2020).[230]
Forscher der Hong Kong University of Science and Technology meldeten Anfang Februar 2020 die Entwicklung eines tragbaren Gerätes, mit dem das neuartige Coronavirus innerhalb von 40 Minuten nachweisbar sein soll. Für das im Vergleich zur herkömmlichen qRT-PCR schnellere Verfahren werden modifizierte Chip-Thermocycler verwendet.[230] Auch Forscher des Institute for Health Innovation & Technology (iHealthtech) an der National University of Singapore berichteten im Februar 2020 darüber, eine Schnellmethode zu entwickeln. Sie basiert auf der seit 2018 verwendeten enVision-Technologie, mit der Nukleinsäuren innerhalb von 30 bis 60 Minuten nachgewiesen werden. Es wird geschätzt, dass bis zur Marktreife des neuen Testverfahrens noch mehrere Monate benötigt werden.[255]
Eine deutsche Forschergruppe der Universität Bonn um Hendrick Streeck hat im April 2020 die Ergebnisse einer Schnelltestvalidierung vorgestellt.[256] Hierbei wurde der CoV-2 Rapid Test im Rahmen eines Screenings der Bevölkerung getestet und mit parallel gewonnenen Proben zur PCR-Diagnostik verglichen. Von insgesamt 49 Personen waren 22 positiv in der PCR; der Schnelltest erkannte jedoch nur acht davon richtig als positiv (Sensitivität 36,4 %). Von den 27 PCR-negativen Personen wurden durch den Schnelltest 24 richtig als negativ diagnostiziert (Spezifität 88,9 %).
Antigenschnelltest
Zunehmend werden auch Antigennachweise für SARS-CoV-2 angeboten. Antigentests sind nicht mit Antikörpertests zu verwechseln. Antigentests weisen spezifische Eiweiße (Proteine) des Virus nach und testen somit auf eine aktuell vorliegende Infektion. Es gibt verschiedene Varianten von Antigen-Tests. Antigenschnelltests im engeren Sinne sind entweder fluoreszenz- oder chemilumineszenzbasierte Teste, die ein Auswertegerät benötigen, oder lateral-flow-Teste zur unmittelbaren visuellen Auswertung vor Ort. Grundsätzlich sind Sensitivität und Spezifizität der Tests geringer als bei PCR-Tests.[238][257][258]
Derzeit laufen unabhängige Evaluierungen verschiedener Tests. Sie deuten darauf hin, dass sich Sensitivität und Spezifizität in der Praxis deutlich von den Herstellerangaben unterscheiden können und dass zwischen den unterschiedlichen Tests erhebliche Leistungsunterschiede bestehen.[257][259] Beispielsweise fand ein Vergleich von 7 Tests Spezifizitätswerte zwischen 98,53 % und 100% bei 5 Produkten, mit zwei Ausreißern mit 94,85 % und 88,24 % Spezifizität.[260]
Wegen der geringeren Spezifizität sollte ein positives Ergebnis im Antigen-Test grundsätzlich mittels PCR bestätigt werden. Die geringere Sensitivität bedeutet, dass ein negatives Ergebnis eine Infektion nicht ausschließt. Insbesondere in der Frühphase der Infektion kann es dazu kommen, dass Infektionen nicht erkannt werden.[258] Nach Einschätzung der Nationalen Akademie der Wissenschaften bilden Antigentests daher nur eine Momentaufnahme der Viruslast der getesteten Person. Ein negatives Ergebnis eines solchen Tests hat demnach nur eine Gültigkeit von etwa einem Tag.[261]
Alle zurzeit auf dem Markt befindlichen Antigen-Schnelltests müssen von geschultem Personal durchgeführt werden. In der Praxis kann das nach Einschätzung des Robert Koch-Instituts auch Personen wie entsprechend geschulte Pflegehilfskräfte umfassen.[262][238]
Kartuschentest
Bei einem Kartuschensystem wird ein technisch aufwändiges, aber dennoch von den Größenabmessungen transportables Gerät (engl. analyzer) verwendet, in dem eine dafür konstruierte Kartusche (engl. cartridge) eingesetzt wird. Die Kartusche wird zuvor mit dem Probenmaterial, z. B. dem Abstrichtupfer bestückt, weitere Chemikalien und biologische Arbeitsstoffe für die Probevorbereitung und die Analyse sind in der Kartusche enthalten. Die für den Einmalgebrauch konzipierte Kartusche verkürzt die Dauer bis zum Vorliegen des Testergebnisses und bietet für den Benutzer den Vorteil, den Kontakt mit den Infektionserregern zu minimieren (vergleiche Abschnitt Risikogruppe nach Biostoffverordnung). Kartuschentests, auch als engl. panel bezeichnet, werden seit 2018 für die Diagnostik von Krankheitserregern, die Atemwegserkrankungen verursachen, eingesetzt. Die Methode basiert auch hier auf der RT-qPCR, der real-time quantitativen Reverse-Transkriptase-Polymerase-Kettenreaktion, da aber gleichzeitig Gene mehrerer Krankheitserreger analysiert werden, wird sie der Multiplex-PCR zugerechnet.[252]
Das Biotechnologieunternehmen Qiagen N.V. entwickelte am Standort in Hilden einen Kartuschentest als Schnelltest, der auf einem bereits international für die Diagnostik von Krankheitserregern zugelassenen Verfahren basiert, mit dem sich u. a. SARS-assoziierte Viren und EHEC nachweisen lassen.[263] Das Verfahren wurde um den Nachweis der im SARS-CoV-2-Genom vorhandenen Gene ORF1b und E erweitert und die Ergebnisse wurden mit denen der RT-PCR-Methode verglichen.[264] Das Unternehmen arbeitete mit der WHO zusammen, um eine Validierung zu erreichen.[263] Das tragbare Diagnosegerät ist für den Einsatz in Arztpraxen oder an Flughäfen geeignet. Als Probenmaterial ist ein Abstrich aus dem Rachenraum oder eine Blutprobe geeignet,[263] Testergebnisse liegen innerhalb von 60 Minuten vor.[264] In Deutschland ist die vorläufige Zulassung durch das Bundesinstitut für Arzneimittel und Medizinprodukte beantragt.[265] Die Diagnosegeräte wurden im Februar 2020 in französischen und chinesischen Krankenhäusern getestet[264] und erhielten die Zulassung der US-amerikanischen und europäischen Behörden (Stand 27. März 2020).[266] Im Vergleich zur routinemäßig eingesetzten RT-qPCR-Methode sind Kartuschentests teurer, zudem wird das dazugehörige Analyse-Gerät benötigt.[266]
Zwei weitere Diagnostik-Firmen in Deutschland entwickeln ebenfalls derartige Schnelltests.[265] Die US-amerikanische Firma Cepheid Inc hat im März 2020 eine beschleunigte Zulassung durch die FDA erhalten. Auch hier handelt es sich um ein Kartuschensystem auf der Basis von RT-qPCR mit dazugehörigem Analyzer, Testergebnisse sollen nach 45 Minuten vorliegen.[266] Von den passenden Analyse-Geräten werden zur Zeit weltweit etwa 23.000 verwendet, zumeist in Krankenhäusern, ein Analyzer kann bis zu vier Kartuschen gleichzeitig aufnehmen (Stand 27. März 2020).[266] Die Robert Bosch GmbH entwickelte zusammen mit der britischen Firma Randox Laboratories ebenfalls einen Kartuschentest, mit dem Testergebnisse nach 2,5 Stunden vorliegen sollen. Das Testsystem kam im April 2020 auf den Markt, zuerst nur mit einer Zulassung für Forschungseinrichtungen.[266] Im September 2020 wurde eine verkürzte Analyszeit von 39 Minuten veröffentlicht.[267]
Antikörpernachweis

Die Weltgesundheitsorganisation (WHO) berichtete Mitte Januar 2020 über die Entwicklung von Antikörpernachweisen als serologische Untersuchung.[246] Dadurch wird es ein Assay geben, beispielsweise ein Immunassay wie ELISA oder einen Lateral Flow Test, mit dem Antikörper aus Patientenproben (Blutserum) durch Antigen-Antikörper-Reaktion nachweisbar sind. Der Lateral Flow Test kann als Schnelltest (siehe oben) bezeichnet werden, im Sinne von Point-of-Care-Testing (deutsch: patientennahe Labordiagnostik), ein bekanntes darauf basierendes Beispiel ist der Schwangerschaftstest.
Je nachdem, auf welche Antikörper geprüft wird, ist noch eine aktuelle Infektion nachweisbar (frühe Antikörper Immunglobulin M (IgM)) oder eine bereits abgeschlossene Infektion durch späte Antikörper Immunglobulin G (IgG),[268] dieser Antikörperklassenwechsel wird als Serokonversion bezeichnet. Es gab den Vorschlag, IgM-Antikörpernachweise zur Diagnose akuter Infektionen zu nutzen,[268] allerdings zeigte die Untersuchung chinesischer Wissenschaftler von 535 Plasmaproben von 173 Patienten, dass im Zeitraum 1 bis 7 Tage nach Einsetzen der Symptome nur bei knapp 30 % der Patienten IgM nachweisbar waren, während es im Zeitraum 8 bis 14 Tage 73 % der Patienten waren. Damit kann der IgM-Antikörpernachweis die PCR-Testung nur ergänzen.[269]
Die WHO und das Robert Koch-Institut in Deutschland rufen dazu auf, Serumproben von bestätigten oder Verdachtsfällen in der Akutphase zu sammeln und zu asservieren.[270] Die WHO empfiehlt, die erste Probe in der ersten Krankheitswoche und die zweite Probe drei bis vier Wochen später zu nehmen. Damit lässt sich eine Serokonversion überprüfen.[246][271] Nach Kontakt mit SARS-CoV-2 werden nach etwa drei Wochen (entspricht etwa zwei Wochen nach Auftreten der Symptome) die Antikörper Immunglobulin A (IgA) gebildet, nach etwa 4 Wochen die IgG-Antikörper.[272] Bei der Untersuchung von 153 Patienten wurde ermittelt, dass die Serokonversion 20–30 Tage nach Einsetzen der Symptome erfolgt, also IgG-Antikörper in ausreichender Menge vorhanden sind.[269] Antikörpertests auf IgA und IgG sind somit nicht für die Akutdiagnostik erkrankter Patienten gedacht und ersetzen nicht die PCR-Analytik.[272] Ihre Ergebnisse liefern epidemiologische Daten, mit denen das Ausmaß des Ausbruchs ermittelt werden kann und helfen bei der Überprüfung der Wirksamkeit von Impfstoffen.[246][273]
Für die Aussagekraft des Antikörpertests ist die Wahl des richtigen Antigens ausschlaggebend. Falls ein bestimmter Antikörper an mehr als einem Antigen bindet, handelt es sich um eine Kreuzreaktivität, die zu falsch positiven Testergebnissen führt, da mehr als das eigentliche Antigen reagiert.[274] Als Antigene von SARS-CoV-2 sind die im Abschnitt Merkmale beschriebenen Strukturen geeignet, beispielsweise das Nukleokapsidprotein (N) oder das Spikeprotein (S) als Ganzes bzw. alternativ die S1- und S2-Domäne. In der Literatur werden mögliche Kreuzreaktionen zu den Coronaviren SARS-CoV-1, MERS-CoV, HCoV-HKU1, HCoV-OC43, HCoV-NL63, HCoV-229E sowie bei Katzen und Schweinen vorkommende Coronaviren genannt.[268][273][275] Bei der Validierung der entwickelten Methoden zum Antikörpernachweis sind die diagnostische Sensitivität (Richtig-positiv-Rate) und Spezifität (Richtig-negativ-Rate) wichtige Qualitätskriterien, die mit Hilfe folgender Tabelle veranschaulicht werden.[266][274]
| krank (infiziert) | gesund (nicht infiziert) | |
|---|---|---|
| Test positiv | positiv (P) | falsch positiv (FP) |
| Test negativ | falsch negativ (FN) | negativ (N) |
Die Sensitivität des Testverfahrens gibt den Anteil der positiv Getesteten zur Gesamtheit der tatsächlich Infizierten an, die Spezifität hingegen gibt den Anteil der negativ Getesteten zur Gesamtheit der tatsächlich Nicht-Infizierten an, als Formeln ausgedrückt:
- bzw.
Für ein zuverlässiges Testergebnis werden für beide Kriterien Werte nahe 100 % angestrebt, eine hohe Sensitivität stellt sicher, dass kein Infizierter versehentlich übersehen wird, eine hohe Spezifität spricht dafür, dass kein „Fehlalarm“ (z. B. durch Kreuzreaktivität) ausgelöst wird.[274] Für die Methodenvalidierung serologischer Nachweise wird als Referenzmethode (auch als Goldstandard bezeichnet) der Neutralisationstest (NT),[274] im Detail der Plaque-Reduktions-Neutralisationstest (PRNT) verwendet. Für die als Proben eingesetzten Patientenseren gilt, dass bei den Patienten zuvor das SARS-CoV-2 durch PCR nachgewiesen wurde bzw. bei den Kontrollen die anderen Coronaviren oder weiteren Viren.[273] Für die Aussagekraft der Spezifität ist weiterhin wichtig, dass Proben mit Antikörpern gegen viele verschiedene Viren getestet werden.

In einem chinesischen Forschungslabor wurden im Januar 2020 erste ELISA-Tests durchgeführt, als Antigen wurde das Nukleokapsidprotein (N) eines Fledermaus-Coronavirus mit Ähnlichkeit zu SARS-CoV-2 verwendet. Damit ließen sich in Serumproben eines Patienten die Antikörper IgG und IgM nachweisen und deren Titer über mehrere Tage während des Krankheitsverlaufes bestimmen. In einem zweiten Test wurden Serumproben, die 20 Tage nach den ersten Symptomen entnommen wurden, untersucht. Alle Patientenseren, aber nicht die Seren von Gesunden zeigten eine stark positive IgG-Reaktion, einige Patientenseren zeigten zusätzlich eine IgM-Reaktion, was auf eine aktuelle Immunantwort, also eine momentane Infektion hindeutet.[109]
Seit März 2020 sind kommerzielle Antikörpernachweistests verfügbar. Im Rahmen der Validierung wurden zwei ELISA-Tests überprüft, die auf einer IgA- und IgG-Reaktion auf das S1-Protein basieren, dabei wurde eine eher geringe Spezifität festgestellt, da es zur Kreuzreaktivität mit dem Humanen Coronavirus OC43 kam und damit zu falsch positiven Ergebnissen. In der Sensitivität schnitt das IgA-ELISA besser ab. Allerdings basiert die Validierungsstudie nur auf einer geringen Anzahl von Patientenseren, einmal n=10 von drei COVID-19-Patienten, zum anderen n=31 von neun COVID-19-Patienten.[273]
Zellkultur
Die Vermehrung des Virus zu Forschungszwecken in einer Zellkultur ist unter anderem in China, Australien, Frankreich, Deutschland und den USA gelungen.[18][276][277][278][279] Die chinesischen Wissenschaftler verwenden hierbei Epithelzellen des menschlichen Atemtrakts, die das mehrschichtige mukoziliäre Epithelgewebe (Flimmerepithel) simulieren, ebenso werden die Zelllinien Vero E6 und Huh-7 (isoliert aus humanem Leberkarzinom) eingesetzt.[18][109]
Behandlung
Für die Krankheit COVID-19 gibt es bisher keine spezifische Behandlung, eine Therapie zielt darauf ab, die Symptome zu lindern. Es wird jedoch untersucht, ob bereits bekannte Virostatika auch bei einer Infektion mit SARS-CoV-2 wirksam sind.
Vorbeugung
Impfstoffe/Impfung gegen COVID-19
Bereits unmittelbar nach Veröffentlichung der RNA-Sequenz des Virus wurde in mehreren Laboren mit der Impfstoffentwicklung begonnen.[280][281] Die internationale Impfstoffinitiative CEPI (Coalition for Epidemic Preparedness Innovations) plante, bis Mitte Juni 2020 erste Tests mit bis dahin entwickelten Impfstoffen durchzuführen. Dafür erhielten mehrere potentiell geeignete Unternehmen finanzielle Unterstützungen.[282] In Deutschland betraf dies u. a. die Tübinger Biotechnologiefirma CureVac, die zusammen mit dem Paul-Ehrlich-Institut an der schnellen Impfstoffentwicklung arbeitete.[283][284]
Mit Stand vom 2. Dezember 2020 sind weltweit noch 51 Vakzine in der klinischen Prüfung, davon 13 in der abschließenden Phase-III-Studie, darunter die RNA-Impfstoffe: Tozinameran (BioNTech/Pfizer) und mRNA-1273 (Moderna) sowie die Vektorimpfstoffe: AZD1222 (AstraZeneca/Oxford) und Ad26.COV2.S (Janssen Pharmaceutica).[285]
Um einen anfänglichen Mangel an Impfstoff einer gerechten Verteilung zuzuführen, wurde für Deutschland ein gemeinsames Positionspapier zur Priorisierung der COVID-19-Impfmaßnahmen durch die Ständige Impfkommission (STIKO) beim Robert Koch-Institut, den Deutschen Ethikrat und die Nationale Akademie der Wissenschaften Leopoldina entwickelt.[286]
Durch eine Impfung kann es allerdings dazu kommen, dass nicht-neutralisierende Antikörper entwickelt werden, die eventuell sogar die Infektion der Makrophagen erleichtern und damit die Infektion verschlimmern. Dies wurde beispielsweise bei der Impfung von Katzen gegen Felines Coronavirus beobachtet;[287] dort tritt diese Infektionsverstärkung nicht nur aufgrund einer Impfung, sondern auch aufgrund einer vorher durchgemachten Erkrankung mit dem Virus auf.[288] → Siehe auch: Infektionsverstärkende Antikörper
Impfung gegen andere Infektionen
Die Berliner Senatsgesundheitsverwaltung empfahl Ende Februar 2020 allen Menschen über 60 Jahre und chronisch Kranken, ihren Impfstatus zu überprüfen und gegebenenfalls die Impfung gegen Pneumokokken (Impfstoffe wie Pneumovax 23 waren jedoch im März 2020 nur noch eingeschränkt lieferbar[289]) und Keuchhusten (Pertussis) durchführen oder auffrischen zu lassen. Da Menschen über 60 Jahren und chronisch Kranke durch SARS-CoV besonders gefährdet sind, seien sie vorsorglich zu schützen.[290][291]
Hygienemaßnahmen
Die wichtigsten dieser Maßnahmen sind:
- Persönliche Händehygiene (regelmäßiges Händewaschen mit Seife, mindestens 20 Sekunden lang)
- Augen, Nase oder Mund nicht mit ungewaschenen Händen berühren
- Einhalten des Mindestabstands (1,5 bis 2 Meter) zu anderen Personen außer solchen desselben Haushalts
- Husten oder Niesen nur in Taschentuch oder Armbeuge, keinesfalls in die Hand
- Tragen einer Mund-Nasen-Bedeckung in öffentlichen Verkehrsmitteln und Gebäuden, insbesondere Spitälern, Heimen und anderen Gemeinschaftseinrichtungen; sowie im Freien, wenn nicht ausreichend Abstand eingehalten werden kann
- Geschlossene Räume ausreichend und häufig lüften
- bei Krankheitsgefühl statt Arztbesuch das Info-Telefon anrufen und zu Hause bleiben
- eine der neu entwickelten Corona-Apps am Handy installieren.
Epidemische Lage
SARS-CoV-2 verursacht die neuartige Erkrankung COVID-19 (für englisch corona virus disease 2019), die im Dezember 2019 in der Millionenstadt Wuhan der chinesischen Provinz Hubei auffällig wurde, sich im Januar 2020 in der Volksrepublik China zur Epidemie entwickelte und sich dann weltweit als COVID-19-Pandemie ausbreitete. Um einer Ausbreitung in Staaten ohne leistungsfähige Gesundheitssysteme entgegenzuwirken, rief die Weltgesundheitsorganisation (WHO) am 30. Januar 2020 die internationale Gesundheitsnotlage aus.[292] Am 11. März 2020 stufte die WHO die bisherige Epidemie zu einer Pandemie hoch.[293]
Meldepflicht
In Deutschland ist der direkte und indirekte Nachweis des neuartigen Coronavirus seit dem 23. Mai 2020 gemäß § 7 Abs. 1 Nr. 44a des Infektionsschutzgesetzes (IfSG) für Labore namentlich meldepflichtig, sofern der Nachweis auf eine akute Infektion hindeutet. Die Meldepflicht wurde bereits zum 1. Februar 2020 durch Verordnung eingeführt. Seit der gesetzlichen Regelung durch das Zweite Gesetz zum Schutz der Bevölkerung bei einer epidemischen Lage von nationaler Tragweite im IfSG war auch das Untersuchungsergebnis (einschließlich negativer Testergebnisse[294][295]) nichtnamentlich durch Labore zu melden (§ 7 Abs. 4 Nr. 1 IfSG alter Fassung). Diese nichtnamentliche Meldepflicht für Untersuchungsergebnisse (und damit für negative Testergebnisse) war jedoch ausgesetzt, solange das Robert Koch-Institut noch nicht über das Deutsche Elektronische Melde- und Informationssystem für den Infektionsschutz (DEMIS) verfügte.[296] Seit dem 19. November 2020 ist diese Meldepflicht aufgehoben.[297] Allerdings besteht für Ärzte noch eine Meldepflicht hinsichtlich der durch das Virus verursachten Atemwegserkrankung COVID-19 in Bezug auf Menschen. Seit den 2. Juli 2020 besteht eine Meldepflicht, wenn Haustiere positiv getestet wurden.[298]
In Österreich besteht ebenfalls Anzeigepflicht, und zwar nach dem Epidemiegesetz 1950[299] zusammen mit einer Verordnung.[300] Die Pflicht zur Anzeige besteht für Verdachts-, Erkrankungs- und Todesfälle aufgrund dieses Virus. Zudem wurde auch die Absonderungsverordnung[301] um das neue Coronavirus erweitert.[302]
Auch in der Schweiz existiert eine Meldepflicht.[303] Diese folgt aus dem Epidemiengesetz[304] der Schweiz in Verbindung mit der Epidemienverordnung[305] und der Verordnung des EDI über die Meldung von Beobachtungen übertragbarer Krankheiten des Menschen[306]. Nach Anhang 1 der Verordnung des EDI müssen Ärzte einen klinischen Verdacht und die Veranlassung einer erregerspezifischen Labordiagnostik und den nötigen epidemiologischen Zusammenhang melden. Nach Anhang 3 der Verordnung des EDI müssen Labore einen positiven und negativen Befund (also Nachweis) melden. Das Bundesamt für Gesundheit hat hierzu Verdachts-, Beprobungs-, Meldekriterien veröffentlicht.[307]
Weblinks
Von Behörden in Deutschland
- SARS-CoV-2: Virologische Basisdaten sowie Virusvarianten. (Stand: 23. Dezember 2020). In: Website des Robert Koch-Instituts (RKI). RKI, 23. Dezember 2020, abgerufen am 24. Dezember 2020.
- COVID-19 (Coronavirus SARS-CoV-2). Übersichtsseite des Robert Koch-Instituts (RKI). In: rki.de. Robert Koch-Institut, 9. Oktober 2020 (wird laufend aktualisiert).
- Antworten auf häufig gestellte Fragen zum Coronavirus SARS-CoV-2. In: rki.de. Robert Koch-Institut, 9. Oktober 2020 (wird laufend aktualisiert).
- Antworten auf häufig gestellte Fragen zum neuartigen Coronavirus (SARS-CoV-2). In: infektionsschutz.de. Bundeszentrale für gesundheitliche Aufklärung, 9. Oktober 2020.
- Kann das neuartige Coronavirus über Lebensmittel und Spielzeug übertragen werden? Aktualisierte Fragen und Antworten des BfR vom 10. September 2020. In: bfr.bund.de. Bundesinstitut für Risikobewertung, 10. September 2020.
- Aktuelle Informationen zum Coronavirus. In: bundesgesundheitsministerium.de. Bundesministerium für Gesundheit (Deutschland), 9. Oktober 2020 (wird laufend aktualisiert).
Von Behörden in Österreich
- Neuartiges Coronavirus (COVID-19). In: sozialministerium.at, Infektionskrankheiten A-Z. Bundesministerium für Soziales, Gesundheit, Pflege und Konsumentenschutz (Österreich), 9. Oktober 2020 (wird laufend aktualisiert).
- Informationen zum Coronavirus. In: sozialministerium.at. Bundesministerium für Soziales, Gesundheit, Pflege und Konsumentenschutz (Österreich), 9. Oktober 2020 (wird laufend aktualisiert).
Von Behörden in der Schweiz
- Neues Coronavirus. In: bag.admin.ch. Bundesamt für Gesundheit (Schweiz), 9. Oktober 2020 (wird laufend aktualisiert).
Von internationalen Organisationen
- Q & A on COVID-19. In: ecdc.europa.eu. Europäisches Zentrum für die Prävention und die Kontrolle von Krankheiten, 16. Februar 2020, abgerufen am 29. Februar 2020 (englisch).
- Weltgesundheitsorganisation: Coronavirus disease (COVID-19) outbreak. In: who.int. (englisch, wird laufend aktualisiert).
Von anderen Anbietern
- Podcasts mit Christian Drosten im NDR, oder die Skripte zur Sendung im PDF
- Coronavirus-Pandemie in Deutschland: Herausforderungen und Interventionsmöglichkeiten. (PDF; 158 kB) Nationale Akademie der Wissenschaften Leopoldina, 21. März 2020.
- The Novel Coronavirus Pneumonia Emergency Response Epidemiology Team: Vital Surveillances: The Epidemiological Characteristics of an Outbreak of 2019 Novel Coronavirus Diseases (COVID-19) – China, 2020. 21. Februar 2020, abgerufen am 1. März 2020 (englisch).
- Größte offizielle Sammlung an Experten-Antworten für Risikogruppe In: Österreichische Online-Plattform für chronisch Kranke, 30. März 2020, abgerufen am 30. März 2020.
- Zita Aretz, Victor Nicolaus: Wie das Coronavirus unsere Zellen infiziert. In: n-tv. 5. April 2020, abgerufen am 6. April 2020 (Der Titel bezieht sich explizit auf SARS-CoV-2. Mit zahlreichen Schemazeichnungen bebildert inklusive Hygienemaßnahmen zur Vermeidung einer Infektion und deren Wirkungsweise).
- Video: Bauplan des Virus
- Covid-19 – Was zu Mutationen von Coronaviren bekannt ist. (Stand: 20. Dezember 2020). In: Deutschlandfunk– Nachrichten-Online. Deutschlandradio – Körperschaft des öffentlichen Rechts, 20. Dezember 2020, abgerufen am 24. Dezember 2020 (kurz und allgemeinverständlich formuliert!).
- Ewen Callaway: The coronavirus is mutating — does it matter? In: Nature – a weekly journal of science . Natur – About the Journal, 16. September 2020, abgerufen am 24. Dezember 2020 (englisch, doi:10.1038/d41586-020-02544-6).
Einzelnachweise
- ↑ a b c d e f Alexander E. Gorbalenya, Susan C. Baker, Ralph S. Baric, Raoul J. de Groot, Christian Drosten, Anastasia A. Gulyaeva, Bart L. Haagmans, Chris Lauber, Andrey M Leontovich, Benjamin W. Neuman, Dmitry Penzar, Stanley Perlman, Leo L.M. Poon, Dmitry Samborskiy, Igor A. Sidorov, Isabel Sola, John Ziebuhr: Severe acute respiratory syndrome-related coronavirus: The species and its viruses – a statement of the Coronavirus Study Group. In: bioRxiv. 11. Februar 2020, bioRxiv: 10.1101/2020.02.07.937862v1 (Preprint-Volltext), S. 1–20, doi:10.1101/2020.02.07.937862 (englisch).
- ↑ a b c d ICTV: ICTV Taxonomy history: Severe acute respiratory syndrome-related coronavirus, EC 51, Berlin, Juli 2019; Email ratification March 2020 (MSL #35)
- ↑ Alissa Eckert, MS, Dan Higgins, MAM: ID#: 23312. In: Centers for Disease Control and Prevention (Hrsg.): Public Health Image Library (PHIL). 2020, abgerufen am 26. Februar 2020.
- ↑ a b c d Epidemiologischer Steckbrief zu SARS-CoV-2 und COVID-19 → Erreger. (Stand: 11. Dezember 2020). In: Website des Robert Koch-Instituts: COVID-19 in Deutschland. Robert Koch-Institut, 11. Dezember 2020, abgerufen am 6. Januar 2021.
- ↑ a b c d Lucy van Dorp, Mislav Acman, Damien Richard, Liam P. Shaw, Charlotte E. Ford: Emergence of genomic diversity and recurrent mutations in SARS-CoV-2. In: Infection, Genetics and Evolution. Band 83, September 2020, S. 104351, doi:10.1016/j.meegid.2020.104351, PMID 32387564, PMC 7199730 (freier Volltext).
- ↑ a b Pneumonia of unknown cause – China. Disease Outbreak News(DONs) – WHO, 5. Januar 2020, abgerufen am 29. April 2020.
- ↑ Florian Rötzer: WHO ruft international Notlage aus 30. Januar 2020.
- ↑ Coronavirus SARS-CoV-2: Risikobewertung zu COVID-19. „Die weltweite Ausbreitung von COVID-19 wurde am 11.03.2020 von der WHO zu einer Pandemie erklärt.“ In: Website des RKI. Robert Koch-Institut (RKI), 26. Mai 2020, abgerufen am 26. Mai 2020.
- ↑ The Size of SARS-CoV-2 Compared to Other Things. 16. Juli 2020, abgerufen am 31. Dezember 2020.
- ↑ Neue Erkenntnisse über Superspreader-Ereignisse – Eine Zusammenfassung aktueller Studien von Klaus Taschwer. Abgerufen am 26. Mai 2020.
- ↑ Covid-19: Welche Rolle spielen „Superspreader“ bei der Ausbreitung des Coronavirus? Deutschlandfunk–Online, 28. Mai 2020, abgerufen am 28. Mai 2020.
- ↑ Alex W.H. Chin et al.: Stability of SARS-CoV-2 in different environmental conditions. In: The Lancet Microbe. Band 1, Nr. 1, e10, 2020, doi:10.1016/S2666-5247(20)30003-3 (open access).
- ↑ Hinweise zu Erkennung, Diagnostik und Therapie von COVID-19 Patienten (PDF). Hrsg.: »Ständiger Arbeitskreis der Kompetenz- und Behandlungszentren für Krankheiten durch hochpathogene Erreger« (STAKOB) – Geschäftsstelle am Robert Koch-Institut, Stand: 5. Nov. 2020, abgerufen am 27. Nov. 2020.
- ↑ Neeltjevan Doremalen, Dylan H Morris, Myndi G Holbrook et al.: Aerosol and Surface Stability of SARS-CoV-2 as Compared with SARS-CoV-1 The New England Journal of Medicine, April 2020.
- ↑ Virologe Drosten zu Aerosol-Übertragung – "Im Alltag eher aufs Lüften konzentrieren als auf ständiges Desinfizieren". Abgerufen am 25. Mai 2020.
- ↑ Stephanie Hegarty: The Chinese doctor who tried to warn others about coronavirus. BBC News, 6. Februar 2020, abgerufen am 6. Februar 2020 (englisch).
- ↑ Deutsche Welle (www.dw.com): Whistleblower-Arzt aus Wuhan gestorben | DW | 07.02.2020. Abgerufen am 12. Mai 2020.
- ↑ a b c d Na Zhu, Dingyu Zhang, Wenling Wang, Xingwang Li, Bo Yang, Jingdong Song, Xiang Zhao, Baoying Huang, Weifeng Shi, Roujian Lu, Peihua Niu, Faxian Zhan, Xuejun Ma, Dayan Wang, Wenbo Xu, Guizhen Wu, George F. Gao, Wenjie Tan for the China Novel Coronavirus Investigating and Research Team: A Novel Coronavirus from Patients with Pneumonia in China, 2019. In: The New England Journal of Medicine. 24. Januar 2020, doi:10.1056/NEJMoa2001017 (englisch).
- ↑ Pneumonia of unknown cause – China. In: Website der WHO. 5. Januar 2020, abgerufen am 14. Januar 2020 (englisch).
- ↑ New virus surging in Asia rattles scientists. In: nature.com. 20. Januar 2020, abgerufen am 28. Januar 2020 (englisch).
- ↑ Wuhan seafood market may not be source of novel virus spreading globally. In: ScienceMag des AAAS. 26. Januar 2020, abgerufen am 23. April 2020 (englisch).
- ↑ Chaolin Huang, Yeming Wang, Xingwang Li, Lili Ren, Jianping Zhao, Yi Hu et al.: Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China, in: The Lancet Band 395, Nr. 10223 vom 15. February 2020, S. 497–506, veröffentlicht am 24. Januar 2020, doi:10.1016/S0140-6736(20)30183-5
- ↑ WHO Statement Regarding Cluster of Pneumonia Cases in Wuhan, China. WHO, 9. Januar 2020, abgerufen am 14. Januar 2020 (englisch).
- ↑ Antworten auf häufig gestellte Fragen zum Coronavirus SARS-CoV-2. In: Website des Robert Koch-Instituts. 18. März 2020, abgerufen am 19. März 2020.
- ↑ a b c F. Wu, S. Zhao, B. Yu, Y.-M. Chen, W. Wang, Y. Hu, Z.-G. Song, Z.-W. Tao, J.-H. Tian, Y.-Y. Pei, M. L. Yuan, Y.-L. Zhang, F.-H. Dai, Y. Liu, Q.-M. Wang, J.-J. Zheng, L. Xu, E. C. Holmes, Y.-Z. Zhang: Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome. In: Website Nucleotide des National Center for Biotechnology Information (NCBI). Abgerufen am 4. Februar 2020.
- ↑ WHO: Laboratory testing for 2019 novel coronavirus (2019-nCoV) in suspected human cases. Auf: who.int vom 14. Januar 2020.
- ↑ Karola Neubert: Erster Test für das neuartige Coronavirus in China ist entwickelt. In: Website Informationsdienst Wissenschaft (idw). 16. Januar 2020, abgerufen am 9. Februar 2020.
- ↑ a b c d e f g h i j k l Victor M. Corman, Olfert Landt, Marco Kaiser, Richard Molenkamp, Adam Meijer: Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. In: Eurosurveillance. Band 25, Nr. 3, 23. Januar 2020, ISSN 1560-7917, S. 2000045, doi:10.2807/1560-7917.ES.2020.25.3.2000045, PMID 31992387, PMC 6988269 (freier Volltext) – (eurosurveillance.org [abgerufen am 28. Dezember 2020]).
- ↑ a b Unlocking the Genetic Code of the Novel Coronavirus: How COVID-19 Made the Leap From Animals to Humans, auf SciTechDaily vom 26. März 2020, Quelle: University of Sydney
- ↑ a b c d e f g h i j k l Alexandre Hassanin: Coronavirus Could Be a ‘Chimera’ of Two Different Viruses, Genome Analysis Suggests, auf: sciencealert vom 24. März 2020 (Quelle: The Conversation)
- ↑ François Balloux et al.: Emergence of genomic diversity and recurrent mutations in SARS-CoV-2. 5. Mai 2020 doi:10.1016/j.meegid.2020.104351, frei zugänglicher Volltext unter: gleicher Artikel in NCBI-NIH-Artikel-Datenbank
- ↑ Aylin Woodward: The Chinese CDC now says the coronavirus didn't jump to people at the Wuhan wet market — instead, it was the site of a superspreader event, auf Business Insider vom 29. Mai 2020
- ↑ Sudhir Kumar, Qiqing Tao, Steven Weaver, Maxwell Sanderford, Marcos A. Caraballo-Ortiz, Sudip Sharma, Sergei L. K. Pond, Sayaka Miura: An evolutionary portrait of the progenitor SARS-CoV-2 and its dominant offshoots in COVID-19 pandemic, auf: bioRxiv vom 29. September 2020, doi:10.1101/2020.09.24.311845 (Preprint), dazu:
Joseph Caspermeyer: COVID-19 Patient Zero: Data Analysis Identifies the “Mother” of All SARS-CoV-2 Genomes, auf: SciTechDaily vom 7. November 2020 - ↑ Bislang ältester Nachweis einer Corona-Infektion könnte aus Italien stammen. In: Der Spiegel. 11. Januar 2021, abgerufen am 12. Januar 2021.
- ↑ Text der Verordnung über die Ausdehnung der Meldepflicht nach § 6 Absatz 1 Satz 1 Nummer 1 und § 7 Absatz 1 Satz 1 des Infektionsschutzgesetzes auf Infektionen mit dem erstmals im Dezember 2019 in Wuhan/Volksrepublik China aufgetretenen neuartigen Coronavirus („2019-nCoV“)
- ↑ Neues Coronavirus. In: bag.admin.ch. Bundesamt für Gesundheit BAG, 13. März 2020, abgerufen am 15. März 2020 (Schweizer Hochdeutsch).
- ↑ Fast 100 weitere Tote durch Covid-19 in China. Süddeutsche Zeitung, 12. Februar 2020, abgerufen am 13. Februar 2020 (Direkt aus dem dpa-Newskanal).
- ↑ Novel Coronavirus (2019-nCoV). ( vom 28. Januar 2020 im Internet Archive)
- ↑ Taxonomy ID: 2697049 Wuhan seafood market pneumonia virus. ( vom 3. Februar 2020 im Internet Archive)
- ↑ Ching-Tse Cheng: WHO declines to name new pneumonia after 'China' or 'Wuhan'. Taiwan News, 14. Januar 2020, abgerufen am 14. Januar 2020 (englisch).
- ↑ WHO issues best practices for naming new human infectious diseases. Weltgesundheitsorganisation, 8. Mai 2015, abgerufen am 6. Februar 2020 (englisch).
- ↑ Taxonomy Browser: Severe acute respiratory syndrome coronavirus 2, Taxonomy ID: 2697049. In: Website National Center for Biotechnology Information (NCBI). Weitere Synonyme in der NCBI-Datenbank: COVID-19, COVID-19 virus,. Abgerufen am 16. Februar 2020 (englisch).
- ↑ Nicky Phillips, Smriti Mallapaty, David Cyranoski: How quickly does the Wuhan virus spread? In: Nature. 21. Januar 2020, doi:10.1038/d41586-020-00146-w (englisch).
- ↑ Novel Coronavirus (2019-nCoV). (PDF; 1,0 MB) Situation Report – 22. WHO, 11. Februar 2020, abgerufen am 13. Februar 2020.
- ↑ a b Lars Fischer, Alina Schadwinkel: Verursacht das Coronavirus Engpässe bei Medikamenten? Stammt das Virus aus dem Pangolin? Website Spektrum.de, 10. Februar 2020, abgerufen am 15. Februar 2020.
- ↑ Shibo Jiang, Zhengli Shi, Yuelong Shu, Jingdong Song, George F. Gao, Wenjie Tan, Deyin Guo: A distinct name is needed for the new coronavirus. In: The Lancet. 19. Februar 2020, doi:10.1016/S0140-6736(20)30419-0 (englisch).
- ↑ a b c d Kristian G. Andersen, Andrew Rambaut, W. Ian Lipkin, Edward C. Holmes, Robert F. Garry: The Proximal Origin of SARS-CoV-2. In: virological.org, Quelle: ARTIC Network, 17. Februar 2020, Nature
- ↑ a b Ben Hu, Lei-Ping Zeng, Xing-Lou Yang, Xing-Yi Ge, Wei Zhang et al.: Discovery of a rich gene pool of bat SARS-related coronaviruses provides new insights into the origin of SARS coronavirus. In: PLOS Pathogens, 30. November 2017, doi:10.1371/journal.ppat.1006698
- ↑ ICTV Master Species List 2018b.v2. MSL #34, März 2019
- ↑ a b c Xingguang Li, Junjie Zai, Qiang Zhao, Qing Nie, Yi Li, Brian T. Foley, Antoine Chaillon: Evolutionary history, potential intermediate animal host, and cross‐species analyses of SARS‐CoV‐2, in: Journal of Medical Virology, 27. Februar 2020, doi:10.1002/jmv.25731, PDF, PMID 32104911, reseachGate
- ↑ Xiaolu Tang, Changcheng Wu, Xiang Li, Yuhe Song, Xinmin Yao, Xinkai Wu, Yuange Duan, Hong Zhang, Yirong Wang, Zhaohui Qian, Jie Cui, Jian Lu: On the origin and continuing evolution of SARS-CoV-2, auf: National Science Review (NSR, Oxford Academic) vom 3. März 2020, nwaa036, doi:10.1093/nsr/nwaa036
- ↑ Wissenschaftler entdecken zwei Hauptuntertypen des neuartigen Coronavirus, auf: People's Dayly Online, China (deutsch)
- ↑ Jessica Hamzelou: Coronavirus: Are there two strains and is one more deadly?, auf NewScientist vom 5. März 2020 (mit Coronavirus ist hier ausschließlich SARS-CoV-2 gemeint – freier Artikel)
- ↑ SARS-CoV-2: Gibt es 2 unterschiedliche Virusstämme?, auf: aerzteblatt.de vom 9. März 2020
- ↑ Gibt es zwei Stämme von Sars-CoV-2?, auf: n-tv.de vom 10. März 2020
- ↑ Korinna Hennig, Christian Drosten: Coronavirus-Update: "Viren mutieren immer", Interview auf ndr.de vom 6. März 2020
- ↑ Peter Forster, Lucy Forster, Colin Renfrew, Michael Forster: Phylogenetic network analysis of SARS-CoV-2 genomes, auf: PNAS vom 8. April 2020, doi:10.1073/pnas.2004999117
- ↑ Genetic Study Identifies Three Variants of SARS-CoV-2 Coronavirus, auf: Sci-News vom 9. April 2020
- ↑ Überblick.
- ↑ a b Daniele Mercatelli, Federico M. Giorgi: Geographic and Genomic Distribution of SARS-CoV-2 Mutations, in: Front. Microbiol., 22. Juli 2020, doi:10.3389/fmicb.2020.01800, dazu:
- The six strains of SARS-CoV-2, auf: ScienceDaily vom 3. August 2020
- ↑ Kai Kupferschmidt: Der Stammbaum der Pandemie, auf: Spektrum.de vom 18. März 2020 (mit Bezug auf Christian Drosten und Andrew Rambaut)
- ↑ Amanda Woods: Iceland scientists found 40 mutations of the coronavirus, report says, auf: New Yor Post vom 24. März 2020
- ↑ Bo Elkjær: Forskere har sporet 40 mutationer af coronavirus – alene på Island, auf: www.information.dk (Iceland outlet Information) vom 24. März 2020, dänisch
- ↑ Vanessa Chalmers: Scientists in Iceland claim they have found FORTY mutations of the coronavirus – and admit seven cases can be traced back to 'a football match in England', auf: www.dailymail.co.uk (MailOnline) vom 24. März 2020
- ↑ Alexander Elliott: Two types of COVID-19 in one individual, auf: www.ruv.is (RÚV news), Island, vom 24. März 2020
- ↑ Poppy Askham: Patient Infected With Two Strains of COVID-19 In Iceland, auf: The Reykjavík Grapevine vom 24. März 2020
- ↑ SARS-CoV-2: Mutationen könnten Ansteckungsfähigkeit weiter erhöhen. In: Ärzteblatt. 6. Mai 2020, abgerufen am 7. Mai 2020.
- ↑ Nadja Podbregar: Wie stark ist das Coronavirus mutiert? In: scinexx. 8. Mai 2020, abgerufen am 9. Mai 2020.
- ↑ Bette Korber, David D. Montefiori et. al.: Spike mutation pipeline reveals the emergence of a more transmissible form of SARS-CoV-2, bioRxiv 30. April 2020
- ↑ Bestätigt: Neue Variante „infektiöser“, auf: science.orf.at vom 3. Juli 2020
- ↑ Nadja Podbregar: Eine mutierte Form von SARS-CoV-2 hat die ursprüngliche Variante fast verdrängt, auf: scinexx.de vom 3. Juli 2020: D614 versus G614
- ↑ Bette Korber, W. M. Fischer, S. Gnanakaran, E. O. Saphire, D. C. Montefiori et al.: Tracking changes in SARS-CoV-2 Spike: evidence that D614G increases infectivity of the COVID-19 virus, in: Cell vom 2. Juli 2020, doi:10.1016/j.cell.2020.06.043
- ↑ Mehr "Stacheln" durch Mutation – Wird das Coronavirus ansteckender?, auf: n-tv.de vom 15. Juni 2020
- ↑ a b Amélie Bottolier-Depois: Here's What We Know About The New COVID-19 Mutations So Far, auf: sciencealert vom 8. Januar 2021. Quelle: AFP
- ↑ Statement der Swiss National COVID-19 Science Task Force bezüglich der neuen SARS-CoV-2 Variante VOC-202012/01 www.sciencetaskforce.ch, 25 Dezember 2020
- ↑ Jury still out on new Covid-19 mutant www.itv.com, 15 Dezember 2020
- ↑ a b Kai Kupferschmidt: U.K. variant puts spotlight on immunocompromised patients’ role in the COVID-19 pandemic, in: Science vom 23. Dezember 2020, doi:10.1126/science.abg2911, dazu:
- Thia Ghose:New Fast-Spreading Coronavirus Strain in The UK: Here's Everything You Need to Know, auf: sciencealert vom 26. Dezember 2020. Quelle: Live Science
- ↑ Großbritannien: Neue Corona-Variante offenbar deutlich ansteckender www.zeit.de, 19. Dezember 2020
- ↑ Johnson sagt Weihnachten ab Neue Corona-Mutation in Großbritannien um 70 Prozent ansteckender. In: Der Tagesspiegel. Abgerufen am 22. Dezember 2020.
- ↑ Statement from Chief Medical Officer, Professor Chris Whitty about new strain of Covid-19, GOV.UK, 19. Dezember 2020. Abgerufen am 20. Dezember 2020.
- ↑ Rapid increase of a SARS-CoV-2 variant with multiple spike protein mutations observed in the United Kingdom. In: European Centre for Disease Prevention and Control. 20. Dezember 2020, abgerufen am 24. Dezember 2020 (englisch).
- ↑ Weitere Virus-Mutation in Großbritannien. In: Tagesschau. 23. Dezember 2020, abgerufen am 24. Dezember 2020.
- ↑ a b Eric Martz: SARS-CoV-2 spike protein mutations. In: Proteopedia. 22. Dezember 2020, abgerufen am 24. Dezember 2020 (englisch).
- ↑ a b c d e Roujian Lu, Xiang Zhao, Juan Li, Peihua Niu, Bo Yang, Honglong Wu, Wenling Wang, Hao Song, Baoying Huang, Na Zhu, Yuhai Bi, Xuejun Ma, Faxian Zhan, Liang Wang, Tao Hu, Hong Zhou, Zhenhong Hu, Weimin Zhou, Li Zhao, Jing Chen, Yao Meng, Ji Wang, Yang Lin, Jianying Yuan, Zhihao Xie, Jinmin Ma, William J Liu, Dayan Wang, Wenbo Xu, Edward C Holmes, George F Gao, Guizhen Wu, Weijun Chen, Weifeng Shi, Wenjie Tan: Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding. In: The Lancet. 29. Januar 2020, doi:10.1016/S0140-6736(20)30251-8 (englisch).
- ↑ Britische Corona-Variante breitet sich aus. In: ntv. 20. Dezember 2020, abgerufen am 24. Dezember 2020.
- ↑ Elias Visontay, Paul Karp: Sydney Covid community transmission spreads outside northern beaches as cluster grows to 83, auf: The Guardian vom 21. Dezember 2021
- ↑ Nachweis der neuen Coronavirus-Variante auch in Frankreich, auf: Deutschlandfunk vom 26. Dezember 2020.< br> Neue Coronavirus-Variante nun auch in Spanien nachgewiesen, auf: Deutschland today vom 26. Dezember 2020
Gemeint ist hier in beiden Fällen die englische Variante VUI2020/12/01 alias B.1.1.7, nicht die inzwischen aufgetauchten Varianten aus Südafrika oder Nigeria. - ↑ Söder sorgt sich um Impfbereitschaft, auf: n-tv.de vom 27. Dezember 2030
- ↑ Nachweis in Niedersachsen − Mutation bereits seit November im Land, auf: n-tv vom 28. Dezember 2020
- ↑ Coronavirus-Liveticker auf n-tv vom 21. Dezember 2020 08:13 und 09:12, abgerufen um 10:31, Quelle: Christian Drosten, Charité und Olivier Véran, französischer Gesundheitsminister
- ↑ Drei Reise-Rückkehrer infiziert – Britische Virus-Variante in Südkorea bestätigt, auf: n-tv.de vom 28. Dezember 2020
- ↑ a b First US Case of Highly Contagious Coronavirus Variant Was Just Detected in Colorado, auf: sciencealert vom 29. Dezember2020. Quelle: Agence France-Presse
- ↑ Corona-Medienkonferenz – Virus-Mutation und zweiter Impfstoff – wo steht die Schweiz? Abgerufen am 13. Januar 2021.
- ↑ Wie gefährlich sind die neuen Mutationen des Coronavirus? In: Deutschlandradio. 23. Dezember 2020, abgerufen am 24. Dezember 2020.
- ↑ Neue Corona-Mutation entdeckt. In: Tagesschau. 19. Dezember 2020, abgerufen am 24. Dezember 2020.
- ↑ Neue Virus-Variante in Großbritannien entdeckt. In: Zeit Online. 23. Dezember 2020, abgerufen am 24. Dezember 2020.
- ↑ Weitere Corona-Mutation in Großbritannien, auf: n-tv vom 23. Dezember 2020
- ↑ Corona Mutation aus Südafrika in der Schweiz nachgewiesen. Abgerufen am 13. Januar 2021.
- ↑ Fall von südafrikanischer Virusvariante in Frankreich, auf: orf.at vom 31.12.2020
- ↑ a b Nachweis in Baden-Württemberg – Mutation aus Südafrika erreicht Deutschland, auf n-tv.de vom 12. Januar 2021
- ↑ Auch Nigeria meldet neue Corona-Variante, auf: n-tv.de vom 24. Dezember 2020
- ↑ Nach Einreise aus Brasilien – Weitere Corona-Variante in Japan gefunden, auf: n-tv.de vom 10. Januar 2021
- ↑ a b c d e NCBI Database Nucleotide, txid2697049 (Severe acute respiratory syndrome coronavirus 2). In: Website Nucleotide des National Center for Biotechnology Information (NCBI). Abgerufen am 16. Februar 2020.
- ↑ Matthew Frieman, Ralph Baric: Mechanisms of severe acute respiratory syndrome pathogenesis and innate immunomodulation. In: Microbiology and Molecular Microbiology Reviews. Band 72, Dezember 2008, S. 672–685, doi:10.1128/MMBR.00015-08, PMID 19052324 (englisch, open access).
- ↑ Peter Dockrill: Scientists Just Found a Mysteriously Hidden 'Gene Within a Gene' in SARS-CoV-2, auf: sciencealert vom 11. November 2020. Quelle: sLife.
- ↑ Wuhan seafood market pneumonia virus. In: Website Genome des National Center for Biotechnology Information (NCBI). Abgerufen am 16. Februar 2020.
- ↑ a b c d e Jasper Fuk-Woo Chan, Shuofeng Yuan, Kin-Hang Kok, Kelvin Kai-Wang To, Hin Chu, Jin Yang, Fanfan Xing, Jieling Liu, Cyril Chik-Yan Yip, Rosana Wing-Shan Poon, Hoi-Wah Tsoi, Simon Kam-Fai Lo, Kwok-Hung Chan, Vincent Kwok-Man Poon, Wan-Mui Chan, Jonathan Daniel Ip, Jian-Piao Cai, Vincent Chi-Chung Cheng, Honglin Chen, Christopher Kim-Ming Hui, Kwok-Yung Yuen: A familial cluster of pneumonia associated with the 2019 novel coronavirus indicating person-to-person transmission: a study of a family cluster. In: The Lancet. 24. Januar 2020, doi:10.1016/S0140-6736(20)30154-9 (englisch).
- ↑ a b c d Hufeisennasenfledermäuse. Schutzgemeinschaft Deutscher Wald, Oberursel vom 16. Dezember 2015
- ↑ a b c d Peng Zhou, Xing-Lou Yang, Xian-Guang Wang, Ben Hu, Lei Zhang, Wei Zhang, Hao-Rui Si, Yan Zhu, Bei Li, Chao-Lin Huang, Hui-Dong Chen, Jing Chen, Yun Luo, Hua Guo, Ren-Di Jiang, Mei-Qin Liu, Ying Chen, Xu-Rui Shen, Xi Wang, Xiao-Shuang Zheng, Kai Zhao, Quan-Jiao Chen, Fei Deng, Lin-Lin Liu, Bing Yan, Fa-Xian Zhan, Yan-Yi Wang, Geng-Fu Xiao, Zheng-Li Shi: A pneumonia outbreak associated with a new coronavirus of probable bat origin. In: Nature. 3. Februar 2020, doi:10.1038/s41586-020-2012-7 (englisch, dieser Artikel wurde am 23. Januar 2020 vorab ohne Peer-Review auf bioRxiv veröffentlicht).
- ↑ D. Paraskevis, E. G. Kostaki, G. Magiorkinis, G. Panayiotakopoulos, G. Sourvinos, S. Tsiodras: Full-genome evolutionary analysis of the novel corona virus (2019-nCoV) rejects the hypothesis of emergence as a result of a recent recombination event. In: bioRxiv. 27. Januar 2020, doi:10.1101/2020.01.26.920249 (englisch).
- ↑ Genom-Analysen klären Herkunft von 2019-nCoV. In: Website Deutsches Ärzteblatt. 30. Januar 2020, abgerufen am 9. Februar 2020.
- ↑ 2019-nCoV: Erste Bilder vom Virus und Erkenntnisse zum klinischen Verlauf. In: Website Deutsches Ärzteblatt. 27. Januar 2020, abgerufen am 11. Februar 2020.
- ↑ Pinpointing the 'silent' mutations that gave the coronavirus an evolutionary edge, auf: ScienceDaily vom 16. Oktober 2020
- ↑ https://link.springer.com/content/pdf/10.1007%2F978-3-8274-2241-5_14.pdf
- ↑ Qiu, Y.; Zhao, Y.; Wang, Q.; Li, J.; ZHou, Z.; Liao, C.; Ge, X. Predicting the Angiotensin Converting Enzyme 2 (ACE2) Utilizing Capability as the Receptor of SARS-CoV-2. Preprints 2020, 2020030091 (doi: 10.20944/preprints202003.0091.v1)
- ↑ Christian JA Sigrist, Alan Bridge, Philippe Le Mercier: A potential role for integrins in host cell entry by SARS-CoV-2. In: Antiviral Research. Band 177, Mai 2020, S. 104759, doi:10.1016/j.antiviral.2020.104759 (elsevier.com [abgerufen am 30. April 2020]).
- ↑ Hoffmann et al., SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a ClinicallyProven Protease Inhibitor, Cell (2020), https://doi.org/10.1016/j.cell.2020.02.052
- ↑ Blake Oberfeld, Aditya Achanta, Kendall Carpenter, Pamela Chen, Nicole M. Gilette: SnapShot: COVID-19. In: Cell. Band 181, Nr. 4, Mai 2020, S. 954–954.e1, doi:10.1016/j.cell.2020.04.013, PMC 7190493 (freier Volltext) – (elsevier.com [abgerufen am 15. Mai 2020]).
- ↑ Adedeji, Adeyemi & Severson, William & Jonsson, Colleen & Singh, Kamalendra & Weiss, Susan & Sarafianos, Stefan. (2013). Novel Inhibitors of SARS-CoV Entry acting by Three Distinct Mechanisms.. Journal of virology. 87. 10.1128/JVI.00998-13.
- ↑ RNA-Viren. Spektrum.de, abgerufen am 9. August 2020.
- ↑ http://hdl.handle.net/2268/4138
- ↑ Erwan Sallard, Jose Halloy, Didier Casane, Etienne Decroly, Jacques vanHelden: Tracing the origins of SARS-COV-2 in coronavirusphylogenies, auf: HAL archives-ouvertes, Version 2 (CCSD), 6. Juli 2020, HAL Id: hal-02891455
- ↑ Joana Damas, Graham M. Hughes, Kathleen C. Keough, Corrie A. Painter, Nicole S. Persky, Marco Corbo, Michael Hiller, Klaus-Peter Koepfli, Andreas R. Pfenning, Huabin Zhao, Diane P. Genereux, Ross Swofford, Katherine S. Pollard, Oliver A. Ryder, Martin T. Nweeia, Kerstin Lindblad-Toh, Emma C. Teeling, Elinor K. Karlsson, Harris A. Lewin; Scott V. Edwards (Hrsg.): Broad host range of SARS-CoV-2 predicted by comparative and structural analysis of ACE2 in vertebrates, in: PNAS vom 21. August 2020, doi:10.1073/pnas.2010146117.
Many Animal Species Vulnerable to SARS-CoV-2 / COVID-19 According to Genomic Analysis, auf: SciTechDaily vom 23. August 2020, Quelle: University of California - Davis.
Nadja Podbregar: Corona: Welche Tierarten sind anfällig?, auf: scinexx.de vom 24. August 2020. - ↑ a b Grahem Lawton: Is a dangerous new coronavirus strain circulating in farmed mink?, auf: NewScientist vom 5./6. November 2020
- ↑ a b Ewen Callaway, David Cyranoski: Why snakes probably aren’t spreading the new China virus – One genetic analysis suggests reptilian reservoir — but researchers doubt that the coronavirus could have originated in animals other than birds or mammals. In: Nature. 23. Januar 2020, doi:10.1038/d41586-020-00180-8 (englisch).
- ↑ Wei Ji, Wei Wang, Xiaofang Zhao, Junjie Zai, Xingguang Li: Homologous recombination within the spike glycoprotein of the newly identified coronavirus may boost cross‐species transmission from snake to human. In: Journal of Medical Virology. 22. Januar 2020, doi:10.1002/jmv.25682 (englisch).
- ↑ Researchers trace coronavirus outbreak in China to snakes. In: Website EurekAlert! 22. Januar 2020, abgerufen am 26. Januar 2020 (englisch).
- ↑ Coronavirus: How worried should we be? BBC News, 27. Januar 2020, abgerufen am 27. Januar 2020 (englisch).
- ↑ a b c Chengxin Zhang et al.: Protein Structure and Sequence Reanalysis of 2019-nCoV Genome Refutes Snakes as Its Intermediate Host and the Unique Similarity between Its Spike Protein Insertions and HIV-1, in: American Chemical Society: J. Proteome Res. vom 22. März 2020, doi:10.1021/acs.jproteome.0c00129; PrePrint, PrePrint Volltext (PDF) vom 8. Februar 2020
- ↑ OSM: Tongguan Town
- ↑ Peng Zhou, Xing-Lou Yang, Zheng-Li Shi et al.: Addendum: A pneumonia outbreak associated with a new coronavirus of probable bat origin, in: nature, 17. November 2020, doi:10.1038/s41586-020-2951-z. Nachtrag zum Artikel der Autoren vom Februar 2020
- ↑ a b Smriti Mallapaty: Coronaviren in Japan und Kambodscha eng verwandt mit Pandemievirus, auf: Spektrum.de vom 6. Dezember 2020
- ↑ Shin Murakami, Tomoya Kitamura1, Jin Suzuki, Ryouta Sato, Toshiki Aoi, Marina Fujii, Hiromichi Matsugo, Haruhiko Kamiki, Hiroho Ishida, Akiko Takenaka-Uema, Masayuki Shimojima, Taisuke Horimoto: Detection and Characterization of Bat Sarbecovirus Phylogenetically Related to SARS-CoV-2, Japan, in: Emerging Infectious Diseases Journal, Band 26, Nr. 12, Dezember 2020, ISSN 1080-6059, doi:10.3201/eid2612.203386
- ↑ NCBI: Bat coronavirus RaTG13 (no rank)
- ↑ a b Jose Halloy, Erwan Sallard, José Halloy, Didier Casane, Etienne Decroly, Jacques van Helden: Tracing the origins of SARS-COV-2 in coronavirus phylogenies, in: HAL vom 16. Juli 2020, HAL Id: hal-02891455 (Preprint)
- ↑ David Cyranoski: Did pangolins spread the China coronavirus to people? In: Nature. 7. Februar 2020, doi:10.1038/d41586-020-00364-2 (englisch).
- ↑ Mike McRae: Coronaviruses Similar to The COVID-19 One Have Just Been Found in Pangolins, auf sciencealert vom 27. März 2020 (mit „COVID-19“ ist nicht die menschliche Krankheit, sondern es sind allgemein Sarbecoviren gemeint, mit „Coronaviruses“ speziell nur SARS-CoV-2). Die Schuppentiere bzw. Teile derselben waren vom chinesischen Zoll beschlagnahmt worden, bei Pan_SL-CoV_GD in der Provinz Guandong, bei Pan_SL-CoV_GX in der Provinz Guangxi.
- ↑ Tommy Tsan-Yuk Lam et al.: Identifying SARS-CoV-2 related coronaviruses in Malayan pangolins, in: Nature vom 26. März 2020, doi:10.1038/s41586-020-2169-0 (Preprint)
- ↑ Pangolins, Not Snakes, May Be Missing Link in Coronavirus Jump From Bats to Humans, auf: SciTechDaily vom 27. März 2020, Quelle: American Chemical Society
- ↑ Tina Hesman Saey: https://www.sciencenews.org/article/coronavirus-covid-19-not-human-made-lab-genetic-analysis-nature, auf ScienceNews vom 26. März 2020
- ↑ Xiaojun Li, Elena E. Giorgi, Manukumar Honnayakanahalli Marichannegowda, Brian Foley, Chuan Xiao, Xiang-Peng Kong, Yue Chen, S. Gnanakaran, Bette Korber, Feng Gao: Emergence of SARS-CoV-2 through recombination and strong purifying selection, in: ScienceAdvances, AAAS, vom 29. Mai 2020, eabb9153, doi:10.1126/sciadv.abb9153
- ↑ Bats, Pangolins and Humans: COVID-19 Virus Likely Emerged From Recombination of Viral Genes Across Different Species, auf: ScitechDaily vom 31. Mai 2020. Zitat: „…the virus’ entire receptor binding motif (RBM), a component that plays a key role in viral entry into host cells, was introduced [into specific bat coronaviruses] through recombination with pangolin coronaviruses.“
- ↑ Evolution of Pandemic Coronavirus Outlines Path From Animals to Humans – Highlights Future Danger, auf: SciTechDaily vom 6. Juni 2020, Quelle: DUKE UNIVERSITY MEDICAL CENTER
- ↑ Rachel L. Graham, Ralph S. Baric: Recombination, Reservoirs, and the Modular Spike: Mechanisms of Coronavirus Cross-Species Transmission, in: ASM: Journal of Virology 84 (7), März 2010, S. 3134-3146, doi:10.1128/JVI.01394-09, PDF
- ↑ David Cyranoski: Virologie: Porträt eines Killers, Online-Ausgabe des Artikels in Spektrum der Wissenschaft Nr. 8, August 2020, S. 40–49.
- ↑ Maciej F. Boni, Philippe Lemey, Xiaowei Jiang, Tommy Tsan-Yuk Lam, Blair W. Perry, Todd A. Castoe, Andrew Rambaut, David L. Robertson: Evolutionary origins of the SARS-CoV-2 sarbecovirus lineage responsible for the COVID-19 pandemic, in: Nature Microbiology vom 28. Juli 2020, doi:10.1038/s41564-020-0771-4; dazu:
Nadja Podbregar: Den Wurzeln von Sars-CoV-2 auf der Spur, auf: wissenschaft.de vom 28. Juli 2020
Nadja Podbregar: Corona: SARS-CoV-2 gibt es schon seit Jahrzehnten, auf: scinexx.de vom 29. Juli 2020
Erin Garcia de Jesus: Close relatives of the coronavirus may have been in bats for decades, auf: ScienceNews vom 28. Juli 2020 - ↑ Marderhunde als Zwischenwirt? Drosten bringt neue Virusquelle ins Spiel, auf n-tv.de vom 26. April 2020
- ↑ Der Marderhund als Coronavirus-Schleuder?
- ↑ James D. Cherry, Paul Krogstad: SARS: The First Pandemic of the 21st Century
- ↑ Angela M. Bosco-Lauth, Airn E. Hartwig, Stephanie M. Porter et al.: Experimental infection of domestic dogs and cats with SARS-CoV-2: Pathogenesis, transmission, and response to reexposure in cats. In: PNAS. Online-Vorabveröffentlichung vom 29. September 2020, doi:10.1073/pnas.2013102117.
- ↑ Q&A on coronaviruses (COVID-19). Abgerufen am 6. März 2020 (englisch).
- ↑ Coronaviren können für Haustiere gefährlich werden. In: Frankfurter Neue Presse. 6. März 2020, abgerufen am 6. März 2020.
- ↑ OIE: Immediate notification. Tai Hang, Islands District, Hong Kong. Auf: oie.int (Weltorganisation für Tiergesundheit (OIE), 29. Februar 2020.
Thomas H. C. Sit, Christopher J. Brackman, Sin Ming Ip et al.: Infection of dogs with SARS-CoV-2. In: Nature. Online-Veröffentlichung vom 14. Mai 2020, doi:10.1038/s41586-020-2334-5. - ↑ Noah Higgins-Dunn: A dog in Hong Kong tests positive for the coronavirus, WHO officials confirm. In: CNBC. 28. Februar 2020, abgerufen am 29. Februar 2020 (englisch).
- ↑ OIE: Immediate notification. Tai Hang, Islands District, Hong Kong. Auf: oie.int vom 29. Februar 2020.
- ↑ Coronavirus: Nein, Hunde erkranken nicht an Covid-19. In: Der Spiegel – Wissenschaft. Abgerufen am 6. März 2020.
- ↑ OIE: Follow-up report No. 3 (Final report): COVID-19 (SARS-COV-2), Hong Kong. Likely human to animal transmission. Auf: oie.int vom 28. März 2020.
- ↑ OIE: Immediate notification. Pok Fu Lam, Southern District, Hong Kong. Auf: oie.int vom 21. März 2020.
- ↑ OIE: Immediate notification. Food Safety and Consumer Affairs Bureau, Ministry of Agriculture, Forestry and Fisheries, Tokyo, Japan. Auf: oie.int vom 25. September 2020.
- ↑ Xuhua Xia: Extreme genomic CpG deficiency in SARS-CoV-2 and evasion of host antiviral defense. In: Molecular Biologa and Evolution. Band 37, Nr. 9, 2020, S. 2699–2705, doi:10.1093/molbev/msaa094.
- ↑ Evidence of Stray Dogs as Possible Origin of COVID-19 Pandemic. Auf: scitechdaily.com vom 14. April 2020, Quelle: University of Ottawa.
- ↑ Angela M. Bosco-Lauth et al.: Experimental infection of domestic dogs and cats with SARS-CoV-2: Pathogenesis, transmission, and response to reexposure in cats. PNAS, 29. September 2020, doi:10.1073/pnas.2013102117
- ↑ Fall in Belgien – Katze positiv auf Coronavirus getestet. Auf n-tv.de vom 27. März 2020.
- ↑ Coronavirus: Belgian cat infected by owner. Auf: brusselstimes.com vom 27. März 2010.
- ↑ Bericht des Agriculture, Fisheries and Conservation Department, Hong Kong. Auf: oie.int, Weltorganisation für Tiergesundheit (OIE), 5. Mai 2020.
- ↑ Qiang Zhang et al.: SARS-CoV-2 neutralizing serum antibodies in cats: a serological investigation. Auf: bioRxiv, Preprint vom 3. April 2020, doi:10.1101/2020.04.01.021196.
- ↑ Coronavirus can infect cats — dogs not so much. Auf: nature.com vom 1. April 2020.
- ↑ Peter J. Halfmann et al.: Transmission of SARS-CoV-2 in Domestic Cats. In: The New England Journal of Medicine, Online-Publikation vom 13. Mai 2020, doi:10.1056/NEJMc2013400.
- ↑ Cats Can Spread COVID-19 Coronavirus Infection to Other Cats. Auf: scitechdaily.com vom 29. März 2020, Quelle: University of Tokyo
- ↑ a b c Lars Fischer: Coronavirus: Welche Rolle spielen Haustiere bei der Pandemie? Auf: spektrum.de vom 4. Juni 2020, Quelle: Nature
- ↑ Joaquim Segalés, Mariona Puig, Jordi Rodon et al.: Detection of SARS-CoV-2 in a cat owned by a COVID-19−affected patient in Spain. In: PNAS. Online-Vorabveröffentlichung vom 18. September 2020, doi:10.1073/pnas.2010817117.
Details Revealed About the First Cat Infected With SARS-CoV-2 Coronavirus in Spain – Including Necropsy Findings. Auf: scitechdaily.com vom 25. September 2020. - ↑ Covid-19 bei einer Katze in der Schweiz. In: Bundesamt für Lebensmittelsicherheit und Veterinärwesen. 3. Dezember 2020, abgerufen am 4. Dezember 2020.
- ↑ Angela M. Bosco-Lauth et al.: Experimental infection of domestic dogs and cats with SARS-CoV-2: Pathogenesis, transmission, and response to reexposure in cats. PNAS, 29. September 2020, doi:10.1073/pnas.2013102117
- ↑ Coronavirus rips through Dutch mink farms, triggering culls to prevent human infections. Auf: sciencemag.org vom 9. Juni 2020.
- ↑ Pelztiere erkranken massenhaft an Covid-19. Auf: sueddeutsche.de vom 14. Mai 2020.
- ↑ Nadia Oreshkova et al.: SARS-CoV2 infection in farmed mink, Netherlands, April 2020. Preprint auf bioRxiv vom 18. Mai 2020, doi:10.1101/2020.05.18.101493.
- ↑ International Society for Infectious Diseases: Coronavirus Disease 2019 Update (209): Netherlands (North Brabant), Farmed Mink, Animal-to-Human, Cat, Epidemiology. Bericht vom 25. Mai 2020.
- ↑ International Society for Infectious Diseases: Coronavirus Disease 2019 Update (215): Netherlands (NB), Animal, Mink-to-Human, Epidemiology, Control. Bericht vom 27. Mai 2020.
- ↑ Bas B. Oude Munnink, Reina S. Sikkema, David F. Nieuwenhuijse et al.: Transmission of SARS-CoV-2 on mink farms between humans and mink and back to humans. In: Science. Online-Publikation vom 10. November 2020, eabe5901, doi:10.1126/science.abe5901.
- ↑ OIE: SARS-CoV-2/COVID-19, United States of America. Mitteilung des United States Department of Agriculture, Washington, vom 26. Juli 2020.
OIE: SARS-CoV-2/COVID-19, United States of America. Mitteilung des United States Department of Agriculture, Washington, vom 2. Oktober 2020. - ↑ David Nield: COVID-19 Has Caused Over 1 Million Mink to Be Killed Due to Culling at Fur Farms, auf: sciencealert vom 14. Oktober 2020.
- ↑ Denmark to cull up to 17 million mink amid coronavirus fears. Auf: bbc.com vom 5. November 2020.
Warum eine mutierte Coronavirus-Variante Dänemark dazu veranlasst, seine gesamte Nerzpelz-Branche zu zerstören. Auf: nzz.ch vom 6. November 2020. - ↑ a b Bärbel Hilbig, Impfstoff gegen mutierte Viren nutzlos ?, Hannoversche Allgemeine Zeitung, 23. Oktober 2020, S. 1.
- ↑ Christian Stichler: Das große Schlachten auf den Nerzfarmen, auf: tagesschau.de, Stand: 1. November 2020
- ↑ Lars Fischer: Die wahre Gefahr durch das Nerz-Coronavirus, auf: spektrum.de vom 6. November 2020
- ↑ NDR: Dänemark: Wie gefährlich ist das mutierte Corona-Virus? Abgerufen am 7. November 2020.
- ↑ Spread of mutated coronavirus in Danish mink ‘hits all the scary buttons,’ but fears may be overblown. Auf: statnews.com vom 5. November 2020.
- ↑ Coronavirus mutiert – Dänemark lässt alle Nerze notschlachten, auf: n-tv.de vom 4. November 2020 (Video)
- ↑ Briten: Einreiseverbot für Dänen – Infizierte Nerze in sechs Ländern entdeckt, auf: n-tv.de vom 7. November 2020
- ↑ Corona-Mutation in Dänemark – WHO gibt Risiko-Einschätzung zu Nerz-Virus, auf: n-tv.de vom 6. November 2020
- ↑ Detection of new SARS-CoV-2 variants related to mink, ecdc.
- ↑ Millionen gekeulter Tiere: Dänemark will tote Nerze wieder ausgraben, auf: n-tv.de vom 20. Dezember 2020
- ↑ Young-Il Kim et al.: Infection and Rapid Transmission of SARS-CoV-2 in Ferrets. In: Cell Host & Microbe, Online-Vorabveröffentlichung vom 6. April 2020, doi:10.1016/j.chom.2020.03.023.
- ↑ a b Neues Coronavirus SARS-CoV-2: Flughunde und Frettchen sind empfänglich, Schweine und Hühner nicht. Auf: idw-online.de vom 2. April 2020.
- ↑ Stephanie Pappas: Endangered ferrets get experimental COVID-19 vaccine, auf: LiveScience vom 1. Januar 2021
- ↑ Nach Massentötungen in Dänemark – Finnland entwickelt Corona-Impfung für Nerze, auf: n-tv vom 12. Januar 2021
- ↑ Entscheidende Testphase läuft – Russland entwickelt Corona-Impfstoff für Tiere, auf: n-tv vom 13. Dezember 2020
- ↑ Gorilla Troop at the San Diego Zoo Safari Park Test Positive for COVID-19. Auf: zoo.sandiegozoo.org vom 11. Januar 2021.
- ↑ USDA-APHIS confirms COVID-19 in gorillas at San Diego Zoo Safari Park. Auf: federallabs.org vom 11. Januar 2021.
- ↑ Nadja Podbregar: Erste Gorillas mit dem Coronavirus infiziert – Menschenaffen im Zoo von San Diego sind an Covid-19 erkrankt. Auf: scinexx.de vom 12. Januar 2021, Quelle: San Diego Zoo Global
- ↑ NCBI: Human coronavirus OC43 (no rank)
- ↑ Nadja Podbregar: Coronavirus: Sind auch Menschenaffen gefährdet? Auf: scinexx.de vom 30. März 2020.
- ↑ Chuan Qin et al.: Reinfection could not occur in SARS-CoV-2 infected rhesus macaques. In: bioRxiv. 14. März 2020, bioRxiv: 10.1101/2020.03.13.990226v1 (Preprint-Volltext), doi:10.1101/2020.03.13.990226.
- ↑ Keine Mehrfachansteckung mit SARS-CoV-2 bei Affen. In: Deutsches Ärzteblatt. Deutscher Ärzteverlag, 18. März 2020, archiviert vom (nicht mehr online verfügbar) am 19. März 2020; abgerufen am 19. März 2020.
- ↑ Können Menschen zweimal Covid-19 kriegen?, auf: n-tv.de vom 18. April 2019, Quelle: ntv.de, Amélie Bottollier-Depois, AFP
- ↑ Abishek Chandrashekar et al.: SARS-CoV-2 infection protects against rechallenge in rhesus macaques. In: Science, Online-Vorabveröffentlichung vom 20. Mai 2020, eabc4776, doi:10.1126/science.abc4776.
- ↑ Barry Rockx, Thijs Kuiken, Sander Herfst et al.: Comparative pathogenesis of COVID-19, MERS, and SARS in a nonhuman primate model. In: Science, Online-Vorabveröffentlichung vom 17. April 2020, abb7314, doi:10.1126/science.abb7314.
- ↑ A Tiger In New York Has tested Positive For Coronavirus. Auf: sciencealert.com vom 6. April 2020. Quelle: Agence France-Presse.
- ↑ OIE: Immediate notification: SARS-CoV-2/COVID-19, United States of America. Bronx County, Bronx, New York. Auf: oie.int vom 6. April 2020.
- ↑ SARS-COV-2/COVID-19, South Africa. Meldung des Department of Agriculture, Forestry and Fisheries, Animal Production and Health an die OIE, 11. August 2020.
- ↑ Jianzhong Shi et al.: Susceptibility of ferrets, cats, dogs, and other domesticated animals to SARS–coronavirus 2. In: Science, Online-Vorabveröffentlichung vom 8. April 2020, eabb7015, doi:10.1126/science.abb7015.
- ↑ Jon Cohen: From mice to monkeys, animals studied for coronavirus answers. In: Science, Band 368, Nr. 6488, 2020, S. 221 f, doi:10.1126/science.368.6488.221, Volltext
- ↑ Kore Schlottau, Melanie Rissmann, Annika Graaf, Jacob Schön, Julia Sehl, Claudia Wylezich, Martin Beer et al.: SARS-CoV-2 in fruit bats, ferrets, pigs, and chickens: an experimental transmission study, in: The Lancet vom 7. Jul 2020, doi:https://doi.org/10.1016/S2666-5247(20)30089-6, PDF
- ↑ https://www.agrarheute.com/tier/rind/corona-studie-empfaenglich-rinder-572258 Corona-Studie: Wie empfänglich sind Rinder?
- ↑ Shi-Hui Sun, You-Chun Wang et al.: A Mouse Model of SARS-CoV-2 Infection and Pathogenesis, in: Cell Host and Microbe vom 26. Mai 2020, doi:10.1016/j.chom.2020.05.020
- ↑ Jacinta Bowler: Scientists Find a Way to Infect Mice With Coronavirus. Here's Why That's So Important, auf: sciencealert vom 1. Juni 2020
- ↑ Andrea J. Pruijssers, Amelia S. George, Alexandra Schäfer, Sarah R. Leist, Lisa E. Gralinksi: Remdesivir Inhibits SARS-CoV-2 in Human Lung Cells and Chimeric SARS-CoV Expressing the SARS-CoV-2 RNA Polymerase in Mice. In: Cell Reports. Band 32, Nr. 3, Juli 2020, S. 107940, doi:10.1016/j.celrep.2020.107940, PMID 32668216, PMC 7340027 (freier Volltext).
- ↑ Tina Hesman Saey: Remdesivir may work even better against COVID-19 than we thought, auf: ScienceNews vom 13. Juli 2020
- ↑ Jan Osterkamp: Bedroht Corona auch Wale und Robben?, auf: spektrum.de vom 7. Dezember 2020
- ↑ Informationen der BAuA: Neuartiges Virus SARS-CoV-2 (bislang 2019-nCoV) durch den ABAS in Risikogruppe 3 eingestuft und Empfehlungen zur Labordiagnostik gegeben. In: Website der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA). 19. Februar 2020, abgerufen am 23. Februar 2020.
- ↑ a b Beschluss 1/2020 des ABAS vom 19. 2. 2020 und Begründung zur vorläufigen Einstufung des Virus SARS-CoV-2 in Risikogruppe 3 und Empfehlungen zu nicht gezielten Tätigkeiten (Labordiagnostik) und gezielten Tätigkeiten mit SARS-CoV-2. (PDF; 140 kB) In: Website der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin. 19. Februar 2020, abgerufen am 23. Februar 2020.
- ↑ Interim Laboratory Biosafety Guidelines for Handling and Processing Specimens Associated with Coronavirus Disease 2019 (COVID-19). In: Website der US-amerikanischen Centers for Disease Control and Prevention (CDC). 16. Februar 2020, abgerufen am 23. Februar 2020 (englisch).
- ↑ Chantal B.F. Vogels et al.: Analytical sensitivity and efficiency comparisons of SARS-COV-2 qRT-PCR primer-probe sets. medRxiv, 26. April 2020 doi:10.1101/2020.03.30.20048108
- ↑ Paul-Ehrlich-Institut: COVID-19-Tests: NAT-Test gilt als Goldstandard, zu NAT-Tests gehört auch die PCR-Methode. Stand 23. März 2020, abgerufen am 25. August 2020.
- ↑ RKI: SARS-CoV-2 Steckbrief zur Coronavirus-Krankheit-2019 (COVID-19), Stand 21. August 2020, Abruf 30. August 2020
- ↑ RKI: Hinweise zur Testung von Patienten auf Infektion mit dem neuartigen Coronavirus SARS-CoV-2, Stand 11. August 2020, Abruf 30. August 2020
- ↑ RKI: zeitliche Verhältnisse von Transmission – Latenz – Inkubation – PCR-Nachweis – Infektiosität – AK-Nachweis bei SARS-CoV-2, 3. Juli 2020, Abruf 30. August 2020, Grafik PNG
- ↑ Kristiana Ludwig: Coronavirus: Positiv getestet, aber nicht ansteckend. Abgerufen am 22. Oktober 2020.
- ↑ COVID-19: Entlassungskriterien aus der Isolierung - In Abstimmung mit der Arbeitsgruppe Infektionsschutz der AOLG, zuletzt abgerufen am 6. September 2020.
- ↑ a b c d e Coronavirus outbreak: New test kit can detect the virus in just 15 minutes. In: MSN. 10. Februar 2020, abgerufen am 16. Februar 2020 (englisch).
- ↑ Produzent von Corona-Tests: Firma im Ausnahmezustand. In: Die Tageszeitung taz.de. 12. März 2020, abgerufen am 14. März 2020.
- ↑ Coronavirus disease (COVID-19) technical guidance: Laboratory testing for 2019-nCoV in humans. In: Website WHO. Weltgesundheitsorganisation (WHO), 2020, abgerufen am 15. März 2020 (englisch).
- ↑ Stefan Nicola: A Berlin Biotech Company Got a Head Start on Coronavirus Tests. In: Bloomberg Businessweek. 12. März 2020, abgerufen am 14. März 2020 (englisch).
- ↑ Frankfurter Forscher entwickeln schnelleren Coronavirus-Test. In: bz-berlin.de. 31. März 2020, abgerufen am 1. April 2020.
- ↑ Direkter Erregernachweis durch RT-PCR. In: Website des Robert Koch-Instituts. 11. August 2020, abgerufen am 30. August 2020.
- ↑ RKI: Nationale Teststrategie – wer wird in Deutschland getestet?, Stand 12. August 2020, Abruf 28. August 2020
- ↑ Antworten auf häufig gestellte Fragen zum Coronavirus SARS-CoV-2 / Krankheit COVID-19. In: rki.de. Robert Koch-Institut, 17. September 2020, abgerufen am 20. September 2020.
- ↑ a b c Deutsche Akademie der Naturforscher Leopoldina (Hrsg.): Coronavirus-Pandemie: Wirksame Regeln für Herbst und Winter aufstellen. 23. September 2020 (leopoldina.org [PDF]).
- ↑ Prof. Dr. Heinz Zeichhardt, Dr. Martin Kammel: Kommentar zum Extra Ringversuch Gruppe 340 Virusgenom-Nachweis – SARS-CoV-2. (PDF) 3. Juni 2020, abgerufen am 10. Juni 2020.
- ↑ a b Foundation for Innovative New Diagnostics: SARS-CoV-2 molecular assay evaluation: results. 9. Juni 2020, abgerufen am 13. Juni 2020 (englisch).
- ↑ Kerstin Wernike, Markus Keller, Franz J. Conraths, Thomas C. Mettenleiter, Martin H. Groschup, Martin Beer: Pitfalls in SARS-CoV-2 PCR Diagnostics. In: Transboundary and Emerging Diseases. 14. Juni 2020, doi:10.1111/tbed.13684 (englisch).
- ↑ a b Yafang Li, Lin Yao, Jiawei Li, Lei Chen, Yiyan Song, Zhifang Cai, Chunhua Yang: Stability issues of RT-PCR testing of SARS-CoV-2 for hospitalized patients clinically diagnosed with COVID-19. In: Journal of medical virology. 26. März 2020, doi:10.1002/jmv.25786, PMID 32219885 (englisch, Epub ahead of print).
- ↑ a b Emily Feng, Amy Cheng: Mystery In Wuhan: Recovered Coronavirus Patients Test Negative … Then Positive. In: NPR.org. National Public Radio, Inc. (USA), 27. März 2020, abgerufen am 14. April 2020 (englisch, teilweise irreführend: Der Satz: “There are false positives with these types of tests,” bezieht sich nicht auf einen bestimmten Test, wie man vermuten könnte).
- ↑ Lauren M. Kucirka: Variation in False-Negative Rate of Reverse Transcriptase Polymerase Chain Reaction–Based SARS-CoV-2 Tests by Time Since Exposure. In: Annals of Internal Medicine. 18. August 2020, doi:10.7326/M20-1495.
- ↑ Jaffar Al-Tawfiq, Ziad A. Memish: Diagnosis of SARS-CoV-2 Infection based on CT scan vs. RT-PCR: Reflecting on Experience from MERS-CoV. In: The Journal of Hospital Infection. 5. März 2020, doi:10.1016/j.jhin.2020.03.001 (englisch).
- ↑ a b c d e Laboratory testing for 2019 novel coronavirus (2019-nCoV) in suspected human cases. In: Website WHO. Weltgesundheitsorganisation (WHO), 17. Januar 2020, abgerufen am 5. Februar 2020 (englisch).
- ↑ Global Initiative on Sharing All Influenza Data (GISAID). In: Website der GISAID (Bundesrepublik Deutschland als Host (Datenbankanbieter)). Abgerufen am 5. Februar 2020 (englisch).
- ↑ CDC Tests for 2019-nCoV. In: Website der US-amerikanischen Centers for Disease Control and Prevention (CDC). 5. Februar 2020, abgerufen am 10. Februar 2020 (englisch).
- ↑ Stephanie Soucheray: CDC sends out nCoV test kits as Wisconsin confirms case. In: Website der University of Minnesota. CIDRAP – Center for Infectious Disease Research and Policy, 5. Februar 2020, abgerufen am 10. Februar 2020 (englisch).
- ↑ Axel Schröder: Corona-Krise: So werden Virusnachweis-Kits produziert. In: Website Deutschlandfunk. 6. März 2020, abgerufen am 15. März 2020.
- ↑ RealStar SARS-CoV-2 RT-PCR Kit 1.0. (PDF; 596 kB) In: Website Altona Diagnostics. Februar 2020, abgerufen am 15. März 2020 (englisch).
- ↑ a b c M. Parčina, U. V. Schneider, B. Visseaux, R. Jozić, I. Hannet, J. G. Lisby: Multicenter evaluation of the QIAstat Respiratory Panel – A new rapid highly multiplexed PCR based assay for diagnosis of acute respiratory tract infections. In: PLOS ONE. Band 15, Nr. 3, 12. März 2020, S. e0230183, doi:10.1371/journal.pone.0230183, PMID 32163484, PMC 7067435 (freier Volltext).
- ↑ New test kits for coronavirus approved in China. In: Website Xinhua. 30. Januar 2020, abgerufen am 10. Februar 2020 (englisch).
- ↑ China's Jiangsu develops rapid test kit for coronavirus. In: Website Xinhua. 31. Januar 2020, abgerufen am 16. Februar 2020 (englisch).
- ↑ Dean Koh: iHealthtech researchers working on Wuhan novel coronavirus (2019-nCoV) detection kit. In: Website mobi health news der National University of Singapore. 6. Februar 2020, abgerufen am 16. Februar 2020 (englisch).
- ↑ M. Döhla, C. Boesecke, B. Schulte, C. Diegmann, E. Sib: Rapid point-of-care testing for SARS-CoV-2 in a community screening setting shows low sensitivity. In: Public Health. Band 182, Mai 2020, S. 170–172, doi:10.1016/j.puhe.2020.04.009, PMID 32334183, PMC 7165286 (freier Volltext).
- ↑ a b Hinweise zur Testung von Patienten auf Infektion mit dem neuartigen Coronavirus SARS-CoV-2. In: rki.de. 30. November 2020, abgerufen am 20. Dezember 2020.
- ↑ a b Nationale Teststrategie – wer wird in Deutschland auf das Vorliegen einer SARS-CoV-2 Infektion getestet? Robert Koch-Institut, 18. Dezember 2020, abgerufen am 20. Dezember 2020.
- ↑ Vergleichende Evaluierung der Sensitivität von SARS-CoV-2 Antigenschnelltests. In: pei.de. Paul-Ehrlich-Institut, 17. Dezember 2020, abgerufen am 20. Dezember 2020.
- ↑ Victor M. Corman, Christian Drosten u. a.: Comparison of seven commercial SARS-CoV-2 rapid Point-of-Care Antigen tests. In: medRxiv. Nr. 6 (medrxiv.org).
- ↑ Nationale Akademie der Wissenschaften Leopoldina (Hrsg.): Coronavirus-Pandemie: Die Feiertage und den Jahreswechsel für einen harten Lockdown nutzen. 8. Dezember 2020 (leopoldina.org [PDF]).
- ↑ Nationale Teststrategie. In: rki.de. 18. Dezember 2020, abgerufen am 20. Dezember 2020.
- ↑ a b c Hildener Unternehmen entwickelt Schnelltest für Corona-Virus. In: wdr.de. 5. Februar 2020, abgerufen am 28. Februar 2020.
- ↑ a b c Pressemitteilung: QIAGEN announces worldwide shipments of QIAstat-Dx test kits for SARS-CoV-2. In: Website Qiagen. 26. Februar 2020, abgerufen am 28. Februar 2020 (englisch).
- ↑ a b Maike Telgheder: Diagnose Coronavirus: Die Industrie entwickelt Schnelltests. In: Website Handelsblatt. 26. Februar 2020, abgerufen am 28. Februar 2020.
- ↑ a b c d e f Andreas Stiller: Ein tieferer Einblick in die Infektions-Tests gegen das Coronavirus SARS-CoV-2. In: heise online. 27. März 2020, abgerufen am 28. März 2020.
- ↑ Joe Miller: Bosch pushes into Covid-19 market with rapid test. In: Finacial Times. 25. September 2020, abgerufen am 18. Dezember 2020 (englisch).
- ↑ a b c S. Y. Xiao, Y. Wu, H. Liu: Evolving status of the 2019 novel coronavirus infection: Proposal of conventional serologic assays for disease diagnosis and infection monitoring. In: Journal of Medical Virology. Band 92, Nummer 5, Mai 2020, vorab online veröffentlicht, S. 464–467, doi:10.1002/jmv.25702, PMID 32031264.
- ↑ a b Juanjuan Zhao, Quan Yuan et al.: Antibody responses to SARS-CoV-2 in patients of novel coronavirus disease 2019. In: Clinical Infectious Diseases. 28. März 2020, doi:10.1093/cid/ciaa344.
- ↑ 2019-nCoV: Verdachtsabklärung und Maßnahmen – Orientierungshilfe für Ärztinnen und Ärzte. In: Website des Robert Koch-Instituts. 23. Januar 2020, abgerufen am 3. April 2020.
- ↑ Hinweise zur Testung von Patienten auf Infektion mit dem neuartigen Coronavirus SARS-CoV-2. In: Website des Robert Koch-Instituts (RKI). 20. Februar 2020, abgerufen am 25. Februar 2020.
- ↑ a b LADR informiert Analytik Nr. 292 03/2020: Testung auf Antikörper gegen COVID-19 (SARS-CoV-2) verfügbar
- ↑ a b c d Nisreen M.A. Okba1, Marcel A. Müller, Wentao Li, Chunyan Wang, Corine H. GeurtsvanKessel, Victor M. Corman, Mart M. Lamers, Reina S. Sikkema, Erwin de Bruin, Felicity D. Chandler, Yazdan Yazdanpanah, Quentin Le Hingrat, Diane Descamps, Nadhira Houhou-Fidouh, Chantal B.E.M. Reusken, Berend-Jan Bosch, Christian Drosten, Marion P.G. Koopmans, Bart L. Haagmans: Severe Acute Respiratory Syndrome Coronavirus 2−Specific Antibody Responses in Coronavirus Disease 2019 Patients. In: Emerging Infectious Diseases. Band 26, Nr. 7, doi:10.3201/eid2607.200841 (Juli 2020, vorab online veröffentlicht).
- ↑ a b c d Antikörper und ihre Antigene; Methoden der mikrobiologischen Diagnostik. In: Helmut Hahn, Stefan H. E. Kaufmann, Thomas F. Schulz, Sebastian Suerbaum (Hrsg.): Medizinische Mikrobiologie und Infektiologie. 6. Auflage. Springer Verlag, Heidelberg 2009, ISBN 978-3-540-46359-7, S. 48–62, 140–150.
- ↑ Some People May Have a Head Start Against Coronavirus, Surprising Evidence Shows, auf: sciencealert vom 4. Juni 2020, Quelle: Business Insider
- ↑ Jef Akst: Australian Lab Cultures New Coronavirus as Infections Climb. In: Website The-Scientist. 29. Januar 2020, abgerufen am 4. Februar 2020 (englisch).
- ↑ Institut Pasteur isolates strains of coronavirus 2019-nCoV detected in France. In: Website EurekAlert! 31. Januar 2020, abgerufen am 4. Februar 2020 (englisch).
- ↑ Erstmals nCoV in Deutschland in Zellkultur isoliert. In: Website Institut für Mikrobiologie der Bundeswehr. 31. Januar 2020, abgerufen am 3. Februar 2020.
- ↑ CDC Grows 2019-nCoV Virus in Cell Culture. In: Website der US-amerikanischen Centers for Disease Control and Prevention (CDC). 6. Februar 2020, abgerufen am 10. Februar 2020 (englisch).
- ↑ Jon Cohen: Scientists are moving at record speed to create new coronavirus vaccines—but they may come too late. In: Science. 27. Januar 2020, doi:10.1126/science.abb0612 (englisch).
- ↑ Finn Mayer-Kuckuk: Suche nach Corona-Impfstoff: Das heiße Rennen. In: Die Tageszeitung: taz. 17. März 2020, ISSN 0931-9085 (taz.de [abgerufen am 18. März 2020]).
- ↑ Lars Fischer, Alina Schadwinkel: Verursacht das Coronavirus Engpässe bei Medikamenten? Stammt das Virus aus dem Pangolin? Website Spektrum.de, 10. Februar 2020, abgerufen am 15. Februar 2020.
- ↑ Sechste Infektion bestätigt: Kind in Bayern an Coronavirus erkrankt. In: Website tagesschau.de. 31. Januar 2020, abgerufen am 9. Februar 2020.
- ↑ Donald Trumps Test auf Coronavirus negativ ausgefallen. In: Der Spiegel – Politik. Abgerufen am 15. März 2020.
- ↑ WHO: Draft landscape of COVID-19 candidate vaccines. (PDF) (PDF). In: Website der World Health Organization (WHO). WHO, 2. Dezember 2020, abgerufen am 3. Dezember 2020 (englisch, Spezifikationen und Links zu weiteren Infos über alle von der WHO aufgelisteten Impfstoffkandidaten).
- ↑ Wie soll der Zugang zu einem COVID-19-Impfstoff geregelt werden?, Positionspapier der Ständigen Impfkommission beim Robert Koch-Institut, des Deutschen Ethikrates und der Nationalen Akademie der Wissenschaften Leopoldina, 9. November 2020. Abgerufen am 9. November 2020.
- ↑ Coronavirus vaccine developers wary of errant antibodies www.nature.com, 5. Juni 2020
- ↑ Pathogenesis of oral type I feline infectious peritonitis virus (FIPV) infection: Antibody-dependent enhancement infection of cats with type I FIPV via the oral route www.ncbi.nlm.nih.gov, Online-Veröffentlichung 23. April 2019
- ↑ www.rki.de: Lieferengpässe.
- ↑ Gesundheitssenatorin begrüßt Ausweitung des Risikogebiets durch Robert-Koch-Institut. In: berlin.de. 26. Februar 2020, abgerufen am 29. Februar 2020.
- ↑ Coronavirus: Wann gilt der Katastrophenfall – und was folgt? In: rbb24.de. 26. Februar 2020, abgerufen am 29. Februar 2020.
- ↑ Weltgesundheitsorganisation: Statement on the second meeting of the International Health Regulations (2005) Emergency Committee regarding the outbreak of novel coronavirus (2019-nCoV). 30. Januar 2020, abgerufen am 30. Januar 2020 (englisch).
- ↑ Tedros Adhanom Ghebreyesus: WHO Director-General's opening remarks at the media briefing on COVID-19 – 11 March 2020. In: Website WHO. 11. März 2020, abgerufen am 12. März 2020 (englisch).
- ↑ Amtliche Begründung im Gesetzentwurf auf BT-Drs. 19/18967, S. 55.
- ↑ Schutzpaket für mehr Coronatests und Pflege-Prämien beschlossen. Deutscher Bundestag, 14. Mai 2020, abgerufen am 14. Mai 2020.
- ↑ Nichtnamentliche Meldepflicht von Untersuchungsergebnissen. Robert Koch-Institut, 4. Juni 2020, abgerufen am 8. Juni 2020.
- ↑ Drittes Gesetz zum Schutz der Bevölkerung bei einer epidemischen Lage von nationaler Tragweite vom 18. November 2020, "S." oder "Nr." fehlt (Hilfe).
- ↑ Meldepflicht bei Corona-Infektionen von Haustieren. Presse- und Informationsamt der Bundesregierung, 2. Juli 2020, abgerufen am 1. November 2020.
- ↑ Epidemiegesetz 1950. Bundesrecht konsolidiert, Gesamte Rechtsvorschrift. In: RIS. 14. Juni 2018, abgerufen am 6. März 2020: „Der Anzeigepflicht unterliegen: […] (2) Der Bundesminister für Gesundheit und Frauen kann, […], durch Verordnung weitere übertragbare Krankheiten der Meldepflicht unterwerfen oder bestehende Meldepflichten erweitern.“
- ↑ 15. Verordnung des Bundesministers für Arbeit, Soziales, Gesundheit und Konsumentenschutz betreffend anzeigepflichtige übertragbare Krankheiten 2020. In: Bundesgesetzblatt für die Republik Österreich. 26. Januar 2020, abgerufen am 4. März 2020: „Der Anzeigepflicht nach dem Epidemiegesetz1950 unterliegen Verdachts-, Erkrankungs- und Todesfälle an 2019-nCoV („2019 neuartiges Coronavirus“).“
- ↑ Absonderungsverordnung. Bundesrecht konsolidiert: Gesamte Rechtsvorschrift für Absonderung Kranker, Krankheitsverdächtiger und Ansteckungsverdächtiger und Bezeichnung von Häusern und Wohnungen, Fassung vom 06.03.2020. In: RIS, Bundesrecht konsolidiert. 31. Januar 2020, abgerufen am 6. März 2020: „Bei Masern oder Infektion mit 2019-nCoV (“2019 neuartiges Coronavirus„) sind die Kranken und Krankheitsverdächtigen abzusondern oder nach den Umständen des Falles lediglich bestimmten Verkehrsbeschränkungen zu unterwerfen.“
- ↑ Verordnung vom 31. Jänner 2020. 21. Verordnung des Bundesministers für Soziales, Gesundheit, Pflege und Konsumentenschutz, mit der die Verordnung des Ministers des Innern im Einvernehmen mit dem Minister für Kultus und Unterricht vom 22. Februar 1915, betreffend die Absonderung Kranker, Krankheitsverdächtiger und Ansteckungsverdächtiger und die Bezeichnung von Häusern und Wohnungen geändert wird. In: Bundesgesetzblatt für die Republik Österreich. 31. Januar 2020, abgerufen am 6. März 2020: „In § 4 3. Satz wird nach dem Wort „Masern“ die Wortfolge „oder Infektion mit 2019-nCoV (“2019 neuartiges Coronavirus„)“ eingefügt.“
- ↑ Neues Coronavirus: Informationen für Gesundheitsfachpersonen. Verdachts- und Meldekriterien sowie Meldeformular. Bundesamt für Gesundheit, 5. März 2020, abgerufen am 5. März 2020.
- ↑ Bundesgesetz über die Bekämpfung übertragbarer Krankheiten des Menschen. Epidemiengesetz, EpG. Bundeskanzlei, 1. Januar 2017, abgerufen am 6. März 2020.
- ↑ Verordnung über die Bekämpfung übertragbarer Krankheiten des Menschen. (Epidemienverordnung, EpV) vom 29. April 2015 (Stand am 1. März 2019). Bundeskanzlei, abgerufen am 6. März 2020.
- ↑ Verordnung des EDI über die Meldung von Beobachtungen übertragbarer Krankheiten des Menschen. vom 1. Dezember 2015 (Stand am 1. Februar 2020). Bundeskanzlei, abgerufen am 6. März 2020.
- ↑ Meldeformulare. COVID-19 Meldung. Bundesamt für Gesundheit, 18. Mai 2020, abgerufen am 8. Juni 2020.